
Un plásmido es una pequeña molécula de ADN extracromosómico dentro de una célula que está físicamente separada del ADN cromosómico y puede replicarse de forma independiente. Se encuentran más comúnmente como pequeñas moléculas de ADN circulares de doble cadena en bacterias ; sin embargo, los plásmidos a veces están presentes en arqueas y organismos eucariotas . [1] [2] Los plásmidos a menudo contienen genes útiles, como resistencia a antibióticos y virulencia . [3] [4] [5] Mientras que los cromosomas son grandes y contienen toda la información genética esencial para vivir en condiciones normales, los plásmidos suelen ser muy pequeños y contienen genes adicionales para circunstancias especiales.
Los plásmidos artificiales se utilizan ampliamente como vectores en la clonación molecular , y sirven para impulsar la replicación de secuencias de ADN recombinante dentro de organismos hospedadores. En el laboratorio, los plásmidos pueden introducirse en una célula mediante transformación . Los plásmidos sintéticos están disponibles para su adquisición a través de Internet por parte de varios proveedores que utilizan secuencias presentadas, generalmente diseñadas con software; si un diseño no funciona, el proveedor puede realizar modificaciones adicionales a partir del diseño presentado. [6] [7] [8]
Los plásmidos se consideran replicones , unidades de ADN capaces de replicarse de forma autónoma dentro de un huésped adecuado. Sin embargo, los plásmidos, como los virus , generalmente no se clasifican como vida . [9] Los plásmidos se transmiten de una bacteria a otra (incluso de otra especie) principalmente a través de la conjugación . [3] Esta transferencia de material genético de huésped a huésped es un mecanismo de transferencia genética horizontal , y los plásmidos se consideran parte del mobiloma . A diferencia de los virus, que encierran su material genético en una capa de proteína protectora llamada cápside , los plásmidos son ADN "desnudo" y no codifican genes necesarios para encapsular el material genético para su transferencia a un nuevo huésped; sin embargo, algunas clases de plásmidos codifican el pilus "sexual" conjugativo necesario para su propia transferencia. Los plásmidos varían en tamaño de 1 a más de 400 k pb , [10] y el número de plásmidos idénticos en una sola célula puede variar de uno a miles.
El término plásmido fue acuñado en 1952 por el biólogo molecular estadounidense Joshua Lederberg para referirse a "cualquier determinante hereditario extracromosómico". [11] [12] El uso temprano del término incluía cualquier material genético bacteriano que existe extracromosómicamente durante al menos una parte de su ciclo de replicación, pero debido a que esa descripción incluye virus bacterianos, la noción de plásmido se refinó con el tiempo para referirse a elementos genéticos que se reproducen de forma autónoma. [13] Más tarde, en 1968, se decidió que el término plásmido debería adoptarse como el término para el elemento genético extracromosómico, [14] y para distinguirlo de los virus, la definición se redujo a elementos genéticos que existen exclusivamente o predominantemente fuera del cromosoma, pueden replicarse de forma autónoma y contribuyen a transferir elementos móviles entre bacterias no relacionadas. [3] [4] [13]

Para que los plásmidos se repliquen de forma independiente dentro de una célula, deben poseer un tramo de ADN que pueda actuar como un origen de replicación . La unidad autorreplicante, en este caso, el plásmido, se llama replicón . Un replicón bacteriano típico puede constar de varios elementos, como el gen para la proteína de iniciación de replicación específica del plásmido (Rep), unidades repetitivas llamadas iterones , cajas DnaA y una región adyacente rica en AT. [13] Los plásmidos más pequeños utilizan las enzimas replicativas del huésped para hacer copias de sí mismos, mientras que los plásmidos más grandes pueden llevar genes específicos para la replicación de esos plásmidos. Algunos tipos de plásmidos también pueden insertarse en el cromosoma del huésped, y estos plásmidos integradores a veces se denominan episomas en procariotas . [15]
Los plásmidos casi siempre portan al menos un gen. Muchos de los genes que porta un plásmido son beneficiosos para las células huésped, por ejemplo: permiten que la célula huésped sobreviva en un entorno que de otro modo sería letal o restrictivo para el crecimiento. Algunos de estos genes codifican rasgos de resistencia a los antibióticos o a los metales pesados, mientras que otros pueden producir factores de virulencia que permiten a una bacteria colonizar un huésped y superar sus defensas o tener funciones metabólicas específicas que permiten a la bacteria utilizar un nutriente particular, incluida la capacidad de degradar compuestos orgánicos recalcitrantes o tóxicos. [16] Los plásmidos también pueden proporcionar a las bacterias la capacidad de fijar nitrógeno . Algunos plásmidos, llamados plásmidos crípticos , desempeñan un papel crucial en la transferencia horizontal de genes , ya que llevan genes de resistencia a los antibióticos . Por lo tanto, son factores importantes en la propagación de la resistencia, que puede dar lugar a fracasos del tratamiento con antibióticos. [17]
Los plásmidos naturales varían mucho en sus propiedades físicas. Su tamaño puede variar desde miniplásmidos muy pequeños de menos de 1 kilopar de bases (kbp) hasta megaplásmidos muy grandes de varios megapares de bases (Mbp). En el extremo superior, hay poca diferencia entre un megaplásmido y un minicromosoma . Los plásmidos son generalmente circulares, pero también se conocen ejemplos de plásmidos lineales. Estos plásmidos lineales requieren mecanismos especializados para replicar sus extremos. [13]
Los plásmidos pueden estar presentes en una célula individual en un número variable, que va desde uno hasta varios cientos. El número normal de copias de plásmido que se puede encontrar en una sola célula se denomina número de copias del plásmido y está determinado por cómo se regula la iniciación de la replicación y el tamaño de la molécula. Los plásmidos más grandes tienden a tener un menor número de copias. [15] Los plásmidos con un bajo número de copias que existen solo como una o unas pocas copias en cada bacteria corren el riesgo de perderse en una de las bacterias segregantes tras la división celular . Estos plásmidos de una sola copia tienen sistemas que intentan distribuir activamente una copia a ambas células hijas. Estos sistemas, que incluyen el sistema parABS y el sistema parMRC , a menudo se denominan sistema de partición o función de partición de un plásmido. [18]
Los plásmidos de forma lineal son desconocidos entre los fitopatógenos con una excepción, Rhodococcus fascians . [19]

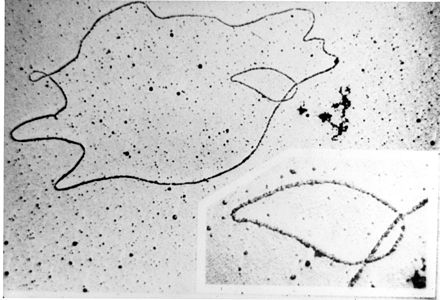

Los plásmidos pueden clasificarse de varias maneras. Los plásmidos pueden clasificarse ampliamente en plásmidos conjugativos y plásmidos no conjugativos. Los plásmidos conjugativos contienen un conjunto de genes de transferencia que promueven la conjugación sexual entre diferentes células. [15] En el complejo proceso de conjugación , los plásmidos pueden transferirse de una bacteria a otra a través de pili sexuales codificados por algunos de los genes de transferencia (ver figura). [20] Los plásmidos no conjugativos son incapaces de iniciar la conjugación, por lo tanto, pueden transferirse solo con la ayuda de plásmidos conjugativos. Una clase intermedia de plásmidos son movilizables y llevan solo un subconjunto de los genes necesarios para la transferencia. Pueden parasitar un plásmido conjugativo, transfiriéndose a alta frecuencia solo en su presencia. [21]
Los plásmidos también pueden clasificarse en grupos de incompatibilidad. Un microbio puede albergar distintos tipos de plásmidos, pero estos solo pueden existir en una única célula bacteriana si son compatibles. Si dos plásmidos no son compatibles, uno u otro se perderá rápidamente de la célula. Por lo tanto, los distintos plásmidos pueden asignarse a distintos grupos de incompatibilidad en función de si pueden coexistir juntos. Los plásmidos incompatibles (que pertenecen al mismo grupo de incompatibilidad) normalmente comparten los mismos mecanismos de replicación o partición y, por lo tanto, no pueden mantenerse juntos en una única célula. [22] [23]
Otra forma de clasificar los plásmidos es por su función. Existen cinco clases principales:
Los plásmidos pueden pertenecer a más de uno de estos grupos funcionales.
Aunque la mayoría de los plásmidos son moléculas de ADN de doble cadena, algunos consisten en ADN monocatenario o predominantemente ARN de doble cadena . Los plásmidos de ARN son replicones de ARN lineales extracromosómicos no infecciosos, tanto encapsidados como no encapsidados, que se han encontrado en hongos y varias plantas, desde algas hasta plantas terrestres. En muchos casos, sin embargo, puede ser difícil o imposible distinguir claramente los plásmidos de ARN de los virus de ARN y otros ARN infecciosos. [30]
Los cromidos son elementos que existen en el límite entre un cromosoma y un plásmido, que se encuentran en aproximadamente el 10% de las especies bacterianas secuenciadas en 2009. Estos elementos llevan genes centrales y tienen un uso de codones similar al cromosoma, pero utilizan un mecanismo de replicación de tipo plásmido como el RepABC de bajo número de copias. Como resultado, se han clasificado de diversas formas como minicromosomas o megaplásmidos en el pasado. [31] En Vibrio , la bacteria sincroniza la replicación del cromosoma y el cromido mediante una proporción de tamaño del genoma conservada. [32]
Los plásmidos construidos artificialmente pueden usarse como vectores en ingeniería genética . Estos plásmidos sirven como herramientas importantes en los laboratorios de genética y biotecnología, donde se usan comúnmente para clonar y amplificar (hacer muchas copias de) o expresar genes particulares. [33] Existe una amplia variedad de plásmidos disponibles comercialmente para tales usos. El gen que se va a replicar normalmente se inserta en un plásmido que típicamente contiene una serie de características para su uso. Estas incluyen un gen que confiere resistencia a antibióticos particulares ( la ampicilina se usa con más frecuencia para cepas bacterianas), un origen de replicación para permitir que las células bacterianas repliquen el ADN del plásmido y un sitio adecuado para la clonación (conocido como sitio de clonación múltiple ).
La inestabilidad estructural del ADN puede definirse como una serie de eventos espontáneos que culminan en un reordenamiento, pérdida o ganancia imprevistos de material genético. Dichos eventos se desencadenan con frecuencia por la transposición de elementos móviles o por la presencia de elementos inestables, como estructuras no canónicas (no B). Las regiones accesorias pertenecientes a la estructura principal bacteriana pueden participar en una amplia gama de fenómenos de inestabilidad estructural. Los catalizadores bien conocidos de la inestabilidad genética incluyen repeticiones directas, invertidas y en tándem, que se sabe que son visibles en una gran cantidad de vectores de clonación y expresión disponibles comercialmente. [34] Las secuencias de inserción también pueden afectar gravemente la función y el rendimiento del plásmido, al provocar deleciones y reordenamientos, activación, regulación negativa o inactivación de la expresión de genes vecinos . [35] Por lo tanto, la reducción o eliminación completa de secuencias de la estructura principal no codificantes extrañas reduciría significativamente la propensión a que se produzcan dichos eventos y, en consecuencia, el potencial recombinogénico general del plásmido. [36] [37]
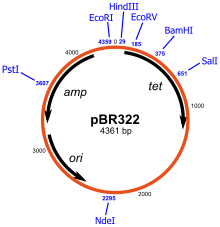
Los plásmidos son los vectores de clonación bacterianos más comúnmente utilizados. [38] Estos vectores de clonación contienen un sitio que permite insertar fragmentos de ADN, por ejemplo, un sitio de clonación múltiple o polienlazador que tiene varios sitios de restricción comúnmente utilizados a los que se pueden ligar fragmentos de ADN . Después de insertar el gen de interés, los plásmidos se introducen en las bacterias mediante un proceso llamado transformación . Estos plásmidos contienen un marcador seleccionable , generalmente un gen de resistencia a antibióticos, que confiere a las bacterias una capacidad para sobrevivir y proliferar en un medio de crecimiento selectivo que contiene los antibióticos particulares. Las células después de la transformación se exponen a los medios selectivos, y solo las células que contienen el plásmido pueden sobrevivir. De esta manera, los antibióticos actúan como un filtro para seleccionar solo las bacterias que contienen el ADN plasmídico. El vector también puede contener otros genes marcadores o genes reporteros para facilitar la selección de plásmidos con insertos clonados. Las bacterias que contienen el plásmido pueden luego cultivarse en grandes cantidades, cosecharse y luego el plásmido de interés puede aislarse utilizando varios métodos de preparación de plásmidos .
Un vector de clonación de plásmidos se utiliza típicamente para clonar fragmentos de ADN de hasta 15 kbp . [39] Para clonar longitudes más largas de ADN, se utilizan fagos lambda con genes de lisogenia eliminados, cósmidos , cromosomas artificiales bacterianos o cromosomas artificiales de levadura .

Otro uso importante de los plásmidos es la producción de grandes cantidades de proteínas. En este caso, los investigadores cultivan bacterias que contienen un plásmido que alberga el gen de interés. Así como la bacteria produce proteínas para conferirle resistencia a los antibióticos, también se la puede inducir a producir grandes cantidades de proteínas a partir del gen insertado. Esta es una forma barata y sencilla de producir la proteína en masa; por ejemplo, utilizando la reproducción rápida de E. coli con un plásmido que contiene el gen de la insulina se obtiene una gran producción de insulina. [40] [41] [42]
Los plásmidos también pueden utilizarse para la transferencia de genes como un posible tratamiento en terapia génica para que puedan expresar la proteína que falta en las células. Algunas formas de terapia génica requieren la inserción de genes terapéuticos en sitios diana cromosómica preseleccionados dentro del genoma humano . Los vectores plasmídicos son uno de los muchos enfoques que podrían utilizarse para este propósito. Las nucleasas de dedo de zinc (ZFN) ofrecen una forma de provocar una ruptura de doble cadena específica del sitio en el genoma de ADN y causar recombinación homóloga . Los plásmidos que codifican ZFN podrían ayudar a entregar un gen terapéutico a un sitio específico para evitar el daño celular , las mutaciones causantes de cáncer o una respuesta inmune . [43]
Históricamente, los plásmidos se han utilizado para modificar genéticamente las células madre embrionarias de ratas con el fin de crear modelos genéticos de enfermedades de ratas. La limitada eficiencia de las técnicas basadas en plásmidos impidió su uso en la creación de modelos de células humanas más precisos. Sin embargo, los avances en las técnicas de recombinación de virus adenoasociados y las nucleasas de dedos de zinc han permitido la creación de una nueva generación de modelos isogénicos de enfermedades humanas .
Los plásmidos ayudan a transportar grupos de genes biogenéticos, un conjunto de genes que contienen todas las enzimas necesarias que conducen a la producción de metabolitos especiales (formalmente conocidos como metabolitos secundarios) . [44] Un beneficio de usar plásmidos para transferir BGC se demuestra al usar un huésped adecuado que puede producir en masa metabolitos especializados, algunas de estas moléculas pueden controlar la población microbiana. [45] Los plásmidos pueden contener y expresar varios BGC con unos pocos plásmidos conocidos por ser exclusivos para transferir BGC. [46] Los BGC también pueden transferirse al cromosoma del organismo huésped, utilizando un vector plasmídico, lo que permite estudios en experimentos de eliminación de genes. [47] Al usar plásmidos para la captación de BGC, los microorganismos pueden obtener una ventaja ya que la producción no se limita a los genes de biosíntesis resistentes a los antibióticos, sino a la producción de toxinas /antitoxinas. [48]
El término episoma fue introducido por François Jacob y Élie Wollman en 1958 para referirse al material genético extracromosómico que puede replicarse de forma autónoma o integrarse en el cromosoma. [49] [50] Sin embargo, desde que se introdujo el término, su uso ha cambiado, ya que plásmido se ha convertido en el término preferido para el ADN extracromosómico que se replica de forma autónoma. En un simposio celebrado en Londres en 1968, algunos participantes sugirieron que se abandonara el término episoma , aunque otros siguieron utilizándolo con un cambio de significado. [51] [52]
Hoy en día, algunos autores utilizan episoma en el contexto de procariotas para referirse a un plásmido que es capaz de integrarse en el cromosoma. Los plásmidos integrativos pueden replicarse y mantenerse de forma estable en una célula a través de múltiples generaciones, pero en alguna etapa, existirán como una molécula de plásmido independiente. [53] En el contexto de eucariotas, el término episoma se utiliza para significar una molécula de ADN circular cerrada extracromosómica no integrada que puede replicarse en el núcleo. [54] [55] Los virus son los ejemplos más comunes de esto, como herpesvirus , adenovirus y poliomavirus , pero algunos son plásmidos. Otros ejemplos incluyen fragmentos cromosómicos aberrantes, como cromosomas dobles diminutos , que pueden surgir durante amplificaciones de genes artificiales o en procesos patológicos (p. ej., transformación de células cancerosas). Los episomas en eucariotas se comportan de manera similar a los plásmidos en procariotas en que el ADN se mantiene y replica de forma estable con la célula huésped. También pueden aparecer episomas virales citoplasmáticos (como en las infecciones por poxvirus ). Algunos episomas, como los herpesvirus, se replican en un mecanismo de círculo rodante , similar a los bacteriófagos (virus fago bacterianos). Otros se replican a través de un mecanismo de replicación bidireccional ( plásmidos de tipo theta ). En cualquier caso, los episomas permanecen físicamente separados de los cromosomas de la célula huésped. Varios virus del cáncer, incluido el virus de Epstein-Barr y el herpesvirus asociado al sarcoma de Kaposi , se mantienen como episomas latentes y cromosómicamente distintos en las células cancerosas, donde los virus expresan oncogenes que promueven la proliferación de células cancerosas. En los cánceres, estos episomas se replican pasivamente junto con los cromosomas del huésped cuando la célula se divide. Cuando estos episomas virales inician la replicación lítica para generar múltiples partículas virales, generalmente activan los mecanismos de defensa de la inmunidad innata celular que matan a la célula huésped.
Algunos plásmidos o huéspedes microbianos incluyen un sistema de adicción o sistema de muerte postsegregacional (PSK), como el sistema hok/sok (host killing/suppressor of killing) del plásmido R1 en Escherichia coli . [56] Esta variante produce tanto un veneno de larga duración como un antídoto de corta duración . En la literatura se han descrito varios tipos de sistemas de adicción de plásmidos (toxina/antitoxina, basados en el metabolismo, sistemas ORT) [ 57] y se han utilizado en aplicaciones biotécnicas (fermentación) o biomédicas (terapia de vacunas). Las células hijas que retienen una copia del plásmido sobreviven, mientras que una célula hija que no hereda el plásmido muere o sufre una tasa de crecimiento reducida debido al veneno persistente de la célula madre. Finalmente, se podría mejorar la productividad general. [ aclaración necesaria ]
Por el contrario, los plásmidos utilizados en biotecnología, como pUC18, pBR322 y vectores derivados, casi nunca contienen sistemas de adicción toxina-antitoxina y, por lo tanto, deben mantenerse bajo presión antibiótica para evitar la pérdida de plásmidos.
Las levaduras albergan de forma natural diversos plásmidos. Entre ellos, destacan los plásmidos de 2 μm (pequeños plásmidos circulares que se utilizan a menudo para la ingeniería genética de la levadura) y los plásmidos pGKL lineales de Kluyveromyces lactis , que son responsables de los fenotipos asesinos. [58]
Otros tipos de plásmidos suelen estar relacionados con los vectores de clonación de levadura, entre los que se incluyen:
Las mitocondrias de muchas plantas superiores contienen moléculas de ADN lineal o circular extracromosómicas autorreplicantes que se han considerado plásmidos. Estos pueden variar de 0,7 kb a 20 kb de tamaño. Los plásmidos se han clasificado generalmente en dos categorías: circulares y lineales. [59] Los plásmidos circulares se han aislado y encontrado en muchas plantas diferentes, siendo los de Vicia faba y Chenopodium album los más estudiados y cuyo mecanismo de replicación es conocido. Los plásmidos circulares pueden replicarse utilizando el modelo de replicación θ (como en Vicia faba ) y a través de la replicación de círculo rodante (como en C. album ). [60] Los plásmidos lineales se han identificado en algunas especies de plantas como Beta vulgaris , Brassica napus , Zea mays , etc. pero son más raros que sus contrapartes circulares.
La función y el origen de estos plásmidos siguen siendo en gran parte desconocidos. Se ha sugerido que los plásmidos circulares comparten un ancestro común, algunos genes en el plásmido mitocondrial tienen contrapartes en el ADN nuclear, lo que sugiere un intercambio entre compartimentos. Mientras tanto, los plásmidos lineales comparten similitudes estructurales, como los invertrones, con el ADN viral y los plásmidos fúngicos; al igual que los plásmidos fúngicos, también tienen un bajo contenido de GC; estas observaciones han llevado a algunas hipótesis de que estos plásmidos lineales tienen orígenes virales o han terminado en las mitocondrias de las plantas a través de la transferencia horizontal de genes desde hongos patógenos. [59] [61]
Los plásmidos se utilizan a menudo para purificar una secuencia específica, ya que se pueden purificar fácilmente a partir del resto del genoma. Para su uso como vectores y para la clonación molecular , los plásmidos a menudo necesitan aislarse.
Existen varios métodos para aislar el ADN plasmídico de las bacterias, que van desde los kits de extracción de plásmidos ( miniprep a maxiprep o bulkprep) , la lisis alcalina , la lisis enzimática y la lisis mecánica. [33] La primera se puede utilizar para averiguar rápidamente si el plásmido es correcto en cualquiera de varios clones bacterianos. El rendimiento es una pequeña cantidad de ADN plasmídico impuro, que es suficiente para el análisis por digestión de restricción y para algunas técnicas de clonación.
En este último caso, se cultivan volúmenes mucho mayores de suspensión bacteriana a partir de los cuales se puede realizar una maxipreparación. En esencia, se trata de una minipreparación a mayor escala seguida de una purificación adicional. Esto da como resultado cantidades relativamente grandes (varios cientos de microgramos) de ADN plasmídico muy puro.
Se han creado muchos kits comerciales para realizar la extracción de plásmidos en diversas escalas, grados de pureza y niveles de automatización.
El ADN plasmídico puede aparecer en una de cinco conformaciones, que (para un tamaño determinado) se mueven a distintas velocidades en un gel durante la electroforesis . Las conformaciones se enumeran a continuación en orden de movilidad electroforética (velocidad para un voltaje aplicado determinado) de la más lenta a la más rápida:
La velocidad de migración de fragmentos lineales pequeños es directamente proporcional al voltaje aplicado a voltajes bajos. A voltajes más altos, los fragmentos más grandes migran a velocidades que aumentan continuamente, pero diferentes. Por lo tanto, la resolución de un gel disminuye con el aumento del voltaje.
A un voltaje bajo especificado, la tasa de migración de fragmentos de ADN lineales pequeños es una función de su longitud. Los fragmentos lineales grandes (de más de 20 kb aproximadamente) migran a una cierta tasa fija independientemente de la longitud. Esto se debe a que las moléculas "respiran", y la mayor parte de la molécula sigue el extremo principal a través de la matriz de gel. Los digestos de restricción se utilizan con frecuencia para analizar plásmidos purificados. Estas enzimas rompen específicamente el ADN en ciertas secuencias cortas. Los fragmentos lineales resultantes forman "bandas" después de la electroforesis en gel . Es posible purificar ciertos fragmentos cortando las bandas del gel y disolviendo el gel para liberar los fragmentos de ADN.
Debido a su conformación ajustada, el ADN superenrollado migra más rápido a través de un gel que el ADN lineal o circular abierto.
El uso de plásmidos como técnica en biología molecular está respaldado por software de bioinformática . Estos programas registran la secuencia de ADN de vectores plasmídicos, ayudan a predecir sitios de corte de enzimas de restricción y a planificar manipulaciones. Ejemplos de paquetes de software que manejan mapas de plásmidos son ApE, Clone Manager , GeneConstructionKit, Geneious, Genome Compiler , LabGenius, Lasergene, MacVector , pDraw32, Serial Cloner, UGENE , VectorFriends, Vector NTI y WebDSV. Estos programas ayudan a realizar experimentos completos in silico antes de hacer experimentos húmedos. [62]
A lo largo de los años se han creado muchos plásmidos y los investigadores los han entregado a bases de datos de plásmidos como las organizaciones sin fines de lucro Addgene y BCCM/GeneCorner. Se pueden encontrar y solicitar plásmidos de esas bases de datos para realizar investigaciones. Los investigadores también suelen subir secuencias de plásmidos a la base de datos del NCBI, de la que se pueden recuperar secuencias de plásmidos específicos.