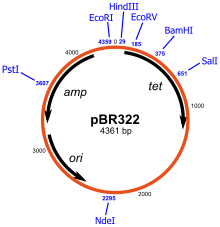
pBR322 es un plásmido y fue uno de los primeros vectores de clonación de E. coli ampliamente utilizados . Creado en 1977 en el laboratorio de Herbert Boyer en la Universidad de California, San Francisco , recibió el nombre de Francisco Bolívar Zapata , el investigador postdoctoral y Raymond L. Rodríguez . La p significa "plásmido" y BR por "Bolívar" y "Rodríguez".
pBR322 tiene una longitud de 4361 pares de bases [1] y tiene dos genes de resistencia a antibióticos : el gen bla que codifica la proteína de resistencia a la ampicilina (Amp R ) y el gen tetA que codifica la proteína de resistencia a la tetraciclina (Tet R ). Contiene el origen de replicación de pMB1 y el gen rop , que codifica un restrictor del número de copias del plásmido . El plásmido tiene sitios de restricción únicos para más de cuarenta enzimas de restricción . Once de estos cuarenta sitios se encuentran dentro del gen Tet R. Hay dos sitios para las enzimas de restricción HindIII y ClaI dentro del promotor del gen Tet R. Hay seis sitios de restricción clave dentro del gen Amp R. La fuente de estos genes de resistencia a antibióticos es pSC101 para la tetraciclina y RSF2124 para la ampicilina. [2]
La secuencia circular está numerada de tal manera que 0 es el centro del sitio único EcoRI y el recuento aumenta a través del gen Tet R. Si tenemos que eliminar la ampicilina, por ejemplo, debemos usar endonucleasas de restricción o tijeras moleculares contra PstI y entonces pBR322 se volverá anti-resistente a la ampicilina. El mismo proceso de inactivación insercional se puede aplicar a la tetraciclina. El gen Amp R es la penicilina beta-lactamasa . Los promotores P1 y P3 son para el gen de la beta-lactamasa. P3 es el promotor natural, y P1 se crea artificialmente mediante la ligación de dos fragmentos de ADN diferentes para crear pBR322. P2 está en la misma región que P1, pero está en la cadena opuesta e inicia la transcripción en la dirección del gen de resistencia a la tetraciclina. [3]
Los primeros experimentos de clonación pueden realizarse utilizando plásmidos naturales como ColE1 y pSC101 . Cada uno de estos plásmidos puede tener sus ventajas y desventajas. Por ejemplo, el plásmido ColE1 y sus derivados tienen la ventaja de un mayor número de copias y permiten la amplificación del plásmido con cloranfenicol para producir un alto rendimiento de plásmido, sin embargo, la detección de inmunidad a la colicina E1 no es técnicamente simple. [4] El plásmido pSC101, un plásmido natural de Salmonella panama , [5] confiere resistencia a la tetraciclina , lo que permite un proceso de detección más simple con selección de antibióticos, pero es un plásmido de bajo número de copias que no da un alto rendimiento de plásmido. Otro plásmido, RSF 2124, que es un derivado de ColE1, confiere resistencia a la ampicilina pero es más grande.
Muchos otros plásmidos fueron construidos artificialmente para crear uno que fuera ideal para fines de clonación, y pBR322 fue considerado el más versátil por muchos y por lo tanto fue el más utilizado. [4] Tiene dos genes de resistencia a antibióticos, como marcadores seleccionables , y una serie de sitios de restricción únicos convenientes que lo hicieron adecuado como un vector de clonación . El plásmido fue construido con material genético de 3 fuentes principales: el gen de resistencia a la tetraciclina de pSC101, el gen de resistencia a la ampicilina de RSF 2124 y los elementos de replicación de pMB1, un pariente cercano del plásmido ColE1 . [6] [7]
Desde entonces, se han construido una gran cantidad de otros plásmidos basados en pBR322 diseñados específicamente para una amplia variedad de propósitos. [8] [9] Los ejemplos incluyen la serie pUC de plásmidos. [10] La mayoría de los vectores de expresión para la expresión de proteínas extracromosómicas y los vectores lanzadera contienen el origen de replicación pBR322, y los fragmentos de pBR322 son muy populares en la construcción de vectores lanzadera o binarios intraespecies y vectores para la integración y escisión dirigidas de ADN del cromosoma. [11]
La secuencia en pBR322 es [3]