



La inactivación del cromosoma X (también llamada lionización , en honor a la genetista inglesa Mary Lyon ) es un proceso mediante el cual una de las copias del cromosoma X se inactiva en las hembras de los mamíferos therian . El cromosoma X inactivo se silencia al empaquetarse en una estructura transcripcionalmente inactiva llamada heterocromatina . Como casi todas las hembras de mamíferos tienen dos cromosomas X, la inactivación de X les impide tener el doble de productos genéticos del cromosoma X que los machos, que solo poseen una copia del cromosoma X (ver compensación de dosis ).
La elección de qué cromosoma X se inactivará en una célula embrionaria particular es aleatoria en mamíferos placentarios como los humanos, pero una vez que se inactiva un cromosoma X permanecerá inactivo durante toda la vida de la célula y sus descendientes en el organismo (su línea celular). ). El resultado es que la elección del cromosoma X inactivado en todas las células del organismo es una distribución aleatoria, a menudo con aproximadamente la mitad de las células que tienen el cromosoma X paterno inactivado y la otra mitad con un cromosoma X materno inactivado; pero comúnmente, la inactivación de X se distribuye de manera desigual entre las líneas celulares dentro de un organismo ( inactivación de X sesgada ).
A diferencia de la inactivación aleatoria de X en los mamíferos placentarios, la inactivación en los marsupiales se aplica exclusivamente al cromosoma X de origen paterno.
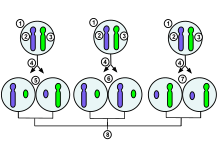
Los párrafos siguientes se refieren únicamente a los roedores y no reflejan el XI en la mayoría de los mamíferos. La inactivación del cromosoma X es parte del ciclo de activación del cromosoma X a lo largo de la vida femenina. El óvulo y el cigoto fertilizado inicialmente utilizan transcripciones maternas, y todo el genoma embrionario se silencia hasta la activación del genoma cigótico . A partir de entonces, todas las células de ratón se someten a una inactivación temprana impresa del cromosoma X de origen paterno en embriones en estadio de 4 a 8 células . [3] [4] [5] [6] Los tejidos extraembrionarios (que dan lugar a la placenta y otros tejidos que sostienen al embrión) retienen esta inactivación impresa temprana y, por lo tanto, solo el cromosoma X materno está activo en estos tejidos.
En el blastocisto temprano , esta inactivación inicial del X impreso se revierte en las células de la masa celular interna (que dan lugar al embrión), y en estas células ambos cromosomas X se vuelven activos nuevamente. Luego, cada una de estas células inactiva de forma independiente y aleatoria una copia del cromosoma X. [5] Este evento de inactivación es irreversible durante la vida del individuo, con la excepción de la línea germinal. En la línea germinal femenina , antes de la entrada meiótica, la inactivación de X se invierte, de modo que después de la meiosis todos los ovocitos haploides contienen un único cromosoma X activo.
Xi marca el cromosoma X inactivo y Xa el activo. X P denota el cromosoma X paterno y X M denota el cromosoma X materno. Cuando el óvulo (que lleva X M ) es fertilizado por un espermatozoide (que lleva Y o XP ) , se forma un cigoto diploide. Desde el cigoto, pasando por la etapa adulta, hasta la siguiente generación de óvulos, el cromosoma X sufre los siguientes cambios:
El ciclo de activación de X se ha estudiado mejor en ratones, pero existen múltiples estudios en humanos. Como la mayor parte de la evidencia proviene de ratones, el esquema anterior representa los eventos en ratones. La finalización de la meiosis se simplifica aquí para mayor claridad. Los pasos 1 a 4 se pueden estudiar en embriones fertilizados in vitro y en células madre diferenciadas; La reactivación X ocurre en el embrión en desarrollo y los pasos posteriores (6 a 7) dentro del cuerpo femenino, por lo que es mucho más difícil de estudiar.
El momento de cada proceso depende de la especie y, en muchos casos, el momento preciso se debate activamente. [Toda la parte del momento humano de la inactivación de X en esta tabla es muy cuestionable y debe eliminarse hasta que esté debidamente fundamentada por datos empíricos]
Los descendientes de cada célula que inactivó un cromosoma X en particular también desactivarán ese mismo cromosoma. Este fenómeno, que se puede observar en la coloración de los gatos carey cuando las hembras son heterocigotas para el gen del pigmento ligado al cromosoma X , no debe confundirse con el mosaicismo , que es un término que se refiere específicamente a las diferencias en el genotipo de varias poblaciones celulares en el mismo individuo; La inactivación del cromosoma X, que es un cambio epigenético que da como resultado un fenotipo diferente, no es un cambio a nivel genotípico . Por lo tanto, para una célula o linaje individual, la inactivación es sesgada o " no aleatoria ", y esto puede dar lugar a síntomas leves en mujeres "portadoras" de trastornos genéticos ligados al cromosoma X. [12]
Las mujeres típicas poseen dos cromosomas X, y en cualquier célula determinada, un cromosoma estará activo (designado como Xa) y el otro estará inactivo (Xi). Sin embargo, los estudios de individuos con copias adicionales del cromosoma X muestran que en las células con más de dos cromosomas X todavía hay un solo Xa y todos los cromosomas X restantes están inactivados. Esto indica que el estado predeterminado del cromosoma X en las mujeres es la inactivación, pero siempre se selecciona un cromosoma X para permanecer activo.
Se entiende que la inactivación del cromosoma X es un proceso aleatorio que ocurre aproximadamente en el momento de la gastrulación en el epiblasto (células que darán lugar al embrión). Los cromosomas X maternos y paternos tienen la misma probabilidad de inactivación. Esto sugeriría que se esperaría que las mujeres padecieran trastornos ligados al cromosoma X aproximadamente un 50% más frecuentemente que los hombres (porque las mujeres tienen dos cromosomas X, mientras que los hombres sólo tienen uno); sin embargo, en realidad, la incidencia de estos trastornos en las mujeres es mucho menor. Una explicación para esta disparidad es que entre el 12% y el 20% [13] de los genes del cromosoma X inactivado siguen expresados, lo que proporciona a las mujeres una protección adicional contra genes defectuosos codificados por el cromosoma X. ¿ Algunos que? ] sugieren que esta disparidad debe ser evidencia de inactivación preferencial (no aleatoria). La inactivación preferencial del cromosoma X paterno ocurre tanto en marsupiales como en linajes celulares que forman las membranas que rodean al embrión, [14] mientras que en los mamíferos placentarios el cromosoma X derivado materno o paterno puede inactivarse en diferentes líneas celulares. [15]
El período de inactivación del cromosoma X explica esta disparidad. La inactivación se produce en el epiblasto durante la gastrulación, que da origen al embrión. [16] La inactivación ocurre a nivel celular, lo que resulta en una expresión en mosaico, en la que parches de células tienen un cromosoma X materno inactivo, mientras que otros parches tienen un cromosoma X paterno inactivo. Por ejemplo, una mujer heterocigota con hemofilia (una enfermedad ligada al cromosoma X) tendría aproximadamente la mitad de sus células hepáticas funcionando correctamente, lo que suele ser suficiente para garantizar una coagulación sanguínea normal. [17] [18] El azar podría dar como resultado un número significativamente mayor de células disfuncionales; sin embargo, tales extremos estadísticos son poco probables. Las diferencias genéticas en el cromosoma también pueden hacer que un cromosoma X tenga más probabilidades de sufrir una inactivación. Además, si un cromosoma X tiene una mutación que impide su crecimiento o lo vuelve inviable, las células que inactivaron aleatoriamente ese X tendrán una ventaja selectiva sobre las células que inactivaron aleatoriamente el alelo normal. Por lo tanto, aunque la inactivación es inicialmente aleatoria, las células que inactivan un alelo normal (dejando activo el alelo mutado) eventualmente crecerán demasiado y serán reemplazadas por células funcionalmente normales en las que casi todas tienen el mismo cromosoma X activado. [17]
Se plantea la hipótesis de que existe un "factor de bloqueo" codificado de forma autosómica que se une al cromosoma X e impide su inactivación. [19] El modelo postula que existe un factor de bloqueo limitante, por lo que una vez que la molécula del factor de bloqueo disponible se une a un cromosoma X, los cromosomas X restantes no están protegidos de la inactivación. Este modelo está respaldado por la existencia de un único Xa en células con muchos cromosomas X y por la existencia de dos cromosomas X activos en líneas celulares con el doble de autosomas normales. [20]
Las secuencias en el centro de inactivación X ( XIC ), presente en el cromosoma X, controlan el silenciamiento del cromosoma X. Se predice que el hipotético factor de bloqueo se unirá a secuencias dentro del XIC.
El efecto de la heterocigosidad X femenina es evidente en algunos rasgos localizados, como el patrón de pelaje único de un gato calicó . Sin embargo, puede resultar más difícil comprender plenamente la expresión de rasgos no localizados en estas hembras, como la expresión de enfermedades.
Dado que los hombres solo tienen una copia del cromosoma X, todos los genes cromosómicos X expresados (o alelos , en el caso de múltiples formas variantes para un gen determinado en la población) se encuentran en esa copia del cromosoma. Las mujeres, sin embargo, expresarán principalmente los genes o alelos ubicados en la copia del cromosoma X que permanece activa. Considerando la situación de un gen o múltiples genes que causan diferencias individuales en un fenotipo particular (es decir, que causan la variación observada en la población para ese fenotipo), en mujeres homocigotas no importa particularmente qué copia del cromosoma está inactivada, ya que los alelos En ambas copias son iguales. Sin embargo, en mujeres que son heterocigotas en los genes causales, la inactivación de una copia del cromosoma sobre la otra puede tener un impacto directo en su valor fenotípico. Debido a este fenómeno, se observa un aumento en la variación fenotípica en mujeres que son heterocigotas en el gen o genes involucrados que en mujeres que son homocigotas en ese gen o esos genes. [21] Hay muchas formas diferentes en que puede desarrollarse la variación fenotípica. En muchos casos, las mujeres heterocigotas pueden ser asintomáticas o presentar solo síntomas menores de un trastorno determinado, como la adrenoleucodistrofia ligada al cromosoma X. [22]
La diferenciación del fenotipo en mujeres heterocigotas se ve favorecida por la presencia de un sesgo de inactivación de X. Normalmente, cada cromosoma X está silenciado en la mitad de las células, pero este proceso se distorsiona cuando se produce la inactivación preferencial de un cromosoma. Se cree que el sesgo ocurre por casualidad o por una característica física de un cromosoma que puede hacer que se silencie con mayor o menor frecuencia, como una mutación desfavorable. [23] [24]
En promedio, cada cromosoma X está inactivado en la mitad de las células, aunque entre el 5 y el 20% de las mujeres muestran un sesgo de inactivación del cromosoma X. [23] En los casos en los que hay sesgo, puede ocurrir una amplia gama de expresión de síntomas, lo que da como resultado una expresión que varía de menor a grave dependiendo de la proporción de sesgo. Se observó un caso extremo de esto cuando gemelas monocigóticas tenían una variación extrema en la expresión de la enfermedad de Menkes (un trastorno ligado al cromosoma X), lo que resultó en la muerte de una gemela mientras que la otra permanecía asintomática. [25]
Se cree que el sesgo de la inactivación del cromosoma X podría deberse a problemas en el mecanismo que causa la inactivación o a problemas en el cromosoma mismo. [23] [24] Sin embargo, el vínculo entre el fenotipo y el sesgo aún se cuestiona y debe examinarse caso por caso. Un estudio que analizó a mujeres sintomáticas y asintomáticas que eran heterocigotas para las distrofias musculares de Duchenne y Becker (DMD) no encontró ningún vínculo aparente entre la expresión de la transcripción y la inactivación de X sesgada. El estudio sugiere que ambos mecanismos están regulados de forma independiente y que hay otros factores desconocidos en juego. [26]
El centro de inactivación de X (o simplemente XIC) en el cromosoma X es necesario y suficiente para provocar la inactivación de X. Las translocaciones cromosómicas que colocan el XIC en un autosoma conducen a la inactivación del autosoma, y los cromosomas X que carecen del XIC no se inactivan. [27] [28]
El XIC contiene cuatro genes de ARN no traducidos , Xist , Tsix , Jpx y Ftx , que participan en la inactivación de X. El XIC también contiene sitios de unión para proteínas reguladoras conocidas y desconocidas . [29]
El gen del transcrito específico inactivo del X ( Xist ) codifica un gran ARN no codificante que se encarga de mediar en el silenciamiento específico del cromosoma X a partir del cual se transcribe. [30] El cromosoma X inactivo está recubierto por ARN Xist, [31] mientras que el Xa no (consulte la figura a la derecha). Los cromosomas X que carecen del gen Xist no se pueden inactivar. [32] Colocar y expresar artificialmente el gen Xist en otro cromosoma conduce al silenciamiento de ese cromosoma. [33] [27]
Antes de la inactivación, ambos cromosomas X expresan débilmente el ARN Xist del gen Xist. Durante el proceso de inactivación, el futuro Xa deja de expresar Xist, mientras que el futuro Xi aumenta drásticamente la producción de ARN de Xist. En el futuro Xi, el ARN Xist recubre progresivamente el cromosoma, extendiéndose desde el XIC; [33] el ARN Xist no se localiza en Xa. El silenciamiento de los genes a lo largo del Xi se produce poco después del recubrimiento por el ARN Xist.
Al igual que Xist, el gen Tsix codifica un ARN grande que no se cree que codifique una proteína. El ARN Tsix se transcribe en antisentido a Xist, lo que significa que el gen Tsix se superpone al gen Xist y se transcribe en la hebra opuesta de ADN del gen Xist. [28] Tsix es un regulador negativo de Xist; Los cromosomas X que carecen de expresión Tsix (y por lo tanto tienen altos niveles de transcripción Xist) se inactivan con mucha más frecuencia que los cromosomas normales.
Al igual que Xist, antes de la inactivación, ambos cromosomas X expresan débilmente el ARN Tsix del gen Tsix. Tras el inicio de la inactivación de X, el futuro Xi deja de expresar el ARN de Tsix (y aumenta la expresión de Xist), mientras que Xa continúa expresando Tsix durante varios días.
Rep A es un ARN largo no codificante que funciona con otro ARN largo no codificante, Xist, para la inactivación de X. Rep A inhibe la función de Tsix, el antisentido de Xist, junto con la eliminación de la expresión de Xite. Promueve la metilación de la región Tsix al atraer PRC2 y así inactivar uno de los cromosomas X. [29]
El cromosoma X inactivo no expresa la mayoría de sus genes, a diferencia del cromosoma X activo. Esto se debe al silenciamiento de Xi por la heterocromatina represiva , que compacta el ADN de Xi e impide la expresión de la mayoría de los genes.
En comparación con el Xa, el Xi tiene altos niveles de metilación del ADN , bajos niveles de acetilación de histonas , bajos niveles de metilación de histona H3 lisina-4 y altos niveles de metilación de histona H3 lisina-9 y marca de metilación H3 lisina-27 que se coloca. por el complejo PRC2 reclutado por Xist , todos los cuales están asociados con el silenciamiento de genes. [34] PRC2 regula la compactación y remodelación de la cromatina en varios procesos, incluida la respuesta al daño del ADN . [35] Además, una variante de histona llamada macroH2A ( H2AFY ) se encuentra exclusivamente en los nucleosomas a lo largo del Xi. [36] [37]
El ADN empaquetado en heterocromatina, como el Xi, está más condensado que el ADN empaquetado en eucromatina , como el Xa. El X inactivo forma un cuerpo discreto dentro del núcleo llamado cuerpo de Barr . [38] El cuerpo de Barr generalmente está ubicado en la periferia del núcleo , se replica tardíamente dentro del ciclo celular y, como contiene Xi, contiene modificaciones de heterocromatina y el ARN Xist.
Una fracción de los genes a lo largo del cromosoma X escapa a la inactivación en el Xi. El gen Xist se expresa en niveles elevados en Xi y no se expresa en Xa. [39] Muchos otros genes escapan a la inactivación; algunos se expresan por igual en Xa y Xi, y otros, aunque se expresan en ambos cromosomas, todavía se expresan predominantemente en Xa. [40] [41] [42] Hasta una cuarta parte de los genes del Xi humano son capaces de escapar. [40] Los estudios en ratones sugieren que en cualquier tipo de célula determinada, entre el 3% y el 15% de los genes escapan a la inactivación, y que la identidad genética que escapa varía entre los tejidos. [41] [42]
Muchos de los genes que escapan a la inactivación están presentes a lo largo de regiones del cromosoma X que, a diferencia de la mayoría del cromosoma X, contienen genes también presentes en el cromosoma Y. Estas regiones se denominan regiones pseudoautosómicas , ya que los individuos de ambos sexos recibirán dos copias de cada gen en estas regiones (como un autosoma), a diferencia de la mayoría de los genes de los cromosomas sexuales. Dado que los individuos de ambos sexos recibirán dos copias de cada gen en una región pseudoautosómica , no se necesita compensación de dosis para las mujeres, por lo que se postula que estas regiones del ADN han desarrollado mecanismos para escapar de la inactivación de X. Los genes de las regiones pseudoautosómicas de Xi no tienen las modificaciones típicas de Xi y tienen poco ARN Xist unido.
La existencia de genes a lo largo del X inactivo que no están silenciados explica los defectos en humanos con números anormales del cromosoma X, como el síndrome de Turner (X0, causado por el gen SHOX [43] ) o el síndrome de Klinefelter (XXY). En teoría, la inactivación del cromosoma X debería eliminar las diferencias en la dosis de genes entre los individuos afectados y los individuos con un complemento cromosómico normal. Sin embargo, en los individuos afectados, la inactivación de X es incompleta y la dosis de estos genes no silenciados diferirá a medida que escapan de la inactivación de X, similar a una aneuploidía autosómica .
Se desconocen los mecanismos precisos que controlan el escape de la inactivación de X, pero se ha demostrado que las regiones silenciadas y de escape tienen marcas de cromatina distintas. [41] [44] Se ha sugerido que el escape de la inactivación de X podría estar mediado por la expresión de ARN largo no codificante (lncRNA) dentro de los dominios cromosómicos que se escapan. [2]
Stanley Michael Gartler utilizó la inactivación del cromosoma X para demostrar el origen clonal de los cánceres. El examen de tejidos normales y tumores de mujeres heterocigotas en busca de isoenzimas del gen G6PD ligado al sexo demostró que las células tumorales de dichos individuos expresan sólo una forma de G6PD, mientras que los tejidos normales están compuestos de una mezcla casi igual de células que expresan los dos fenotipos diferentes. Este patrón sugiere que una sola célula, y no una población, se convierte en cáncer. [45] Sin embargo, se ha demostrado que este patrón es incorrecto para muchos tipos de cáncer, lo que sugiere que algunos cánceres pueden ser de origen policlonal. [46]
Además, medir el estado de metilación (inactivación) del receptor polimórfico de andrógenos humanos (HUMARA) ubicado en el cromosoma X se considera el método más preciso para evaluar la clonalidad en biopsias de cáncer femenino. [47] Se analizó una gran variedad de tumores mediante este método; algunos, como el carcinoma de células renales, [48] se encontraron monoclonales, mientras que otros (por ejemplo, el mesotelioma [49] ) se informaron como policlonales.
Los investigadores también han investigado el uso de la inactivación del cromosoma X para silenciar la actividad de los cromosomas autosómicos. Por ejemplo, Jiang et al. insertó una copia del gen Xist en una copia del cromosoma 21 en células madre derivadas de un individuo con trisomía 21 ( síndrome de Down ). [50] El gen Xist insertado induce la formación de cuerpos de Barr, desencadena modificaciones estables de la heterocromatina y silencia la mayoría de los genes en la copia adicional del cromosoma 21. En estas células madre modificadas, el silenciamiento del gen mediado por Xist parece revertir algunos de los defectos. asociado con el síndrome de Down.
En 1959 Susumu Ohno demostró que los dos cromosomas X de los mamíferos eran diferentes: uno parecía similar a los autosomas ; el otro era condensado y heterocromático. [51] Este hallazgo sugirió, de forma independiente a dos grupos de investigadores, que uno de los cromosomas X experimentó inactivación.
En 1961, Mary Lyon propuso la inactivación aleatoria de un cromosoma X femenino para explicar el fenotipo moteado de ratones hembra heterocigotos para los genes del color del pelaje . [52] La hipótesis de Lyon también explicó los hallazgos de que una copia del cromosoma X en las células femeninas estaba altamente condensada y que los ratones con solo una copia del cromosoma X se desarrollaban como hembras infértiles. Esto sugirió [53] a Ernest Beutler , que estudiaba mujeres heterocigotas para detectar la deficiencia de glucosa-6-fosfato deshidrogenasa (G6PD), que había dos poblaciones de eritrocitos de glóbulos rojos en tales heterocigotos: células deficientes y células normales, [54] dependiendo de si el El cromosoma X inactivado (en el núcleo de la célula precursora de los glóbulos rojos) contiene el alelo G6PD normal o defectuoso.
Figura 1 El ARN Xist abarca el X del cual se transcribe.
{{cite journal}}: Enlace externo en |quote=Tabla 1 Características del territorio X inactivo
{{cite journal}}: Enlace externo en |quote=