En bioquímica , una nucleasa (también conocida arcaicamente como nucleodespolimerasa o polinucleotidasa ) es una enzima capaz de escindir los enlaces fosfodiéster que unen los nucleótidos para formar ácidos nucleicos . Las nucleasas afectan de diversas formas a las roturas de cadena simple y doble en sus moléculas objetivo. En los organismos vivos, son maquinaria esencial para muchos aspectos de la reparación del ADN . Los defectos en ciertas nucleasas pueden causar inestabilidad genética o inmunodeficiencia . [1] Las nucleasas también se utilizan ampliamente en la clonación molecular . [2]
Existen dos clasificaciones principales basadas en el lugar de actividad. Las exonucleasas digieren los ácidos nucleicos desde los extremos. Las endonucleasas actúan en regiones en el medio de las moléculas diana. Se subdividen en desoxirribonucleasas y ribonucleasas . Las primeras actúan sobre el ADN , las segundas sobre el ARN . [2]
A finales de la década de 1960, los científicos Stuart Linn y Werner Arber aislaron ejemplos de los dos tipos de enzimas responsables de la restricción del crecimiento de fagos en las bacterias Escherichia coli ( E. coli ). [3] [4] Una de estas enzimas agregó un grupo metilo al ADN, generando ADN metilado , mientras que la otra escindió el ADN no metilado en una amplia variedad de ubicaciones a lo largo de la longitud de la molécula. El primer tipo de enzima se llamó " metilasa " y la otra " nucleasa de restricción ". Estas herramientas enzimáticas fueron importantes para los científicos que estaban reuniendo las herramientas necesarias para " cortar y pegar " moléculas de ADN. Lo que se necesitaba entonces era una herramienta que cortara el ADN en sitios específicos, en lugar de en sitios aleatorios a lo largo de la longitud de la molécula, para que los científicos pudieran cortar las moléculas de ADN de una manera predecible y reproducible.
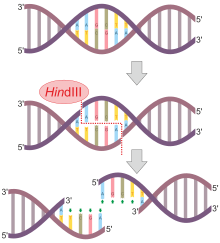
Un avance importante se produjo cuando HO Smith , KW Wilcox y TJ Kelly , trabajando en la Universidad Johns Hopkins en 1968, aislaron y caracterizaron la primera nucleasa de restricción cuyo funcionamiento dependía de una secuencia de nucleótidos de ADN específica . Trabajando con la bacteria Haemophilus influenzae , este grupo aisló una enzima, llamada HindII, que siempre corta las moléculas de ADN en un punto particular dentro de una secuencia específica de seis pares de bases. Descubrieron que la enzima Hind II siempre corta directamente en el centro de esta secuencia (entre el 3.º y el 4.º par de bases).
La mayoría de las nucleasas están clasificadas por el número de la Comisión de Enzimas del "Comité de Nomenclatura de la Unión Internacional de Bioquímica y Biología Molecular " como hidrolasas (número CE 3). Las nucleasas pertenecen, al igual que la fosfodiesterasa , la lipasa y la fosfatasa, a las esterasas (número CE 3.1), un subgrupo de las hidrolasas. Las esterasas a las que pertenecen las nucleasas están clasificadas con los números CE 3.1.11 - 3.1.31.
La estructura primaria de las nucleasas está, en general, poco conservada y mínimamente conservada en los sitios activos, cuyas superficies están formadas principalmente por residuos de aminoácidos ácidos y básicos. Las nucleasas se pueden clasificar en familias de plegamiento. [5]
Una nucleasa debe asociarse con un ácido nucleico antes de poder escindir la molécula, lo que implica un cierto grado de reconocimiento. Las nucleasas emplean asociaciones específicas y no específicas en sus modos de reconocimiento y unión. Ambos modos desempeñan papeles importantes en los organismos vivos, especialmente en la reparación del ADN. [7]
Las endonucleasas no específicas que participan en la reparación del ADN pueden escanear el ADN en busca de secuencias diana o daños . Dicha nucleasa se difunde a lo largo del ADN hasta que encuentra un objetivo, en el que los residuos de su sitio activo interactúan con los grupos químicos del ADN. En el caso de endonucleasas como EcoRV , BamHI y PvuII, esta unión no específica implica interacciones electrostáticas entre el área superficial mínima de la proteína y el ADN. Esta asociación débil deja la forma general del ADN sin deformar, permaneciendo en forma B. [7 ]
AEn cambio, la nucleasa específica del sitio forma asociaciones mucho más fuertes. Atrae el ADN hacia el surco profundo de su dominio de unión al ADN . Esto produce una deformación significativa de la estructura terciaria del ADN y se logra con superficies ricas en residuos básicos (cargados positivamente). Participa en una amplia interacción electrostática con el ADN. [7]
Algunas nucleasas que participan en la reparación del ADN presentan una especificidad de secuencia parcial, pero la mayoría no son específicas y reconocen anomalías estructurales producidas en la cadena principal del ADN por desajustes de pares de bases . [7]
Para más detalles, véase endonucleasa de colgajo .
Existen más de 900 enzimas de restricción, algunas específicas de la secuencia y otras no, que se han aislado de más de 230 cepas de bacterias desde el descubrimiento inicial de Hind II. Estas enzimas de restricción generalmente tienen nombres que reflejan su origen: la primera letra del nombre proviene del género y las dos siguientes letras provienen de la especie de la célula procariota de la que se aislaron. Por ejemplo, Eco RI proviene de la bacteria Escherichia coli RY13, mientras que HindII proviene de la cepa Rd de Haemophilus influenzae . Los números que siguen a los nombres de las nucleasas indican el orden en el que se aislaron las enzimas de cepas individuales de bacterias: Eco RI , Eco RII .
Una endonucleasa de restricción funciona "escaneando" la longitud de una molécula de ADN. Una vez que encuentra su secuencia de reconocimiento específica, se une a la molécula de ADN y realiza un corte en cada una de las dos cadenas principales de azúcar y fosfato. Las posiciones de estos dos cortes, tanto en relación entre sí como con la secuencia de reconocimiento en sí, están determinadas por la identidad de la endonucleasa de restricción. Diferentes endonucleasas producen diferentes conjuntos de cortes, pero una endonucleasa siempre cortará una secuencia de bases particular de la misma manera, sin importar sobre qué molécula de ADN esté actuando. Una vez que se han realizado los cortes, la molécula de ADN se romperá en fragmentos.
No todas las endonucleasas de restricción cortan simétricamente y dejan extremos romos como el caso de Hind II descrito anteriormente. Muchas endonucleasas cortan las cadenas principales del ADN en posiciones que no están directamente opuestas entre sí, creando salientes. Por ejemplo, la nucleasa Eco RI tiene la secuencia de reconocimiento 5'—GAATTC—3'.
Cuando la enzima encuentra esta secuencia, corta cada cadena principal entre los residuos de base G y A más cercanos. Una vez que se han realizado los cortes, los fragmentos resultantes se mantienen unidos solo por los enlaces de hidrógeno relativamente débiles que mantienen unidas las bases complementarias entre sí. La debilidad de estos enlaces permite que los fragmentos de ADN se separen entre sí. Cada fragmento resultante tiene un extremo 5' saliente compuesto de bases no apareadas. Otras enzimas crean cortes en la cadena principal del ADN que dan como resultado extremos 3' salientes. Los extremos salientes, tanto 3' como 5', a veces se denominan " extremos pegajosos " porque tienden a unirse con secuencias complementarias de bases. En otras palabras, si una longitud de bases no apareadas 5'—AATT—3'encuentra otra longitud desapareada con la secuencia, 3'—TTAA—5'se unirán entre sí; son "pegajosas" entre sí. Luego, se utiliza la enzima ligasa para unir las cadenas principales de fosfato de las dos moléculas. El origen celular, o incluso el origen de la especie, de los extremos pegajosos no afecta su adhesividad. Cualquier par de secuencias complementarias tenderá a unirse, incluso si una de las secuencias proviene de un fragmento de ADN humano y la otra de un fragmento de ADN bacteriano. De hecho, es esta cualidad de adherencia la que permite la producción de moléculas de ADN recombinante, moléculas que están compuestas de ADN de diferentes fuentes, y que ha dado origen a la tecnología de la ingeniería genética .
Dado que todas las células dependen del ADN como medio de información genética, el control de calidad genética es una función esencial de todos los organismos. La replicación del ADN es un proceso propenso a errores, y las moléculas de ADN en sí mismas son vulnerables a la modificación por muchos factores estresantes metabólicos y ambientales. Ejemplos ubicuos incluyen especies reactivas de oxígeno , ultravioleta cercano y radiación ionizante . Muchas nucleasas participan en la reparación del ADN al reconocer los sitios dañados y escindirlos del ADN circundante. Estas enzimas funcionan de forma independiente o en complejos . La mayoría de las nucleasas involucradas en la reparación del ADN no son específicas de la secuencia. Reconocen los sitios dañados a través de la deformación de la estructura secundaria del ADN bicatenario (dsADN). [5]
Durante la replicación del ADN , las ADN polimerasas alargan nuevas hebras de ADN contra hebras de plantilla complementarias. La mayoría de las ADN polimerasas comprenden dos dominios enzimáticos diferentes : una polimerasa y una exonucleasa de corrección de errores . La polimerasa alarga la nueva hebra en la dirección 5' → 3'. La exonucleasa elimina los nucleótidos erróneos de la misma hebra en la dirección 3' → 5'. Esta actividad de la exonucleasa es esencial para la capacidad de una ADN polimerasa de corregir errores. Las deleciones que inactivan o eliminan estas nucleasas aumentan las tasas de mutación y mortalidad en los microbios afectados y el cáncer en ratones. [8]
Muchas formas de daño del ADN detienen la progresión de la horquilla de replicación , lo que hace que las polimerasas de ADN y la maquinaria asociada abandonen la horquilla. Luego debe ser procesada por proteínas específicas de la horquilla. La más notable es MUS81 , cuyas deleciones provocan sensibilidad a los daños por rayos UV o por metilación en la levadura , además de defectos meióticos. [5]
Una tarea omnipresente en las células es la eliminación de los cebadores de ARN del fragmento de Okazaki de la replicación. La mayoría de estos cebadores son extirpados del ADN de cadena retrasada recién sintetizado por endonucleasas de la familia RNasa H. En eucariotas y en arqueas , la endonucleasa de flap FEN1 también participa en el procesamiento de los fragmentos de Okazaki. [5]
La reparación de los errores de apareamiento del ADN en cualquier organismo se lleva a cabo mediante un conjunto de endonucleasas específicas para los errores de apareamiento. En los procariotas, esta función la desempeñan principalmente MutSLH y las proteínas asociadas con la reparación de parches muy cortos (reparación VSP).
El sistema MutSLH (que comprende MutS , MutL y MutH) corrige mutaciones puntuales y pequeños giros . MutS reconoce y se une a los desajustes, donde recluta a MutL y MutH. MutL media la interacción entre MutS y MutH, y mejora la actividad endonucleásica de este último. MutH reconoce 5'—GATC—3'los sitios hemimetilados y corta junto a Gla hebra no metilada (la hebra sintetizada más recientemente).
La reparación de VSP es iniciada por la endonucleasa Vsr. Corrige un T/Gdesajuste específico causado por la desaminación espontánea de citosinas metiladas a timinas. Vsr reconoce la secuencia , donde corta la cadena de ADN en el lado 5' de la timina desajusteada (subrayada en la secuencia anterior). Una de las exonucleasas RecJ, ExoVII o ExoI degrada entonces el sitio antes de que la ADN polimerasa resintetice el espacio en la cadena. [5]5'—CTWGG—3'
La formación de sitios AP es un fenómeno común en el ADN de doble cadena. Es el resultado de la hidrólisis espontánea y de la actividad de las glicosilasas del ADN como paso intermedio en la reparación por escisión de bases . Estos sitios AP son eliminados por las endonucleasas AP , que producen roturas de una sola cadena alrededor del sitio. [5]
La reparación por escisión de nucleótidos , que no debe confundirse con la reparación por escisión de bases, implica la eliminación y el reemplazo de nucleótidos dañados. Los casos de entrecruzamiento , aductos y lesiones (generadas por luz ultravioleta o especies reactivas de oxígeno ) pueden desencadenar esta vía de reparación. Los tramos cortos de ADN monocatenario que contienen dicho nucleótido dañado se eliminan del ADN dúplex mediante endonucleasas separadas que efectúan mellas antes y después del daño. Las deleciones o mutaciones que afectan a estas nucleasas provocan una mayor sensibilidad al daño ultravioleta y la carcinogénesis. Tales anomalías pueden incluso afectar el desarrollo neuronal.
En las bacterias, ambos cortes los realiza el complejo UvrB-UvrC . En la levadura en ciernes, Rad2 y el complejo Rad1-Rad10 realizan los cortes 5' y 3', respectivamente. En los mamíferos, los homólogos XPG y XPF - ERCC1 afectan a las mismas muescas respectivas. [9]
Las roturas de doble cadena , tanto intencionales como no intencionales, ocurren regularmente en las células. Las roturas no intencionales son generadas comúnmente por la radiación ionizante , varios agentes químicos exógenos y endógenos y horquillas de replicación detenidas. Las roturas intencionales se generan como intermediarios en la meiosis y la recombinación V(D)J , que se reparan principalmente a través de la recombinación homóloga y la unión de extremos no homólogos . Ambos casos requieren que los extremos en las roturas de doble cadena sean procesados por nucleasas antes de que pueda tener lugar la reparación. Una de estas nucleasas es Mre11 complejada con Rad50 . Las mutaciones de Mre11 pueden precipitar un trastorno similar a la ataxia-telangiectasia . [9]
La recombinación V(D)J implica la apertura de estructuras de tallo-bucles asociadas con roturas de doble cadena y la posterior unión de ambos extremos. El complejo Artemis-DNAPK cs participa en esta reacción. Aunque Artemis exhibe actividad de exonucleasa 5' → 3' ssDNA cuando está sola, su formación de complejos con DNA-PK cs permite el procesamiento endonucleásico de los tallo-bucles. Los defectos de cualquiera de las proteínas confieren inmunodeficiencia grave. [9]
Por otro lado, la recombinación homóloga implica dos dúplex de ADN homólogos conectados por bucles D o uniones de Holliday . En las bacterias, las endonucleasas como RuvC resuelven las uniones de Holliday en dos dsADN separados al escindir las uniones en dos sitios simétricos cerca del centro de unión. En eucariotas, FEN1 , XPF - ERCC1 y MUS81 escinden los bucles D, y Cce1 / Ydc2 procesa las uniones de Holliday en las mitocondrias. [9]
La frecuencia con la que una determinada nucleasa corta una molécula de ADN determinada depende de la complejidad del ADN y de la longitud de la secuencia de reconocimiento de la nucleasa; debido a la probabilidad estadística de encontrar las bases en un orden determinado por casualidad, una secuencia de reconocimiento más larga dará como resultado una digestión menos frecuente. Por ejemplo, se podría predecir que una determinada secuencia de cuatro bases (que corresponde al sitio de reconocimiento de una nucleasa hipotética) se produciría cada 256 pares de bases en promedio (donde 4^4=256), pero se esperaría que cualquier secuencia de seis bases se produjera una vez cada 4.096 pares de bases en promedio (4^6=4096).
Una familia única de nucleasas son las meganucleasas , que se caracterizan por tener secuencias de reconocimiento más grandes y, por lo tanto, menos comunes, que constan de 12 a 40 pares de bases. Estas nucleasas son particularmente útiles para la ingeniería genética y las aplicaciones de ingeniería genómica en organismos complejos como plantas y mamíferos, donde los genomas típicamente más grandes (con miles de millones de pares de bases) darían lugar a una digestión específica del sitio frecuente y perjudicial utilizando nucleasas tradicionales.