La fosforilación oxidativa (UK / ɒ k ˈ s ɪ d . ə . t ɪ v / , US / ˈ ɑː k . s ɪ ˌ d eɪ . t ɪ v / [1] ) o la fosforilación ligada al transporte de electrones u oxidación terminal es la Vía metabólica en la que las células utilizan enzimas para oxidar nutrientes , liberando así energía química para producir trifosfato de adenosina (ATP). En los eucariotas , esto ocurre dentro de las mitocondrias . Casi todos los organismos aeróbicos realizan fosforilación oxidativa. Esta vía es tan generalizada porque libera más energía que los procesos de fermentación alternativos , como la glucólisis anaeróbica .
La energía almacenada en los enlaces químicos de la glucosa es liberada por la célula en el ciclo del ácido cítrico , produciendo dióxido de carbono y los energéticos donadores de electrones NADH y FADH . La fosforilación oxidativa utiliza estas moléculas y O 2 para producir ATP , que se utiliza en toda la célula siempre que se necesita energía. Durante la fosforilación oxidativa, los electrones se transfieren desde los donadores de electrones a una serie de aceptores de electrones en una serie de reacciones redox que terminan en oxígeno, cuya reacción libera la mitad de la energía total. [2]
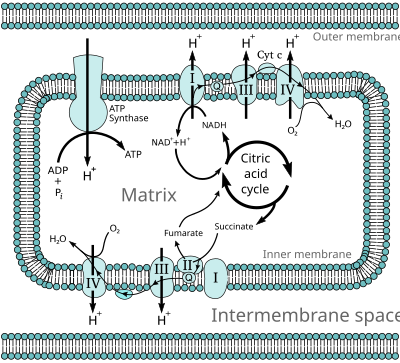
En los eucariotas , estas reacciones redox son catalizadas por una serie de complejos proteicos dentro de la membrana interna de las mitocondrias de la célula, mientras que, en los procariotas , estas proteínas se encuentran en la membrana externa de la célula. Estos conjuntos de proteínas enlazadas se denominan cadena de transporte de electrones . En los eucariotas están implicados cinco complejos proteicos principales, mientras que en los procariotas están presentes muchas enzimas diferentes, utilizando una variedad de donadores y aceptores de electrones.
La energía transferida por los electrones que fluyen a través de esta cadena de transporte de electrones se utiliza para transportar protones a través de la membrana mitocondrial interna , en un proceso llamado transporte de electrones . Esto genera energía potencial en forma de un gradiente de pH y el potencial eléctrico resultante a través de esta membrana. Esta reserva de energía se aprovecha cuando los protones regresan a través de la membrana y bajan por el gradiente de energía potencial, a través de una gran enzima llamada ATP sintasa en un proceso llamado quimiosmosis . La ATP sintasa utiliza la energía para transformar el adenosín difosfato (ADP) en adenosín trifosfato, en una reacción de fosforilación . La reacción es impulsada por el flujo de protones, que fuerza la rotación de una parte de la enzima. La ATP sintasa es un motor mecánico rotativo.
Aunque la fosforilación oxidativa es una parte vital del metabolismo, produce especies reactivas de oxígeno como el superóxido y el peróxido de hidrógeno , que conducen a la propagación de radicales libres , dañando las células y contribuyendo a enfermedades y, posiblemente, al envejecimiento y la senescencia . Las enzimas que llevan a cabo esta vía metabólica también son el objetivo de muchos fármacos y venenos que inhiben sus actividades.
La fosforilación oxidativa funciona mediante el uso de reacciones químicas que liberan energía para impulsar reacciones que requieren energía. Se dice que los dos conjuntos de reacciones están acoplados . Esto significa que uno no puede ocurrir sin el otro. La cadena de reacciones redox que impulsa el flujo de electrones a través de la cadena de transporte de electrones, desde donantes de electrones como el NADH hasta aceptores de electrones como el oxígeno y el hidrógeno (protones), es un proceso exergónico : libera energía, mientras que la síntesis de ATP es un proceso exergónico. Proceso endergónico , que requiere un aporte de energía. Tanto la cadena de transporte de electrones como la ATP sintasa están incrustadas en una membrana, y la energía se transfiere de la cadena de transporte de electrones a la ATP sintasa mediante movimientos de protones a través de esta membrana, en un proceso llamado quimiosmosis . [3] Una corriente de protones es impulsada desde el lado N negativo de la membrana hacia el lado P positivo a través de las enzimas que bombean protones de la cadena de transporte de electrones. El movimiento de los protones crea un gradiente electroquímico a través de la membrana, lo que se denomina fuerza motriz del protón . Tiene dos componentes: una diferencia en la concentración de protones (un gradiente de H + , Δ pH ) y una diferencia de potencial eléctrico , teniendo el lado N una carga negativa. [4]
La ATP sintasa libera esta energía almacenada al completar el circuito y permitir que los protones fluyan a favor del gradiente electroquímico, de regreso al lado N de la membrana. [5] El gradiente electroquímico impulsa la rotación de parte de la estructura de la enzima y acopla este movimiento a la síntesis de ATP.
Los dos componentes de la fuerza motriz del protón son termodinámicamente equivalentes: en las mitocondrias, la mayor parte de la energía la proporciona el potencial; En las bacterias alcalófilas , la energía eléctrica incluso tiene que compensar una diferencia inversa del pH. A la inversa, los cloroplastos operan principalmente con ΔpH. Sin embargo, también requieren un pequeño potencial de membrana para la cinética de síntesis de ATP. En el caso de la fusobacterium Propionigenium modestum , impulsa la contrarrotación de las subunidades a y c del motor F O de la ATP sintasa. [4]
La cantidad de energía liberada por la fosforilación oxidativa es alta, en comparación con la cantidad producida por la fermentación anaeróbica . La glucólisis produce sólo 2 moléculas de ATP, pero entre 30 y 36 ATP se producen mediante la fosforilación oxidativa de las 10 moléculas de NADH y 2 de succinato, que se obtienen al convertir una molécula de glucosa en dióxido de carbono y agua, [6] mientras que cada ciclo de beta oxidación de un ácido graso produce alrededor de 14 ATP. Estos rendimientos de ATP son valores máximos teóricos; en la práctica, algunos protones atraviesan la membrana, lo que reduce la producción de ATP. [7]
La cadena de transporte de electrones transporta protones y electrones, pasa electrones de los donantes a los aceptores y transporta protones a través de una membrana. Estos procesos utilizan moléculas de transferencia tanto solubles como unidas a proteínas. En las mitocondrias, los electrones son transferidos dentro del espacio intermembrana mediante la proteína de transferencia de electrones citocromo c, soluble en agua . [8] Este transporta sólo electrones, y estos se transfieren mediante la reducción y oxidación de un átomo de hierro que la proteína mantiene dentro de un grupo hemo en su estructura. El citocromo c también se encuentra en algunas bacterias, donde se ubica dentro del espacio periplásmico . [9]
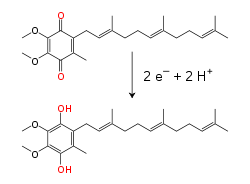
Dentro de la membrana mitocondrial interna, la coenzima Q10 (Q) , transportadora de electrones soluble en lípidos , transporta electrones y protones mediante un ciclo redox . [10] Esta pequeña molécula de benzoquinona es muy hidrofóbica , por lo que se difunde libremente dentro de la membrana. Cuando Q acepta dos electrones y dos protones, se reduce a la forma de ubiquinol (QH 2 ); cuando QH 2 libera dos electrones y dos protones, se oxida nuevamente a la forma de ubiquinona (Q). Como resultado, si dos enzimas se disponen de manera que Q se reduzca en un lado de la membrana y QH 2 se oxide en el otro, la ubiquinona acoplará estas reacciones y transportará protones a través de la membrana. [11] Algunas cadenas de transporte de electrones bacterianas utilizan diferentes quinonas, como la menaquinona , además de la ubiquinona. [12]
Dentro de las proteínas, los electrones se transfieren entre cofactores de flavina , [5] [13] grupos de hierro-azufre y citocromos. Hay varios tipos de grupos de hierro y azufre. El tipo más simple que se encuentra en la cadena de transferencia de electrones consta de dos átomos de hierro unidos por dos átomos de azufre inorgánico ; estos se denominan grupos [2Fe-2S]. El segundo tipo, llamado [4Fe–4S], contiene un cubo de cuatro átomos de hierro y cuatro átomos de azufre. Cada átomo de hierro en estos grupos está coordinado por un aminoácido adicional , generalmente por el átomo de azufre de la cisteína . Los cofactores de iones metálicos sufren reacciones redox sin unirse ni liberar protones, por lo que en la cadena de transporte de electrones sirven únicamente para transportar electrones a través de proteínas. Los electrones se mueven distancias bastante largas a través de las proteínas saltando a lo largo de cadenas de estos cofactores. [14] Esto ocurre mediante túneles cuánticos , que son rápidos en distancias de menos de 1,4 × 10 −9 m. [15]
Muchos procesos bioquímicos catabólicos , como la glucólisis , el ciclo del ácido cítrico y la beta oxidación , producen la coenzima reducida NADH . Esta coenzima contiene electrones que tienen un alto potencial de transferencia ; en otras palabras, liberarán una gran cantidad de energía tras la oxidación. Sin embargo, la célula no libera esta energía de una vez, ya que sería una reacción incontrolable. En cambio, los electrones se eliminan del NADH y se pasan al oxígeno a través de una serie de enzimas, cada una de las cuales libera una pequeña cantidad de energía. Este conjunto de enzimas, formado por los complejos I al IV, se denomina cadena de transporte de electrones y se encuentra en la membrana interna de la mitocondria. El succinato también se oxida mediante la cadena de transporte de electrones, pero ingresa a la vía en un punto diferente.
En los eucariotas , las enzimas de este sistema de transporte de electrones utilizan la energía liberada del O 2 por el NADH para bombear protones a través de la membrana interna de la mitocondria. Esto hace que los protones se acumulen en el espacio intermembrana y genera un gradiente electroquímico a través de la membrana. La energía almacenada en este potencial es luego utilizada por la ATP sintasa para producir ATP. La fosforilación oxidativa en la mitocondria eucariota es el ejemplo mejor comprendido de este proceso. La mitocondria está presente en casi todos los eucariotas, con la excepción de los protozoos anaeróbicos como Trichomonas vaginalis que, en cambio, reducen los protones a hidrógeno en una mitocondria remanente llamada hidrogenosoma . [dieciséis]
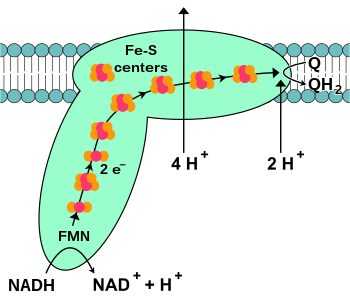
La NADH-coenzima Q oxidorreductasa , también conocida como NADH deshidrogenasa o complejo I , es la primera proteína de la cadena de transporte de electrones. [18] El complejo I es una enzima gigante ; el complejo I de los mamíferos tiene 46 subunidades y una masa molecular de aproximadamente 1000 kilodaltons (kDa). [19] La estructura se conoce en detalle sólo de una bacteria; [20] [21] en la mayoría de los organismos el complejo se asemeja a una bota con una gran "bola" que sobresale de la membrana hacia la mitocondria. [22] [23] Los genes que codifican las proteínas individuales están contenidos tanto en el núcleo celular como en el genoma mitocondrial , como es el caso de muchas enzimas presentes en la mitocondria.
La reacción catalizada por esta enzima es la oxidación de dos electrones del NADH por la coenzima Q10 o ubiquinona (representada como Q en la siguiente ecuación), una quinona soluble en lípidos que se encuentra en la membrana de la mitocondria:
El comienzo de la reacción, y de hecho de toda la cadena electrónica, es la unión de una molécula de NADH al complejo I y la donación de dos electrones. Los electrones ingresan al complejo I a través de un grupo protésico unido al complejo, el mononucleótido de flavina (FMN). La adición de electrones al FMN lo convierte a su forma reducida, FMNH 2 . Luego, los electrones se transfieren a través de una serie de grupos de hierro y azufre: el segundo tipo de grupo protésico presente en el complejo. [20] Hay grupos de hierro y azufre [2Fe–2S] y [4Fe–4S] en el complejo I.
A medida que los electrones atraviesan este complejo, se bombean cuatro protones desde la matriz al espacio intermembrana. No está claro exactamente cómo ocurre esto, pero parece implicar cambios conformacionales en el complejo I que hacen que la proteína se una a protones en el lado N de la membrana y los libere en el lado P de la membrana. [24] Finalmente, los electrones se transfieren de la cadena de grupos de hierro y azufre a una molécula de ubiquinona en la membrana. [18] La reducción de la ubiquinona también contribuye a la generación de un gradiente de protones, ya que se toman dos protones de la matriz a medida que se reduce a ubiquinol (QH 2 ).

La succinato-Q oxidorreductasa , también conocida como complejo II o succinato deshidrogenasa , es un segundo punto de entrada a la cadena de transporte de electrones. [25] Es inusual porque es la única enzima que forma parte tanto del ciclo del ácido cítrico como de la cadena de transporte de electrones. El complejo II consta de cuatro subunidades proteicas y contiene un cofactor de flavina adenina dinucleótido (FAD) unido, grupos hierro-azufre y un grupo hemo que no participa en la transferencia de electrones a la coenzima Q, pero se cree que es importante para disminuir la producción de reactivos. especies de oxígeno. [26] [27] Oxida el succinato a fumarato y reduce la ubiquinona. Como esta reacción libera menos energía que la oxidación del NADH, el complejo II no transporta protones a través de la membrana y no contribuye al gradiente de protones.
En algunos eucariotas, como el gusano parásito Ascaris suum , una enzima similar al complejo II, la fumarato reductasa (menaquinol:fumarato oxidorreductasa o QFR), opera a la inversa para oxidar el ubiquinol y reducir el fumarato. Esto permite al gusano sobrevivir en el ambiente anaeróbico del intestino grueso , llevando a cabo una fosforilación oxidativa anaeróbica con fumarato como aceptor de electrones. [28] Otra función no convencional del complejo II se observa en el parásito de la malaria Plasmodium falciparum . Aquí, la acción inversa del complejo II como oxidasa es importante en la regeneración del ubiquinol, que el parásito utiliza en una forma inusual de biosíntesis de pirimidina . [29]
La flavoproteína-ubiquinona oxidorreductasa de transferencia de electrones (ETF-Q oxidorreductasa), también conocida como flavoproteína deshidrogenasa de transferencia de electrones , es un tercer punto de entrada a la cadena de transporte de electrones. Es una enzima que acepta electrones de la flavoproteína que transfiere electrones en la matriz mitocondrial y utiliza estos electrones para reducir la ubiquinona. [30] Esta enzima contiene una flavina y un grupo [4Fe-4S], pero, a diferencia de los otros complejos respiratorios, se adhiere a la superficie de la membrana y no cruza la bicapa lipídica. [31]
En los mamíferos, esta vía metabólica es importante en la beta oxidación de los ácidos grasos y el catabolismo de los aminoácidos y la colina , ya que acepta electrones de múltiples acetil-CoA deshidrogenasas. [32] [33] En las plantas, la oxidorreductasa ETF-Q también es importante en las respuestas metabólicas que permiten la supervivencia en períodos prolongados de oscuridad. [34]

La Q-citocromo c oxidorreductasa también se conoce como citocromo c reductasa , complejo citocromo bc 1 o simplemente complejo III . [35] [36] En los mamíferos, esta enzima es un dímero , y cada complejo de subunidades contiene 11 subunidades de proteínas, un grupo de hierro y azufre [2Fe-2S] y tres citocromos : un citocromo c 1 y dos citocromos b . [37] Un citocromo es un tipo de proteína transmisora de electrones que contiene al menos un grupo hemo . Los átomos de hierro dentro de los grupos hemo del complejo III alternan entre un estado ferroso reducido (+2) y férrico oxidado (+3) a medida que los electrones se transfieren a través de la proteína.
La reacción catalizada por el complejo III es la oxidación de una molécula de ubiquinol y la reducción de dos moléculas de citocromo c , una proteína hemo ligeramente asociada con la mitocondria. A diferencia de la coenzima Q, que transporta dos electrones, el citocromo c transporta solo un electrón.
Como sólo uno de los electrones puede transferirse desde el donante de QH 2 a un aceptor de citocromo c a la vez, el mecanismo de reacción del complejo III es más elaborado que el de los otros complejos respiratorios y ocurre en dos pasos llamados ciclo Q. [38] En el primer paso, la enzima se une a tres sustratos, primero, QH 2 , que luego se oxida y pasa un electrón al segundo sustrato, el citocromo c. Los dos protones liberados del QH 2 pasan al espacio intermembrana. El tercer sustrato es Q, que acepta el segundo electrón del QH 2 y se reduce a Q.− , que es el radical libre ubisemiquinona . Los dos primeros sustratos se liberan, pero este intermediario de ubisemiquinona permanece unido. En el segundo paso, se une una segunda molécula de QH 2 y nuevamente pasa su primer electrón a un aceptor del citocromo c. El segundo electrón pasa a la ubisemiquinona unida, reduciéndola a QH 2 a medida que gana dos protones de la matriz mitocondrial. Este QH 2 luego se libera de la enzima. [39]
A medida que la coenzima Q se reduce a ubiquinol en el lado interno de la membrana y se oxida a ubiquinona en el otro, se produce una transferencia neta de protones a través de la membrana, lo que aumenta el gradiente de protones. [5] El mecanismo bastante complejo de dos pasos mediante el cual esto ocurre es importante, ya que aumenta la eficiencia de la transferencia de protones. Si, en lugar del ciclo Q, se usara una molécula de QH 2 para reducir directamente dos moléculas de citocromo c, la eficiencia se reduciría a la mitad, con sólo un protón transferido por cada citocromo c reducido. [5]

La citocromo c oxidasa , también conocida como complejo IV , es el complejo proteico final de la cadena de transporte de electrones. [40] La enzima de los mamíferos tiene una estructura extremadamente complicada y contiene 13 subunidades, dos grupos hemo, así como múltiples cofactores de iones metálicos: en total, tres átomos de cobre , uno de magnesio y uno de zinc . [41]
Esta enzima media la reacción final en la cadena de transporte de electrones y transfiere electrones a oxígeno e hidrógeno (protones), mientras bombea protones a través de la membrana. [42] El oxígeno aceptor de electrones final se reduce a agua en este paso. Tanto el bombeo directo de protones como el consumo de protones de la matriz en la reducción de oxígeno contribuyen al gradiente de protones. La reacción catalizada es la oxidación del citocromo c y la reducción de oxígeno:
Muchos organismos eucariotas tienen cadenas de transporte de electrones que difieren de las muy estudiadas enzimas de los mamíferos descritas anteriormente. Por ejemplo, las plantas tienen NADH oxidasas alternativas, que oxidan el NADH en el citosol en lugar de en la matriz mitocondrial, y pasan estos electrones al depósito de ubiquinona. [43] Estas enzimas no transportan protones y, por lo tanto, reducen la ubiquinona sin alterar el gradiente electroquímico a través de la membrana interna. [44]
Otro ejemplo de cadena de transporte de electrones divergente es la oxidasa alternativa , que se encuentra en las plantas , así como en algunos hongos , protistas y posiblemente en algunos animales. [45] [46] Esta enzima transfiere electrones directamente del ubiquinol al oxígeno. [47]
Las vías de transporte de electrones producidas por estas NADH alternativas y ubiquinona oxidasas tienen rendimientos de ATP más bajos que la vía completa. Las ventajas que produce una vía más corta no están del todo claras. Sin embargo, la oxidasa alternativa se produce en respuesta a estreses como el frío, las especies reactivas de oxígeno y la infección por patógenos, así como otros factores que inhiben la cadena de transporte de electrones completa. [48] [49] Por lo tanto, las vías alternativas podrían mejorar la resistencia de un organismo a las lesiones, al reducir el estrés oxidativo . [50]
El modelo original de cómo se organizan los complejos de la cadena respiratoria era que se difunden libre e independientemente en la membrana mitocondrial. [51] Sin embargo, datos recientes sugieren que los complejos podrían formar estructuras de orden superior llamadas supercomplejos o " respirasomas ". [52] En este modelo, los diversos complejos existen como conjuntos organizados de enzimas que interactúan. [53] Estas asociaciones podrían permitir la canalización de sustratos entre los diversos complejos enzimáticos, aumentando la velocidad y la eficiencia de la transferencia de electrones. [54] Dentro de tales supercomplejos de mamíferos, algunos componentes estarían presentes en cantidades mayores que otros, y algunos datos sugieren una relación entre los complejos I/II/III/IV y la ATP sintasa de aproximadamente 1:1:3:7:4. [55] Sin embargo, el debate sobre esta hipótesis supercompleja no está completamente resuelto, ya que algunos datos no parecen encajar con este modelo. [19] [56]
En contraste con la similitud general en estructura y función de las cadenas de transporte de electrones en los eucariotas, las bacterias y arqueas poseen una gran variedad de enzimas de transferencia de electrones. Estos utilizan un conjunto igualmente amplio de productos químicos como sustratos. [57] Al igual que los eucariotas, el transporte de electrones procarióticos utiliza la energía liberada por la oxidación de un sustrato para bombear iones a través de una membrana y generar un gradiente electroquímico. En las bacterias, la fosforilación oxidativa en Escherichia coli se comprende con mayor detalle, mientras que los sistemas de arqueas se conocen poco en la actualidad. [58]
La principal diferencia entre la fosforilación oxidativa eucariota y procariótica es que las bacterias y arqueas utilizan muchas sustancias diferentes para donar o aceptar electrones. Esto permite que los procariotas crezcan en una amplia variedad de condiciones ambientales. [59] En E. coli , por ejemplo, la fosforilación oxidativa puede ser impulsada por una gran cantidad de pares de agentes reductores y agentes oxidantes, que se enumeran a continuación. El potencial del punto medio de una sustancia química mide cuánta energía se libera cuando se oxida o se reduce; los agentes reductores tienen potenciales negativos y los agentes oxidantes tienen potenciales positivos.
Como se muestra arriba, E. coli puede crecer con agentes reductores como formiato, hidrógeno o lactato como donadores de electrones, y nitrato, DMSO u oxígeno como aceptores. [59] Cuanto mayor es la diferencia en el potencial del punto medio entre un agente oxidante y un agente reductor, más energía se libera cuando reaccionan. De estos compuestos, el par succinato/fumarato es inusual, ya que su potencial de punto medio es cercano a cero. Por lo tanto, el succinato se puede oxidar a fumarato si se dispone de un agente oxidante fuerte, como oxígeno, o el fumarato se puede reducir a succinato utilizando un agente reductor fuerte, como el formiato. Estas reacciones alternativas son catalizadas por la succinato deshidrogenasa y la fumarato reductasa , respectivamente. [61]
Algunos procariotas utilizan pares redox que tienen sólo una pequeña diferencia en el potencial del punto medio. Por ejemplo, las bacterias nitrificantes como Nitrobacter oxidan el nitrito a nitrato, donando electrones al oxígeno. La pequeña cantidad de energía liberada en esta reacción es suficiente para bombear protones y generar ATP, pero no suficiente para producir NADH o NADPH directamente para su uso en anabolismo . [62] Este problema se resuelve utilizando una nitrito oxidorreductasa para producir suficiente fuerza motriz de protones para hacer funcionar parte de la cadena de transporte de electrones en reversa, lo que hace que el complejo I genere NADH. [63] [64]
Los procariotas controlan el uso de estos donantes y aceptores de electrones variando las enzimas que se producen, en respuesta a las condiciones ambientales. [65] Esta flexibilidad es posible porque diferentes oxidasas y reductasas utilizan el mismo grupo de ubiquinona. Esto permite que muchas combinaciones de enzimas funcionen juntas, unidas por el intermedio común ubiquinol. [60] Por lo tanto, estas cadenas respiratorias tienen un diseño modular , con conjuntos de sistemas enzimáticos fácilmente intercambiables.
Además de esta diversidad metabólica, los procariotas también poseen una variedad de isoenzimas : diferentes enzimas que catalizan la misma reacción. Por ejemplo, en E. coli , existen dos tipos diferentes de ubiquinol oxidasa que utilizan oxígeno como aceptor de electrones. En condiciones altamente aeróbicas, la célula utiliza una oxidasa con baja afinidad por el oxígeno que puede transportar dos protones por electrón. Sin embargo, si los niveles de oxígeno caen, cambian a una oxidasa que transfiere sólo un protón por electrón, pero que tiene una alta afinidad por el oxígeno. [66]
La ATP sintasa, también llamada complejo V , es la enzima final en la vía de la fosforilación oxidativa. Esta enzima se encuentra en todas las formas de vida y funciona de la misma manera tanto en procariotas como en eucariotas. [67] La enzima utiliza la energía almacenada en un gradiente de protones a través de una membrana para impulsar la síntesis de ATP a partir de ADP y fosfato (P i ). Las estimaciones del número de protones necesarios para sintetizar un ATP han oscilado entre tres y cuatro, [68] [69] y algunos sugieren que las células pueden variar esta proporción para adaptarse a diferentes condiciones. [70]
Esta reacción de fosforilación es un equilibrio que puede modificarse alterando la fuerza motriz del protón. En ausencia de una fuerza motriz de protones, la reacción de la ATP sintasa se desarrollará de derecha a izquierda, hidrolizando el ATP y bombeando protones fuera de la matriz a través de la membrana. Sin embargo, cuando la fuerza motriz del protón es alta, la reacción se ve obligada a realizarse en la dirección opuesta; procede de izquierda a derecha, lo que permite que los protones fluyan a favor de su gradiente de concentración y conviertan el ADP en ATP. [67] De hecho, en las H+-ATPasas de tipo vacuolar estrechamente relacionadas , la reacción de hidrólisis se utiliza para acidificar los compartimentos celulares, bombeando protones e hidrolizando ATP. [71]
La ATP sintasa es un complejo proteico masivo con forma de hongo. El complejo enzimático de los mamíferos contiene 16 subunidades y tiene una masa de aproximadamente 600 kilodaltons . [72] La porción incrustada dentro de la membrana se llama FO y contiene un anillo de subunidades c y el canal de protones. El tallo y la cabeza esférica se denomina F 1 y es el lugar de síntesis de ATP. El complejo en forma de bola al final de la porción F 1 contiene seis proteínas de dos tipos diferentes (tres subunidades α y tres subunidades β), mientras que el "tallo" consta de una proteína: la subunidad γ, con la punta del tallo extendiéndose hacia la bola de subunidades α y β. [73] Tanto las subunidades α como β se unen a nucleótidos, pero sólo las subunidades β catalizan la reacción de síntesis de ATP. A lo largo del costado de la porción F 1 y de regreso a la membrana hay una subunidad larga en forma de varilla que ancla las subunidades α y β en la base de la enzima.
A medida que los protones cruzan la membrana a través del canal en la base de la ATP sintasa, el motor impulsado por protones FO gira . [74] La rotación podría ser causada por cambios en la ionización de los aminoácidos en el anillo de las subunidades c que causan interacciones electrostáticas que impulsan el anillo de las subunidades c más allá del canal de protones. [75] Este anillo giratorio a su vez impulsa la rotación del eje central (el tallo de la subunidad γ) dentro de las subunidades α y β. El brazo lateral, que actúa como estator , impide que las subunidades α y β giren entre sí . Este movimiento de la punta de la subunidad γ dentro de la bola de las subunidades α y β proporciona energía para que los sitios activos de las subunidades β experimenten un ciclo de movimientos que produce y luego libera ATP. [76]

Esta reacción de síntesis de ATP se denomina mecanismo de cambio de unión e involucra el sitio activo de una subunidad β que circula entre tres estados. [77] En el estado "abierto", el ADP y el fosfato ingresan al sitio activo (que se muestra en marrón en el diagrama). Luego, la proteína se cierra alrededor de las moléculas y las une libremente: el estado "suelto" (que se muestra en rojo). Luego, la enzima cambia de forma nuevamente y fuerza a estas moléculas a unirse, con el sitio activo en el estado "estrecho" resultante (que se muestra en rosa) uniendo la molécula de ATP recién producida con muy alta afinidad . Finalmente, el sitio activo regresa al estado abierto, liberando ATP y uniendo más ADP y fosfato, listo para el siguiente ciclo.
En algunas bacterias y arqueas, la síntesis de ATP está impulsada por el movimiento de iones de sodio a través de la membrana celular, en lugar del movimiento de protones. [78] [79] Archaea como Methanococcus también contiene la A 1 A o sintasa, una forma de enzima que contiene proteínas adicionales con poca similitud en secuencia con otras subunidades de ATP sintasa bacterianas y eucariotas. Es posible que, en algunas especies, la forma A 1 A o de la enzima sea una ATP sintasa especializada impulsada por sodio, [80] pero esto puede no ser cierto en todos los casos. [79]
El transporte de electrones desde el par redox NAD + /NADH hasta el par redox final 1/2 O 2 / H 2 O se puede resumir como
1/2 O 2 + NADH + H + → H 2 O + NAD +
La diferencia de potencial entre estos dos pares redox es de 1,14 voltios, lo que equivale a -52 kcal/mol o -2600 kJ por 6 moles de O 2 .
Cuando se oxida un NADH a través de la cadena de transferencia de electrones, se producen tres ATP, lo que equivale a 7,3 kcal/mol x 3 = 21,9 kcal/mol.
La conservación de la energía se puede calcular mediante la siguiente fórmula.
Eficiencia = (21,9 x 100%) / 52 = 42%
Entonces podemos concluir que cuando se oxida NADH, aproximadamente el 42% de la energía se conserva en forma de tres ATP y la energía restante (58%) se pierde en forma de calor (a menos que se subestime la energía química del ATP en condiciones fisiológicas).
El oxígeno molecular es un buen aceptor terminal de electrones porque es un fuerte agente oxidante. La reducción de oxígeno implica intermediarios potencialmente dañinos. [81] Aunque la transferencia de cuatro electrones y cuatro protones reduce el oxígeno al agua, que es inofensivo, la transferencia de uno o dos electrones produce aniones superóxido o peróxido , que son peligrosamente reactivos.
Estas especies reactivas de oxígeno y sus productos de reacción, como el radical hidroxilo , son muy perjudiciales para las células, ya que oxidan proteínas y provocan mutaciones en el ADN . Este daño celular puede contribuir a la enfermedad y se propone como una de las causas del envejecimiento . [82] [83]
El complejo de citocromo c oxidasa es muy eficaz para reducir el oxígeno a agua y libera muy pocos intermediarios parcialmente reducidos; sin embargo, la cadena de transporte de electrones produce pequeñas cantidades de anión superóxido y peróxido. [84] Particularmente importante es la reducción de la coenzima Q en el complejo III, ya que se forma un radical libre de ubisemiquinona altamente reactivo como intermediario en el ciclo Q. Esta especie inestable puede provocar una "fuga" de electrones cuando los electrones se transfieren directamente al oxígeno, formando superóxido. [85] Como la producción de especies reactivas de oxígeno por parte de estos complejos de bombeo de protones es mayor en potenciales de membrana altos, se ha propuesto que las mitocondrias regulen su actividad para mantener el potencial de membrana dentro de un rango estrecho que equilibre la producción de ATP con la generación de oxidantes. [86] Por ejemplo, los oxidantes pueden activar proteínas desacopladoras que reducen el potencial de membrana. [87]
Para contrarrestar estas especies reactivas de oxígeno, las células contienen numerosos sistemas antioxidantes , incluidas vitaminas antioxidantes como la vitamina C y la vitamina E , y enzimas antioxidantes como la superóxido dismutasa , la catalasa y las peroxidasas , [81] que desintoxican las especies reactivas, limitando el daño a las células. celúla.
Como el oxígeno es fundamental para la fosforilación oxidativa, una escasez en el nivel de O 2 probablemente altere las tasas de producción de ATP. Sin embargo, la fuerza motriz de los protones y la producción de ATP pueden mantenerse mediante acidosis intracelular. [88] Los protones citosólicos que se han acumulado con la hidrólisis del ATP y la acidosis láctica pueden difundirse libremente a través de la membrana externa mitocondrial y acidificar el espacio entre membranas, contribuyendo así directamente a la fuerza motriz de los protones y a la producción de ATP.
Existen varios fármacos y toxinas bien conocidos que inhiben la fosforilación oxidativa. Aunque cualquiera de estas toxinas inhibe sólo una enzima en la cadena de transporte de electrones, la inhibición de cualquier paso de este proceso detendrá el resto del proceso. Por ejemplo, si la oligomicina inhibe la ATP sintasa, los protones no pueden regresar a la mitocondria. [89] Como resultado, las bombas de protones no pueden funcionar, ya que el gradiente se vuelve demasiado fuerte para que puedan superarlo. Entonces el NADH ya no se oxida y el ciclo del ácido cítrico deja de funcionar porque la concentración de NAD + cae por debajo de la concentración que estas enzimas pueden utilizar.
Muchos inhibidores específicos de sitio de la cadena de transporte de electrones han contribuido al conocimiento actual de la respiración mitocondrial. La síntesis de ATP también depende de la cadena de transporte de electrones, por lo que todos los inhibidores específicos de sitio también inhiben la formación de ATP. El veneno para peces rotenona , el barbitúrico amytal y el antibiótico piericidina A inhiben el NADH y la coenzima Q. [90]
El monóxido de carbono, el cianuro, el sulfuro de hidrógeno y la azida inhiben eficazmente la citocromo oxidasa. El monóxido de carbono reacciona con la forma reducida del citocromo, mientras que el cianuro y la azida reaccionan con la forma oxidada. Un antibiótico, la antimicina A , y la antilewisita británica , un antídoto utilizado contra las armas químicas, son los dos inhibidores importantes del sitio entre el citocromo B y el C1. [90]
No todos los inhibidores de la fosforilación oxidativa son toxinas. En el tejido adiposo marrón , los canales de protones regulados llamados proteínas desacoplantes pueden desacoplar la respiración de la síntesis de ATP. [95] Esta respiración rápida produce calor y es particularmente importante como forma de mantener la temperatura corporal de los animales en hibernación , aunque estas proteínas también pueden tener una función más general en las respuestas de las células al estrés. [96]
El campo de la fosforilación oxidativa comenzó con el informe de 1906 de Arthur Harden sobre el papel vital del fosfato en la fermentación celular , pero inicialmente sólo se sabía que estaban involucrados los fosfatos de azúcar . [97] Sin embargo, a principios de la década de 1940, Herman Kalckar estableció firmemente el vínculo entre la oxidación de azúcares y la generación de ATP , [98] confirmando el papel central del ATP en la transferencia de energía que había sido propuesto por Fritz Albert Lipmann en 1941. [99] Posteriormente, en 1949, Morris Friedkin y Albert L. Lehninger demostraron que la coenzima NADH vinculaba vías metabólicas como el ciclo del ácido cítrico y la síntesis de ATP. [100] El término fosforilación oxidativa fue acuñado por Volodymyr Belitser en 1939. [101] [102]
Durante otros veinte años, el mecanismo por el cual se genera ATP siguió siendo un misterio, y los científicos buscaban un elusivo "intermedio de alta energía" que vinculara las reacciones de oxidación y fosforilación. [103] Este enigma fue resuelto por Peter D. Mitchell con la publicación de la teoría quimiosmótica en 1961. [104] Al principio, esta propuesta fue muy controvertida, pero fue aceptada lentamente y Mitchell recibió el premio Nobel en 1978. [ 105] [106] La investigación posterior se concentró en purificar y caracterizar las enzimas involucradas, con contribuciones importantes de David E. Green sobre los complejos de la cadena de transporte de electrones, así como de Efraim Racker sobre la ATP sintasa. [107] Paul D. Boyer proporcionó un paso crítico hacia la solución del mecanismo de la ATP sintasa , con su desarrollo en 1973 del mecanismo de "cambio de unión", seguido de su propuesta radical de catálisis rotacional en 1982. [77] [ 108] Trabajos más recientes han incluido estudios estructurales sobre las enzimas implicadas en la fosforilación oxidativa realizados por John E. Walker , y Walker y Boyer recibieron el Premio Nobel en 1997. [109]