El poliovirus , agente causante de la polio (también conocida como poliomielitis), es un serotipo de la especie Enterovirus C , de la familia Picornaviridae . [1] Existen tres serotipos de poliovirus, numerados 1, 2 y 3.
El virus de la poliomielitis está compuesto por un genoma de ARN y una cápside proteica . El genoma es un genoma de ARN monocatenario de sentido positivo (+ssRNA) que tiene aproximadamente 7500 nucleótidos de longitud. [2] La partícula viral tiene aproximadamente 30 nm de diámetro con simetría icosaédrica . Debido a su genoma corto y su composición simple (solo una hebra de ARN y una capa proteica icosaédrica sin envoltura que la encapsula ), el virus de la poliomielitis es ampliamente considerado como el virus significativo más simple. [3]
El poliovirus es uno de los virus mejor caracterizados y se ha convertido en un sistema modelo útil para comprender la biología de los virus de ARN .
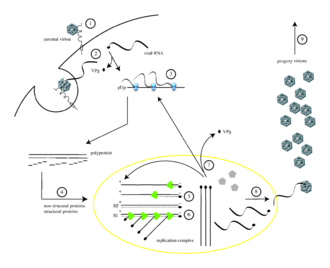
El poliovirus infecta las células humanas al unirse a un receptor similar a la inmunoglobulina , CD155 (también conocido como receptor del poliovirus o PVR) [5] [6] en la superficie celular. [7] La interacción del poliovirus y el CD155 facilita un cambio conformacional irreversible de la partícula viral necesaria para la entrada viral. [8] [9] Después de la unión a la membrana de la célula huésped , se pensaba que la entrada del ácido nucleico viral se producía de una de dos maneras: a través de la formación de un poro en la membrana plasmática a través del cual el ARN se "inyecta" en el citoplasma de la célula huésped , o mediante la captación del virus por endocitosis mediada por receptores . [10] La evidencia experimental reciente apoya la última hipótesis y sugiere que el poliovirus se une al CD155 y es captado por endocitosis. Inmediatamente después de la internalización de la partícula, se libera el ARN viral. [11]
El poliovirus es un virus de ARN de cadena positiva . Por lo tanto, el genoma encerrado dentro de la partícula viral puede usarse como ARN mensajero y ser traducido inmediatamente por la célula huésped. Al entrar, el virus secuestra la maquinaria de traducción de la célula, lo que provoca la inhibición de la síntesis de proteínas celulares a favor de la producción de proteínas específicas del virus. [12] A diferencia de los ARNm de la célula huésped, el extremo 5' del ARN del poliovirus es extremadamente largo (más de 700 nucleótidos) y altamente estructurado. Esta región del genoma viral se denomina sitio de entrada al ribosoma interno (IRES). Esta región consta de muchas estructuras secundarias y 3 o 4 dominios. El dominio 3 es un elemento de ARN autoplegable que contiene motivos estructurales conservados en varios bucles de tallo estables unidos por dos uniones de cuatro vías. Como IRES consta de muchos dominios, estos dominios a su vez constan de muchos bucles que contribuyen a la traducción modificada sin una tapa final 5' secuestrando ribosomas. El bucle de interacción del dominio 3 se conoce como tetraloop GNRA. Los residuos de adenosinas A180 y A181 en el tetraloop GUAA forman enlaces de hidrógeno a través de interacciones de apareamiento de bases no canónicas con los pares de bases de los receptores C230/G242 y G231/C241, respectivamente. [13] Las mutaciones genéticas en esta región impiden la producción de proteínas virales. [14] [15] [16] El primer IRES que se descubrió se encontró en el ARN del poliovirus. [17]
El ARNm del poliovirus se traduce como un polipéptido largo . Luego, este polipéptido es autoescindido por proteasas internas en aproximadamente 10 proteínas virales individuales. No todas las escisiones se producen con la misma eficiencia. Por lo tanto, las cantidades de proteínas producidas por la escisión del polipéptido varían: por ejemplo, se producen cantidades más pequeñas de pol 3D que de proteínas de la cápside, VP1–4. [18] : 165 [19] Estas proteínas virales individuales son: [3] [20]

Después de la traducción, transcripción y replicación del genoma, que implican un solo proceso, se realiza la síntesis de (+) ARN). Para que el (+) ARN infectante se replique, se deben transcribir múltiples copias de (−) ARN y luego usarlas como plantillas para la síntesis de (+) ARN. Los intermediarios replicativos (RI), que son una asociación de moléculas de ARN que consisten en un ARN plantilla y varios ARN en crecimiento de longitud variable, se ven tanto en los complejos de replicación para (−) ARN como para (+) ARN. Para la síntesis de cada ARN de cadena negativa y cadena positiva, la proteína VPg en el poliovirus funciona como un cebador. La ARN polimerasa dependiente de ARN del poliovirus agrega dos nucleótidos de uracilo (UU) a la proteína VPg utilizando la cola de poli(A) en el extremo 3' del genoma +ssRNA como un patrón para la síntesis del ARN antigénico de cadena negativa. Para iniciar esta síntesis de −ssRNA, se necesita el hidroxilo de tirosina de VPg. Pero para el inicio de la síntesis de ARN de cadena positiva, se necesita la uridilación de VPg dependiente de CRE. Esto significa que VPg se utiliza una vez más como cebador, pero esta vez agrega los dos trifosfatos de uridina utilizando un factor de replicación que actúa en cis (CRE) como plantilla. [22] [23]
El CRE del poliovirus se identifica como un tallo de pares de bases no logrados y un bucle final que consta de 61 nt. El CRE se encuentra en los enterovirus. Es un elemento estructural secundario de ARN altamente conservado y se encuentra en la región codificante de poliproteínas del genoma. El complejo se puede translocar a la región 5' del genoma que no tiene actividad codificante, al menos a 3,7 kb de distancia de la ubicación inicial. Este proceso puede ocurrir sin influir negativamente en la actividad. Las copias de CRE no influyen negativamente en la replicación. El proceso de uridilación de VPg que tiene lugar en CRE necesita la presencia de 3CD pro , que es una proteína de unión al ARN. Se une al CRE de forma directa y específica. Debido a su presencia, VPg puede unirse al CRE de forma adecuada y la producción primaria se lleva a cabo sin problemas. [24]
Algunas de las moléculas de ARN (+) se utilizan como plantillas para la posterior síntesis de ARN (−), algunas funcionan como ARNm y algunas están destinadas a ser los genomas de los viriones descendientes. [18]
En el ensamblaje de nuevas partículas virales (es decir, el empaquetamiento del genoma de la progenie en una procápside que puede sobrevivir fuera de la célula huésped), incluyendo, respectivamente: [18] : 161, 165
El poliovirus completamente ensamblado abandona los confines de su célula huésped por lisis [25] de 4 a 6 horas después del inicio de la infección en células de mamíferos cultivadas. [26] El mecanismo de liberación viral de la célula no está claro, [2] pero cada célula moribunda puede liberar hasta 10.000 viriones de polio . [26]
Drake demostró que el virus de la poliomielitis es capaz de experimentar una reactivación múltiple. [27] Es decir, cuando los virus de la poliomielitis se irradiaban con luz ultravioleta y se les permitía sufrir múltiples infecciones de células hospedadoras, se podía formar una progenie viable incluso con dosis de luz ultravioleta que inactivaban el virus en infecciones únicas. El virus de la poliomielitis puede experimentar una recombinación genética cuando al menos dos genomas virales están presentes en la misma célula hospedadora. Kirkegaard y Baltimore [28] presentaron evidencia de que la ARN polimerasa dependiente de ARN (RdRP) cataliza la recombinación mediante un mecanismo de elección de copia en el que la RdRP cambia entre plantillas de (+)ssRNA durante la síntesis de la cadena negativa. La recombinación en los virus de ARN parece ser un mecanismo adaptativo para reparar el daño del genoma. [29] [30]
El poliovirus es estructuralmente similar a otros enterovirus humanos ( coxsackievirus , echovirus y rinovirus ), que también utilizan moléculas similares a las inmunoglobulinas para reconocer y entrar en las células huésped. [6] El análisis filogenético de las secuencias de ARN y proteínas del poliovirus sugiere que puede haber evolucionado a partir de un ancestro del virus Coxsackie A del grupo C a través de una mutación en la cápside. [31] La especiación distinta del poliovirus probablemente ocurrió como resultado de un cambio en la especificidad del receptor celular de la molécula de adhesión intercelular-1 (ICAM-1) (utilizada por los virus Coxsackie A del grupo C) a CD155 , lo que lleva a un cambio en la patogenicidad y permite que el virus infecte el tejido nervioso.
La tasa de mutación en el virus es relativamente alta incluso para un virus ARN, con una tasa de sustitución sinónima de 1,0 × 10 −2 sustituciones/sitio/año y una tasa de sustitución no sinónima de 3,0 × 10 −4 sustituciones/sitio/año. [32] La distribución de bases dentro del genoma no es aleatoria; la adenosina es menos común de lo esperado en el extremo 5' y más en el extremo 3'. [33] El uso de codones no es aleatorio; los codones que terminan en adenosina son los preferidos y los que terminan en citosina o guanina se evitan. El uso de codones difiere entre los tres genotipos y parece estar impulsado por la mutación en lugar de la selección. [34]
Los tres serotipos del poliovirus, PV-1, PV-2 y PV-3, tienen cada uno una proteína de la cápside ligeramente diferente. Las proteínas de la cápside definen la especificidad del receptor celular y la antigenicidad del virus. El PV-1 es la forma más común encontrada en la naturaleza, pero las tres formas son extremadamente infecciosas . [35] A marzo de 2020, el PV-1 salvaje está altamente localizado en regiones de Pakistán y Afganistán. La certificación de la erradicación de la transmisión autóctona del PV-2 salvaje ocurrió en septiembre de 2015, después de haber sido detectado por última vez en 1999, [36] y en octubre de 2019 para el PV-3 salvaje después de haber sido detectado por última vez en 2012. [37] Sin embargo, el poliovirus circulante derivado de la vacuna (poliovirus variante, cVDPV) de los tres serotipos continúa circulando y causando parálisis, habiéndose detectado en 32 países en 2023. [38]
Se utilizan cepas específicas de cada serotipo para preparar vacunas contra la polio . La vacuna antipoliomielítica inactiva se prepara mediante la inactivación con formalina de tres cepas de referencia virulentas y salvajes: Mahoney o Brunenders (PV-1), MEF-1/Lansing (PV-2) y Saukett/Leon (PV-3). La vacuna antipoliomielítica oral contiene cepas vivas atenuadas (debilitadas) de los tres serotipos del virus de la polio. El paso de las cepas del virus en células epiteliales de riñón de mono introduce mutaciones en el IRES viral y dificulta (o atenúa) la capacidad del virus para infectar el tejido nervioso. [26]
Los poliovirus se clasificaban anteriormente como una especie distinta perteneciente al género Enterovirus en la familia Picornaviridae . En 2008, el poliovirus dejó de ser reconocido como una especie y los tres serotipos se asignaron a la especie enterovirus humano C (posteriormente renombrado Enterovirus C ) en el género Enterovirus en la familia Picornaviridae . La especie tipo del género Enterovirus se cambió de Poliovirus a Enterovirus (humano) C. [39]
El principal determinante de la infección de cualquier virus es su capacidad de entrar en una célula y producir partículas infecciosas adicionales. Se cree que la presencia de CD155 define los animales y tejidos que pueden ser infectados por el poliovirus. El CD155 se encuentra (fuera de los laboratorios) solo en las células de humanos, primates superiores y monos del Viejo Mundo . Sin embargo, el poliovirus es estrictamente un patógeno humano y no infecta naturalmente a ninguna otra especie (aunque los chimpancés y los monos del Viejo Mundo pueden infectarse experimentalmente). [40]
El gen CD155 parece haber sido objeto de una selección positiva . [41] La proteína tiene varios dominios, de los cuales el dominio D1 contiene el sitio de unión del virus de la polio. Dentro de este dominio, 37 aminoácidos son responsables de la unión del virus.
El poliovirus es un enterovirus . La infección se produce por vía fecal-oral , lo que significa que uno ingiere el virus y la replicación viral ocurre en el tracto gastrointestinal . [42] El virus se elimina en las heces de las personas infectadas. En el 95% de los casos solo ocurre una presencia primaria y transitoria de viremia (virus en el torrente sanguíneo), y la infección por poliovirus es asintomática . En aproximadamente el 5% de los casos, el virus se propaga y se replica en otros sitios como la grasa parda , el tejido reticuloendotelial y el músculo . La replicación viral sostenida causa viremia secundaria y conduce al desarrollo de síntomas menores como fiebre, dolor de cabeza y dolor de garganta. [43] La poliomielitis paralítica ocurre en menos del 1% de las infecciones por poliovirus. La enfermedad paralítica ocurre cuando el virus ingresa al sistema nervioso central (SNC) y se replica en las neuronas motoras dentro de la médula espinal , el tronco encefálico o la corteza motora , lo que resulta en la destrucción selectiva de las neuronas motoras que conduce a una parálisis temporal o permanente . Este es un evento muy raro en los bebés, que aún tienen anticuerpos anti-poliovirus adquiridos de sus madres. [44] En casos raros, la poliomielitis paralítica conduce a un paro respiratorio y la muerte. En los casos de enfermedad paralítica, con frecuencia se observan dolor muscular y espasmos antes del inicio de la debilidad y la parálisis. La parálisis generalmente persiste de días a semanas antes de la recuperación. [45]
En muchos aspectos, se piensa que la fase neurológica de la infección es una desviación accidental de la infección gastrointestinal normal. [10] Los mecanismos por los cuales el poliovirus entra al SNC son poco conocidos. Se han sugerido tres hipótesis no mutuamente excluyentes para explicar su entrada. Todas las teorías requieren viremia primaria. La primera hipótesis predice que los viriones pasan directamente de la sangre al sistema nervioso central al cruzar la barrera hematoencefálica independientemente de CD155. [46] Una segunda hipótesis sugiere que los viriones son transportados desde los tejidos periféricos que han sido bañados en la sangre virémica, por ejemplo el tejido muscular, a la médula espinal a través de vías nerviosas mediante transporte axonal retrógrado . [47] [48] [49] Una tercera hipótesis es que el virus es importado al SNC a través de monocitos o macrófagos infectados . [4]
La poliomielitis es una enfermedad del sistema nervioso central. Sin embargo, se cree que el CD155 está presente en la superficie de la mayoría o de todas las células humanas. Por lo tanto, la expresión del receptor no explica por qué el poliovirus infecta preferentemente ciertos tejidos. Esto sugiere que el tropismo tisular se determina después de la infección celular. Trabajos recientes han sugerido que la respuesta del interferón tipo I (específicamente la del interferón alfa y beta) es un factor importante que define qué tipos de células apoyan la replicación del poliovirus. [50] En ratones que expresan CD155 (a través de ingeniería genética) pero carecen del receptor de interferón tipo I, el poliovirus no solo se replica en un repertorio expandido de tipos de tejidos, sino que estos ratones también pueden infectarse por vía oral con el virus. [51]
El poliovirus tiene dos mecanismos clave para evadir el sistema inmunológico . En primer lugar, puede sobrevivir a las condiciones altamente ácidas del estómago, lo que permite que los virus ingeridos infecten al huésped y se propaguen por todo el cuerpo a través del sistema linfático . [3] En segundo lugar, debido a que puede replicarse muy rápidamente, el virus abruma los órganos del huésped antes de que se pueda montar una respuesta inmune. [52] Si se dan detalles en la fase de unión; los poliovirus con cañones en la superficie del virión tienen sitios de unión del virus ubicados en bolsillos en las bases de los cañones. Los cañones son demasiado estrechos para el acceso de los anticuerpos , por lo que los sitios de unión del virus están protegidos de la vigilancia inmunológica del huésped, mientras que el resto de la superficie del virión puede mutar para evitar la respuesta inmunológica del huésped. [53]
Las personas que están expuestas al poliovirus, ya sea a través de la infección o por inmunización con la vacuna antipoliomielítica , desarrollan inmunidad . En individuos inmunes, los anticuerpos contra el poliovirus están presentes en las amígdalas y el tracto gastrointestinal (específicamente anticuerpos IgA ) y son capaces de bloquear la replicación del poliovirus; los anticuerpos IgG e IgM contra el poliovirus pueden prevenir la propagación del virus a las neuronas motoras del sistema nervioso central. [26] La infección con un serotipo de poliovirus no proporciona inmunidad contra los otros serotipos; sin embargo, los segundos ataques dentro del mismo individuo son extremadamente raros. [54]


La erradicación de la polio , el objetivo del cese permanente a nivel mundial de la circulación del virus de la polio y, por lo tanto, la eliminación de la poliomielitis (polio) que causa, es el objetivo de un esfuerzo multinacional de salud pública iniciado en 1988, dirigido por la Organización Mundial de la Salud (OMS), el Fondo de las Naciones Unidas para la Infancia (UNICEF) y la Fundación Rotaria . [55] Estas organizaciones, junto con los Centros para el Control y la Prevención de Enfermedades (CDC) de los Estados Unidos y la Fundación Gates , han encabezado la campaña a través de la Iniciativa Mundial para la Erradicación de la Polio (GPEI). La erradicación exitosa de enfermedades infecciosas se ha logrado dos veces antes, con la viruela en humanos [56] y la peste bovina en rumiantes. [57]
La prevención de la propagación de la enfermedad se logra mediante la vacunación . Hay dos tipos de vacuna contra la polio : la vacuna oral contra la polio (OPV), que utiliza un poliovirus debilitado , y la vacuna inactivada contra la polio (IPV), que se inyecta. La OPV es menos costosa y más fácil de administrar, y puede propagar la inmunidad más allá de la persona vacunada, creando inmunidad de contacto . Ha sido la vacuna predominantemente utilizada. Sin embargo, en condiciones de circulación prolongada del virus de la vacuna en poblaciones subvacunadas, las mutaciones pueden reactivar el virus para producir una cepa inductora de polio, mientras que la OPV también puede, en raras circunstancias, inducir polio o infección asintomática persistente en individuos vacunados, en particular aquellos que son inmunodeficientes . La IPV, al estar inactivada, no conlleva estos riesgos, pero no induce inmunidad de contacto. La IPV es más costosa y la logística de su distribución es más desafiante.
Nigeria es el último país que ha detenido oficialmente la transmisión endémica del poliovirus salvaje, con su último caso notificado en 2016. [58] De las tres cepas de WPV, el último caso salvaje registrado causado por el tipo 2 (WPV2) fue en 1999, y el WPV2 fue declarado erradicado en 2015. Se sabe por última vez que el tipo 3 (WPV3) causó polio en 2012, y se declaró erradicado en 2019. [59] Todos los casos de virus salvaje desde esa fecha se han debido al tipo 1 (WPV1). [60]
En agosto de 2024 [actualizar], Afganistán y Pakistán son los únicos dos países donde la enfermedad todavía está clasificada como endémica. [61] [62] Los casos recientes de polio surgen de dos fuentes, el poliovirus " salvaje " original (WPV) y las cepas de vacuna oral mutadas mucho más prevalentes, conocidas como poliovirus circulante derivado de la vacuna (cVDPV) o poliovirus variante. Las vacunas contra cada una de las tres cepas salvajes de polio han dado lugar a cepas de cVDPV, siendo el cVDPV2 el más destacado. El cVDPV causó 524 casos confirmados de polio paralítica en todo el mundo en 2023, y se detectó en 32 países. [60]Aunque los humanos son los únicos huéspedes naturales conocidos del poliovirus, los monos pueden ser infectados experimentalmente y se los ha utilizado durante mucho tiempo para estudiar el poliovirus. En 1990-91, dos laboratorios desarrollaron un modelo animal pequeño de poliomielitis. Se diseñaron ratones para que expresaran un receptor humano del poliovirus (hPVR). [63] [64]
A diferencia de los ratones normales, los ratones transgénicos con receptor de poliovirus (TgPVR) son susceptibles al poliovirus inyectado por vía intravenosa o intramuscular , y cuando se inyecta directamente en la médula espinal o el cerebro . [65] Tras la infección, los ratones TgPVR muestran signos de parálisis que se asemejan a los de la poliomielitis en humanos y monos, y los sistemas nerviosos centrales de los ratones paralizados son histocitoquímicamente similares a los de los humanos y los monos. Este modelo de ratón de infección por poliovirus humano ha demostrado ser una herramienta invaluable para comprender la biología y patogenicidad del poliovirus. [66]
Se han estudiado en profundidad tres tipos distintos de ratones TgPVR: [67]
El desarrollo del ratón TgPVR ha tenido un profundo efecto en la producción de la vacuna oral contra el poliovirus (OPV). Anteriormente, el control de la seguridad de la OPV debía realizarse utilizando monos, porque sólo los primates son susceptibles al virus. En 1999, la Organización Mundial de la Salud aprobó el uso del ratón TgPVR como método alternativo para evaluar la eficacia de la vacuna contra el poliovirus tipo 3. En 2000, el modelo de ratón fue aprobado para pruebas de vacunas contra el poliovirus tipo 1 y tipo 2. [69]
Una modificación del poliovirus, llamada PVSRIPO , se probó en ensayos clínicos tempranos como posible tratamiento para el cáncer. [70] A septiembre de 2022 [actualizar], se están realizando varios ensayos clínicos. [71] [72]
Un inconveniente del virus atenuado utilizado en la vacuna oral antipoliomielítica Sabin es su potencial para causar poliomielitis paralítica asociada a la vacuna (VAPP) en aproximadamente un individuo por cada 2,7 millones de dosis administradas. [73] Además, el virus vivo también puede circular en poblaciones subvacunadas (poliovirus circulante derivado de la vacuna, cVDPV) y con el tiempo puede revertir a una forma neurovirulenta que causa polio paralítica. [73] Los investigadores han desarrollado una cepa de vacuna de poliovirus tipo 2 que es genéticamente más estable y tiene menos probabilidades de recuperar la virulencia que la cepa original Sabin2, con tres modificaciones genéticas clave. [74] La vacuna derivada de esta cepa, el nuevo virus oral de la polio tipo 2 (nOPV2), recibió una licencia de emergencia en 2021 y, posteriormente, una licencia completa en diciembre de 2023. [75] Se están desarrollando vacunas genéticamente estabilizadas dirigidas a los tipos 1 y 3 del poliovirus, con la intención de que eventualmente reemplacen por completo a las vacunas Sabin. [76]
El poliovirus fue aislado por primera vez en 1909 por Karl Landsteiner y Erwin Popper . [35] La estructura del virus fue dilucidada por primera vez en 1958 utilizando difracción de rayos X por un equipo del Birkbeck College dirigido por Rosalind Franklin , [77] [78] mostrando que el virus del polio tiene simetría icosaédrica. [79]
En 1981, el genoma del poliovirus fue publicado por dos equipos diferentes de investigadores: por Vincent Racaniello y David Baltimore en el MIT [80] y por Naomi Kitamura y Eckard Wimmer en la Universidad Stony Brook . [81]
La estructura tridimensional del poliovirus fue determinada en 1985 por James Hogle en el Instituto de Investigación Scripps utilizando cristalografía de rayos X. [82]

En 1981, Racaniello y Baltimore utilizaron la tecnología del ADN recombinante para generar el primer clon infeccioso de un virus ARN animal, el poliovirus. El ADN que codifica el genoma ARN del poliovirus se introdujo en células de mamíferos cultivadas y se produjo el poliovirus infeccioso. [83] La creación del clon infeccioso impulsó la comprensión de la biología del poliovirus y se ha convertido en una tecnología estándar utilizada para estudiar muchos otros virus.
En 2002, el grupo de Eckard Wimmer de la Universidad Stony Brook logró sintetizar el virus de la poliomielitis a partir de su código químico, produciendo el primer virus sintético del mundo. [84] Los científicos convirtieron primero la secuencia de ARN publicada del virus de la poliomielitis, de 7741 bases de longitud, en una secuencia de ADN, ya que el ADN era más fácil de sintetizar. Se obtuvieron fragmentos cortos de esta secuencia de ADN por correo y se ensamblaron. Luego, una empresa de síntesis genética ensambló el genoma viral completo . Se incorporaron diecinueve marcadores al ADN sintetizado, de modo que pudiera distinguirse del virus de la poliomielitis natural. Se utilizaron enzimas para convertir el ADN nuevamente en ARN, su estado natural. Luego se utilizaron otras enzimas para traducir el ARN en un polipéptido, produciendo partículas virales funcionales. Todo este minucioso proceso llevó dos años. El virus sintético recién creado se inyectó en ratones transgénicos PVR, para determinar si la versión sintética podía causar enfermedades. El virus sintético pudo replicarse, infectar y causar parálisis o muerte en ratones. Sin embargo, la versión sintética era entre 1.000 y 10.000 veces más débil que el virus original, probablemente debido a uno de los marcadores añadidos. [85]