La ribonucleótido reductasa ( RNR ), también conocida como ribonucleósido difosfato reductasa ( rNDP ), es una enzima que cataliza la formación de desoxirribonucleótidos a partir de ribonucleótidos . [1] Cataliza esta formación eliminando el grupo 2'-hidroxilo del anillo de ribosa de los nucleósidos difosfato. Esta reducción produce desoxirribonucleótidos. [2] Los desoxirribonucleótidos a su vez se utilizan en la síntesis del ADN . La reacción catalizada por RNR se conserva estrictamente en todos los organismos vivos. [3] Además, RNR desempeña un papel fundamental en la regulación de la tasa total de síntesis de ADN, de modo que el ADN y la masa celular se mantienen en una proporción constante durante la división celular y la reparación del ADN . [4] Una característica algo inusual de la enzima RNR es que cataliza una reacción que se produce a través de un mecanismo de acción de radicales libres . [5] [6] Los sustratos para RNR son ADP , GDP , CDP y UDP . El dTDP (desoxitimidina difosfato) es sintetizado por otra enzima ( timidilato quinasa ) a partir de dTMP (desoxitimidina monofosfato).
Las ribonucleótidos reductasas se dividen en tres clases. Las enzimas RNR de clase I se construyen a partir de subunidades alfa grandes y subunidades beta pequeñas que se asocian para formar un tetrámero heterodimérico activo . Al reducir los NDP a 2'-dNDP, la enzima cataliza la síntesis de novo de desoxirribonucleótidos (dNTP), que son precursores de la síntesis de ADN y esenciales para la proliferación celular . [7] Los RNR de clase II producen un radical 5'-desoxiadenosilo mediante escisión homolítica del enlace C-Co en la adenosilcobalamina. Además, los RNR de clase III contienen un radical glicilo estable. [8]
Los humanos portan RNR de Clase I. La subunidad alfa está codificada por el gen RRM1 mientras que existen dos isoformas de la subunidad beta, codificadas por los genes RRM2 y RRM2B:
Cada monómero alfa de Clase I consta de tres dominios : [9]
En Pfam , el segundo dominio se ha interpretado como dos dominios separados:
La subunidad beta de Clase I suele contener un centro dimetálico y un radical tirosilo estable . En los seres humanos, la subunidad beta depende de un cofactor di-hierro. En E. coli , el radical tirosilo está ubicado en la posición 122 (Y122), proporcionando el radical estable para las subunidades RNR2 de Clase I. [13] En A. aegypti , este radical tirosilo está ubicado en la posición 184 (Y184). [14] El radical tirosilo está profundamente enterrado dentro de la proteína en un ambiente hidrofóbico, ubicado cerca del centro de hierro que se utiliza en la estabilización de un radical tirosilo. La estructura de dos hierros unidos por μ-oxo está dominada por ligandos que sirven como sitios de unión del hierro: cuatro carboxilatos [ aspartato (D146), glutamato (E177, E240 y E274)] y dos histidinas (H180 y H277). [14] La asociación se produce entre el extremo C de RNR2 y el extremo C de RNR1. [9] La actividad enzimática depende de la asociación de las subunidades RNR1 y RNR2. El sitio activo consta de los grupos ditiol activos de RNR1, así como del centro diférrico y el radical tirosilo de la subunidad RNR2.
Otros residuos de RNR2, como el aspartato (D273), el triptófano (W48) y la tirosina (Y356) estabilizan aún más el radical tirosilo del sitio activo, permitiendo así la transferencia de electrones. [9] Estos residuos ayudan en la transferencia del electrón radical de la tirosina (Y122) de RNR2 a la cisteína (C439) de RNR1. La transferencia de electrones comienza en la tirosina RNR2 (Y122) y continúa en RNR2 hasta el triptófano (W48), que está separado de la tirosina RNR1 (Y731) por 2,5 nanómetros . La transferencia de electrones de RNR2 a RNR1 se produce a través de tirosina (Y356 a Y731) y continúa a través de tirosina (Y730) hasta cisteína (C439) en el sitio activo. [15] Las mutaciones dirigidas al sitio de la estructura primaria de RNR indican que todos los residuos citados anteriormente participan en la transferencia a larga distancia del radical libre al sitio activo. [9]
En los mosquitos A. aegypti , RNR1 retiene la mayoría de los residuos de aminoácidos cruciales, incluidos el aspartato (D64) y la valina (V292 o V284), que son necesarios en la regulación alostérica ; residuos de prolina (P210 y P610), leucina (L453 y L473) y metionina (M603) que se encuentran en el sitio activo hidrofóbico; residuos de cisteína (C225, C436 y C451) que participan en la eliminación de un átomo de hidrógeno y la transferencia del electrón radical en el sitio activo; residuos de cisteína (C225 y C436), asparagina (N434) y glutamato (E441) que se unen al sustrato ribonucleótido; residuos de tirosina (Y723 e Y743) que dictan la transferencia de radicales; y residuos de cisteína (C838 y C841) que se utilizan en la regeneración de grupos ditiol en el sitio activo. [14]

La enzima ribonucleótido reductasa (RNR) cataliza la síntesis de novo de dNDP. [16] La catálisis de ribonucleósidos 5'-difosfatos (NDP) implica una reducción en el carbono 2' de la ribosa 5-fosfato para formar 2'-desoxirribonucleósidos 5'-difosfatos (dNDP) reducidos con derivados 2'-desoxi. Esta reducción se inicia con la generación de un radical libre. Después de una única reducción, la RNR requiere electrones donados de los grupos ditiol de la proteína tiorredoxina . La regeneración de la tioredoxina se produce cuando el fosfato de dinucleótido de nicotinamida y adenina ( NADPH ) proporciona dos átomos de hidrógeno que se utilizan para reducir los grupos disulfuro de la tioredoxina.
Tres clases de RNR tienen mecanismos similares para la reducción de NDP, pero difieren en el dominio que genera el radical libre, el metal específico en la estructura de la metaloproteína y los donantes de electrones. Todas las clases utilizan química de radicales libres. [9] Las reductasas de clase I utilizan un centro de hierro con conversión ferrosa a férrica para generar un radical libre tirosilo. La reducción de los sustratos de NDP se produce en condiciones aeróbicas. Las reductasas de clase I se dividen en IA e IB debido a diferencias en la regulación. Las reductasas de clase IA se distribuyen en eucariotas , eubacterias , bacteriófagos y virus . Las reductasas de clase IB se encuentran en las eubacterias. Las reductasas de clase IB también pueden utilizar un radical generado con la estabilización de un centro de manganeso binuclear . Las reductasas de clase II generan el radical libre 5'-desoxiadenosilo a partir de cobalamina (coenzima B12) y tienen una estructura más simple que las reductasas de clase I y clase III. La reducción de NDP o ribonucleótidos 5'-trifosfatos (NTP) se produce en condiciones aeróbicas o anaeróbicas. Las reductasas de clase II se distribuyen en arqueobacterias , eubacterias y bacteriófagos. Las reductasas de clase III utilizan un radical de glicina generado con la ayuda de una S-adenosil metionina y un centro de azufre de hierro. La reducción de los NTP se limita a condiciones anaeróbicas. Las reductasas de clase III se distribuyen en arqueobacterias, eubacterias y bacteriófagos. [9] [14] Los organismos no se limitan a tener una clase de enzimas. Por ejemplo, E. coli tiene RNR de clase I y clase III.
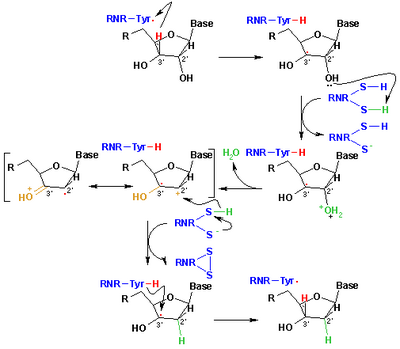
El mecanismo actualmente aceptado para la reducción de ribonucleótidos a desoxirribonucleótidos se representa en el siguiente esquema. El primer paso implica la abstracción del 3'-H del sustrato 1 por el radical Cys439. Posteriormente, la reacción implica la eliminación de una molécula de agua del carbono C-2' del ribonucleótido, catalizada por Cys225 y Glu441. En el tercer paso se produce una transferencia de átomos de hidrógeno desde Cys225 al carbono C-2' del radical 2'-cetilo 3, previa transferencia de protones desde Cys462 a Cys225. Al final de esta etapa, se obtienen un puente disulfuro radical aniónico y el intermedio cetona de capa cerrada 4. Este intermediario se ha identificado durante la conversión de varios análogos de sustrato sustituidos en 2', así como con el sustrato natural [17] que interactúa con mutantes enzimáticos. El siguiente paso es la oxidación del puente disulfuro aniónico, con la reducción concomitante del sustrato, generando 5. La densidad de espín cambia de los átomos de azufre al átomo C-3' del sustrato, con transferencia simultánea de protones desde Glu441 al carbono C. -3'. El último paso es el inverso del primer paso e implica una transferencia de hidrógeno de Cys439 a C-3', regenerando el radical inicial y dando como resultado el producto final 6.
Los modelos teóricos de algunos pasos de estos mecanismos utilizando el modelo completo de la proteína R1 se pueden encontrar en los estudios realizados por Cerqueira et al. . [18] [19]

La RNR de clase I comprende las subunidades RNR1 y RNR2, que pueden asociarse para formar un tetrámero heterodimérico. [5] RNR1 contiene ambos sitios alostéricos, que median la regulación de la especificidad y actividad del sustrato. [11] Dependiendo de la configuración alostérica, uno de los cuatro ribonucleótidos se une al sitio activo.
La regulación de RNR está diseñada para mantener cantidades equilibradas de dNTP. La unión de moléculas efectoras aumenta o disminuye la actividad RNR. Cuando el ATP se une al sitio de actividad alostérica, activa el RNR. Por el contrario, cuando dATP se une a este sitio, desactiva RNR. [9] Además de controlar la actividad, el mecanismo alostérico también regula la especificidad del sustrato y garantiza que la enzima produzca una cantidad igual de cada dNTP para la síntesis de ADN. [9] En todas las clases, la unión de ATP o dATP al sitio alostérico induce la reducción de citidina 5'-difosfato (CDP) y uridina 5'-difosfato (UDP); 2'-desoxiguanosina 5'-trifosfato (dGTP) induce la reducción de adenosina 5'-difosfato (ADP); y el 2'-desoxitimidina 5'-trifosfato (dTTP) induce la reducción de guanosina 5'-difosfato (PIB) (Figura 1).
Las reductasas de clase IB no son inhibidas por dATP porque carecen de aproximadamente 50 aminoácidos N-terminales necesarios para el sitio de actividad alostérica. [20] Además, es importante que la actividad de la ribonucleótido reductasa esté bajo control transcripcional y postranscripcional porque la síntesis de ADN libre de daños depende de un conjunto equilibrado de desoxirribonucleótidos. [21] Las células eucariotas con reductasas de clase IA tienen un mecanismo de control negativo para desactivar la síntesis de dNTP a medida que se acumulan. Este mecanismo protege a la célula de los efectos tóxicos y mutagénicos que pueden surgir de la sobreproducción de dNTP porque los cambios en las reservas equilibradas de dNTP provocan daño en el ADN y muerte celular. [22] [23] Aunque la sobreproducción de dNTP o un suministro desequilibrado de ellos puede provocar una mala incorporación de nucleótidos en el ADN, el suministro de dNTP puede permitir la reparación del ADN. p53R2 es una pequeña subunidad de la ribonucleótido reductasa que puede inducir dicha reparación. Los cambios dentro de este homólogo R2 inducido por p53 pueden provocar un agotamiento del ADN mitocondrial y, en consecuencia, p53R2 constituye un factor importante en el suministro de dNTP. [24]
RNR puede utilizar el modelo de regulación alostérica de morfeína . [25]
Generalmente, los inhibidores de RNR de Clase I se pueden dividir en tres grupos principales: inhibidores de la traducción, que bloquean la síntesis de la enzima; inhibidores de la dimerización que impiden la asociación de las dos subunidades RNR (R1 y R2); e inhibidores catalíticos que inactivan la subunidad R1 y/o la subunidad R2. [18]
La RNR de clase I puede ser inhibida por péptidos similares al extremo C de RNR2. Estos péptidos pueden competir con RNR2 por la unión a RNR1 y, como resultado, RNR1 no forma un complejo enzimáticamente activo con RNR2. [26] [27] Aunque el extremo C de las proteínas RNR2 es diferente entre especies, RNR2 puede interactuar con RNR1 entre especies. [28] Cuando el extremo C-terminal de RNR2 de ratón se reemplazó con los residuos de aminoácidos del extremo C-terminal de RNR2 de E. coli (7 o 33), la subunidad quimérica RNR2 todavía se une a las subunidades RNR1 de ratón. Sin embargo, carecen de actividad enzimática debido probablemente a la eliminación de residuos implicados en la transferencia del electrón del radical libre desde la subunidad RNR2 a la subunidad RNR1. [27]
Los péptidos pequeños pueden inhibir específicamente la unión de las subunidades RNR2 con RNR1 cuando comparten una similitud significativa con el extremo C terminal normal de RNR2. [29] Esta inhibición de la unión de RNR2 a RNR1 se ha probado con éxito en el RNR del virus del herpes simple (VHS). Cuando se utilizó un oligómero de 7 aminoácidos (GAVVNDL) truncado del extremo C de la subunidad RNR2 en ensayos de competición, impidió que el RNR2 normal formara un complejo enzimáticamente activo con RNR1. [30] También se han utilizado con éxito otros inhibidores peptídicos pequeños similares al extremo C terminal de RNR2 para inhibir la actividad enzimática RNR del HSV y, por tanto, la replicación del HSV. [31] En modelos de ratones con queratitis estromal y neovascularización corneal ( enfermedad ocular por HSV ), se ha informado que un pequeño análogo C-terminal de RNR2, BILD 1263, inhibe la RNR y es eficaz para prevenir estas enfermedades. [32] En algunos casos, aunque el tratamiento con pequeños análogos C-terminales puede no detener la propagación de la enfermedad, aún pueden ayudar en la curación. En el HSV resistente al aciclovir (PAAr5), se ha informado que un pequeño inhibidor peptídico BILD 1633 es de 5 a 10 veces más potente que BILD 1263 contra la infección cutánea por PAAr5. [33] Un enfoque de terapia combinada (BILD 1633 y aciclovir) es más eficaz para curar lesiones tópicas en ratones. Estos datos sugieren que los inhibidores de péptidos pequeños que compiten con RNR2 por la unión a RNR1 son útiles para prevenir la propagación del HSV.
El galio inhibe el RNR2 sustituyendo al Fe 3+ en el sitio activo. El maltolato de galio es una forma de galio biodisponible por vía oral que aprovecha esta actividad inhibidora para tratar el cáncer, las infecciones y otras enfermedades. [34]
Los fármacos hidroxiurea [35] y motexafina gadolinio interfieren con la acción de esta enzima. [36]