
La replicación del ADN eucariota es un mecanismo conservado que limita la replicación del ADN a una sola vez por ciclo celular. La replicación del ADN cromosómico es fundamental para la duplicación de una célula y es necesaria para el mantenimiento del genoma eucariota .
La replicación del ADN es la acción de las polimerasas de ADN que sintetizan una cadena de ADN complementaria a la cadena molde original. Para sintetizar el ADN, las helicasas de ADN desenrollan el ADN bicatenario antes que las polimerasas, formando una horquilla de replicación que contiene dos cadenas molde de cadena sencilla. Los procesos de replicación permiten copiar una sola doble hélice de ADN en dos hélices de ADN, que se dividen en las células hijas en la mitosis . Las principales funciones enzimáticas llevadas a cabo en la horquilla de replicación están bien conservadas desde procariotas hasta eucariotas , pero la maquinaria de replicación en la replicación del ADN eucariota es un complejo mucho más grande, que coordina muchas proteínas en el sitio de replicación, formando el replisoma . [1]
El replisoma es responsable de copiar la totalidad del ADN genómico en cada célula proliferativa. Este proceso permite la transmisión de alta fidelidad de la información genética/hereditaria de la célula parental a la célula hija y, por lo tanto, es esencial para todos los organismos. Gran parte del ciclo celular se basa en garantizar que la replicación del ADN se produzca sin errores. [1]
En la fase G 1 del ciclo celular se inician muchos de los procesos reguladores de la replicación del ADN. En los eucariotas, la gran mayoría de la síntesis de ADN se produce durante la fase S del ciclo celular, y todo el genoma debe desenrollarse y duplicarse para formar dos copias hijas. Durante la fase G 2 , se corrige cualquier ADN dañado o errores de replicación. Finalmente, una copia de los genomas se segrega en cada célula hija en la mitosis o fase M. [2] Cada una de estas copias hijas contiene una hebra del ADN dúplex parental y una hebra antiparalela naciente.
Este mecanismo se conserva desde los procariotas hasta los eucariotas y se conoce como replicación de ADN semiconservativa . El proceso de replicación semiconservativa para el sitio de replicación de ADN es una estructura de ADN en forma de horquilla, la horquilla de replicación, donde la hélice de ADN está abierta, o desenrollada, exponiendo los nucleótidos de ADN no apareados para su reconocimiento y el apareamiento de bases para la incorporación de nucleótidos libres en el ADN bicatenario. [3]
El inicio de la replicación del ADN eucariota es la primera etapa de la síntesis del ADN, en la que se desenrolla la doble hélice del ADN y se produce un evento de cebado inicial por parte de la ADN polimerasa α en la cadena líder. El evento de cebado en la cadena rezagada establece una horquilla de replicación. El cebado de la hélice del ADN consiste en la síntesis de un cebador de ARN para permitir la síntesis del ADN por parte de la ADN polimerasa α. El cebado se produce una vez en el origen de la cadena líder y al comienzo de cada fragmento de Okazaki en la cadena rezagada.
La replicación comienza en los orígenes de replicación . Las secuencias de ADN que contienen estos sitios se aislaron inicialmente a fines de la década de 1970 sobre la base de su capacidad para soportar la replicación de plásmidos , de ahí la designación de secuencias de replicación autónoma (ARS). Los orígenes varían ampliamente en su eficiencia, y algunos se utilizan en casi todos los ciclos celulares, mientras que otros pueden usarse en solo una de cada mil fases S. [4] El número total de ARS de levadura es de al menos 1600, pero puede ser más de 5000 si se cuentan menos sitios activos, [5] es decir, puede haber una ARS cada 2000 a 8000 pares de bases.
Múltiples proteínas replicativas se ensamblan y se disocian de estos orígenes replicativos para iniciar la replicación del ADN. [6] siendo la formación del complejo de prerreplicación (pre-RC) un intermediario clave en el proceso de iniciación de la replicación.
La asociación del complejo de reconocimiento de origen (ORC) con un origen de replicación recluta a la proteína del ciclo de división celular 6 (Cdc6) para formar una plataforma para la carga de las proteínas del complejo de mantenimiento de minicromosomas (Mcm 2-7) , facilitada por la proteína de licencia de cromatina y factor 1 de replicación de ADN (Cdt1). El ORC, Cdc6 y Cdt1 juntos son necesarios para la asociación estable del complejo Mcm2-7 con los orígenes replicativos durante la fase G 1 del ciclo celular. [7]
Los orígenes eucariotas de replicación controlan la formación de varios complejos proteicos que conducen al ensamblaje de dos horquillas de replicación de ADN bidireccionales. Estos eventos se inician con la formación del complejo de pre-replicación (pre-RC) en los orígenes de replicación. Este proceso tiene lugar en la etapa G 1 del ciclo celular. La formación de pre-RC implica el ensamblaje ordenado de muchos factores de replicación, incluido el complejo de reconocimiento de origen (ORC), la proteína Cdc6, la proteína Cdt1 y las proteínas de mantenimiento de minicromosomas (Mcm2-7). [8] [9] Una vez que se forma el pre-RC, la activación del complejo es desencadenada por dos quinasas , la quinasa dependiente de ciclina 2 (CDK) y la quinasa dependiente de Dbf4 (DDK) que ayudan a la transición del pre-RC al complejo de iniciación antes del inicio de la replicación del ADN. Esta transición implica el ensamblaje ordenado de factores de replicación adicionales para desenrollar el ADN y acumular las múltiples polimerasas de ADN eucariotas alrededor del ADN desenrollado. Un aspecto central de la cuestión de cómo se establecen las horquillas de replicación bidireccionales en los orígenes de replicación es el mecanismo por el cual ORC recluta dos complejos Mcm2-7 cabeza a cabeza en cada origen de replicación para formar el complejo de pre-replicación. [10] [11] [12]
El primer paso en el ensamblaje del complejo de pre-replicación (pre-RC) es la unión del complejo de reconocimiento de origen (ORC) al origen de replicación. En la mitosis tardía, la proteína Cdc6 se une al ORC unido seguido de la unión del complejo Cdt1-Mcm2-7. [13] ORC, Cdc6 y Cdt1 son necesarios para cargar el complejo de mantenimiento de minicromosomas de seis proteínas (Mcm 2-7) en el ADN. El ORC es un complejo proteico de seis subunidades, Orc1p-6, que selecciona los sitios de origen replicativo en el ADN para el inicio de la replicación y la unión de ORC a la cromatina se regula a través del ciclo celular. [8] [14] Generalmente, la función y el tamaño de las subunidades de ORC se conservan en muchos genomas eucariotas con la diferencia de que sus sitios de unión al ADN divergen.
El complejo de reconocimiento de origen más estudiado es el de Saccharomyces cerevisiae o levadura, que se sabe que se une a la secuencia de replicación autónoma (ARS). [15] El ORC de S. cerevisiae interactúa específicamente con los elementos A y B1 de los orígenes de replicación de levadura, abarcando una región de 30 pares de bases . [16] La unión a estas secuencias requiere ATP . [8] [16]
Se ha determinado la estructura atómica del ORC de S. cerevisiae unido al ADN de ARS. [16] Orc1, Orc2, Orc3, Orc4 y Orc5 rodean el elemento A mediante dos tipos de interacciones, no específicas de base y específicas de base, que doblan el ADN en el elemento A. Las cinco subunidades entran en contacto con la cadena principal de fosfato de azúcar en múltiples puntos del elemento A para formar un agarre apretado sin especificidad de base. Orc1 y Orc2 entran en contacto con el surco menor del elemento A, mientras que un dominio de hélice alada de Orc4 entra en contacto con los grupos metilo de las T invariantes en el surco mayor del elemento A a través de una hélice de inserción (IH). La ausencia de esta IH en los metazoos [16] explica la falta de especificidad de secuencia en el ORC humano. [17] La eliminación de la IH del ORC de Sc hace que pierda su especificidad para el elemento A y se una de forma promiscua y preferencial (83 %) a las regiones promotoras. [18] El ADN ARS también se dobla en el elemento B1 a través de interacciones con Orc2, Orc5 y Orc6. [16] La flexión del ADN de origen por ORC parece estar conservada evolutivamente, lo que sugiere que puede ser necesaria para el mecanismo de carga del complejo Mcm2-7. [16] [19]
Cuando el ORC se une al ADN en los orígenes de replicación, sirve como andamiaje para el ensamblaje de otros factores de iniciación clave del complejo prerreplicativo. [20] Este ensamblaje del complejo prerreplicativo durante la etapa G 1 del ciclo celular es necesario antes de la activación de la replicación del ADN durante la fase S. [21] La eliminación de al menos parte del complejo (Orc1) del cromosoma en la metafase es parte de la regulación del ORC de los mamíferos para garantizar que se elimine la formación del complejo prerreplicativo antes de la finalización de la metafase. [22]
La unión de la proteína del ciclo de división celular 6 (Cdc6) al complejo de reconocimiento de origen (ORC) es un paso esencial en el ensamblaje del complejo de pre-replicación (pre-RC) en los orígenes de replicación. Cdc6 se une al ORC en el ADN de una manera dependiente de ATP, lo que induce un cambio en el patrón de unión al origen que requiere Orc1 ATPasa . [23] Cdc6 requiere ORC para asociarse con la cromatina y, a su vez, es necesario para que el heptámero Cdt1-Mcm2-7 [13] se una a la cromatina. [24] El complejo ORC-Cdc6 forma una estructura en forma de anillo y es análogo a otras máquinas proteicas dependientes de ATP. Los niveles y la actividad de Cdc6 regulan la frecuencia con la que se utilizan los orígenes de replicación durante el ciclo celular.
La proteína factor 1 de replicación de ADN y licencia de cromatina (Cdt1) es necesaria para la licencia de cromatina para la replicación de ADN. [25] [26] En S. cerevisiae , Cdt1 facilita la carga del complejo Mcm2-7 uno a la vez en el cromosoma estabilizando la estructura de anillo abierto zurdo del hexámero único Mcm2-7. [13] [27] [28] Se ha demostrado que Cdt1 se asocia con el extremo C de Cdc6 para promover cooperativamente la asociación de proteínas Mcm a la cromatina. [29] La estructura crio-EM del complejo OCCM (ORC-Cdc6-Cdt1-MCM) muestra que Cdt1-CTD interactúa con Mcm6-WHD. [30] En los metazoos, la actividad de Cdt1 durante el ciclo celular está estrechamente regulada por su asociación con la proteína geminina , que inhibe la actividad de Cdt1 durante la fase S para evitar la re-replicación del ADN y evita su ubiquitinación y posterior proteólisis . [31]
Las proteínas de mantenimiento de minicromosomas (Mcm) recibieron su nombre a raíz de una prueba genética para detectar mutantes de iniciación de replicación de ADN en S. cerevisiae que afectan la estabilidad del plásmido de una manera específica de ARS . [32] Mcm2, Mcm3, Mcm4, Mcm5, Mcm6 y Mcm7 forman un complejo hexamérico que tiene una estructura de anillo abierto con un espacio entre Mcm2 y Mcm5. [13] El ensamblaje de las proteínas Mcm en la cromatina requiere la función coordinada del complejo de reconocimiento de origen (ORC), Cdc6 y Cdt1. [33] Una vez que las proteínas Mcm se han cargado en la cromatina, ORC y Cdc6 se pueden eliminar de la cromatina sin impedir la replicación posterior del ADN. Esta observación sugiere que la función principal del complejo de pre-replicación es cargar correctamente las proteínas Mcm. [34]

Las proteínas Mcm en la cromatina forman un doble hexámero cabeza a cabeza con los dos anillos ligeramente inclinados, torcidos y descentrados para crear una torcedura en el canal central donde el ADN unido es capturado en la interfaz de los dos anillos. [35] [36] Cada anillo hexamérico Mcm2-7 primero sirve como andamio para el ensamblaje del replisoma y luego como núcleo de la helicasa catalítica CMG (Cdc45-MCM-GINS), que es un componente principal del replisoma. Cada proteína Mcm está altamente relacionada con todas las demás, pero las secuencias únicas que distinguen a cada uno de los tipos de subunidades [18] se conservan en los eucariotas. Todos los eucariotas tienen exactamente seis análogos de la proteína Mcm que caen cada uno en una de las clases existentes (Mcm2-7), lo que indica que cada proteína Mcm tiene una función única e importante. [37] [11]
Las proteínas de mantenimiento de minicromosomas son necesarias para la actividad de la helicasa del ADN. La inactivación de cualquiera de las seis proteínas Mcm durante la fase S impide irreversiblemente la progresión adicional de la horquilla de replicación, lo que sugiere que la helicasa no se puede reciclar y debe ensamblarse en los orígenes de replicación. [38] Junto con la actividad de helicasa del complejo de proteínas de mantenimiento de minicromosomas, el complejo también tiene actividad ATPasa asociada. [39] Los estudios han demostrado que dentro del complejo de proteínas Mcm hay pares catalíticos específicos de proteínas Mcm que funcionan juntas para coordinar la hidrólisis de ATP. [40] Estos estudios, confirmados por las estructuras crio-EM de los complejos Mcm2-7, [13] [35] mostraron que el complejo Mcm es un hexámero con subunidades dispuestas en un anillo en el orden de Mcm2-Mcm6-Mcm4-Mcm7-Mcm3-Mcm5-. Ambos miembros de cada par catalítico contribuyen a la conformación que permite la unión y la hidrólisis del ATP y la mezcla de subunidades activas e inactivas presumiblemente permite que el complejo hexamérico Mcm complete la unión y la hidrólisis del ATP como un todo para crear una actividad ATPasa coordinada. [41]
La localización nuclear de las proteínas de mantenimiento de minicromosomas está regulada en células de levadura en gemación. [42] [43] Las proteínas Mcm están presentes en el núcleo en la etapa G 1 y la fase S del ciclo celular, pero se exportan al citoplasma durante la etapa G 2 y la fase M. Se requiere un complejo Mcm de seis subunidades completo e intacto para ingresar al núcleo celular. [44] En S. cerevisiae , la exportación nuclear es promovida por la actividad de la quinasa dependiente de ciclina (CDK). Las proteínas Mcm que están asociadas con la cromatina están protegidas de la maquinaria de exportación de CDK debido a la falta de accesibilidad a CDK. [45]
Durante la etapa G 1 del ciclo celular, los factores de iniciación de la replicación, el complejo de reconocimiento de origen (ORC), Cdc6, Cdt1 y el complejo proteico de mantenimiento de minicromosomas (Mcm), se unen secuencialmente al ADN para formar un dímero cabeza a cabeza del complejo de anillo MCM, conocido como el complejo de pre-replicación (pre-RC). Mientras que el pre-RC de levadura forma un complejo de ADN cerrado, [35] [36] [46] el pre-RC humano forma un complejo abierto. [47] En la transición de la etapa G 1 a la fase S del ciclo celular, la proteína quinasa dependiente de ciclina específica de la fase S (CDK) y la quinasa Cdc7/Dbf4 (DDK) transforman el pre-RC inerte en un complejo activo capaz de ensamblar dos replisomas bidireccionales. Las estructuras CryoEM [48] [49] [50] mostraron que dos DDK se acoplan independientemente a la interfaz del doble hexámero MCM que se extiende a lo largo de los dos anillos. La fosforilación secuencial de múltiples sustratos en los NTE de Mcm4, Mcm2 y Mcm6 se logra mediante un mecanismo de oscilación mediante el cual Dbf4 asume diferentes estados de oscilación para posicionar a Cdc7 sobre sus múltiples sustratos. [50] La fosforilación del doble hexámero MCM, en particular el Mcm4-NSD, por DDK es esencial para la viabilidad en levadura. [51] El reclutamiento de Cdc45 y GINS sigue después de la activación de los MCM por DDK y CDK.
La proteína del ciclo de división celular 45 (Cdc45) es un componente fundamental para la conversión del complejo prerreplicativo en el complejo de iniciación. La proteína Cdc45 se ensambla en los orígenes de replicación antes de la iniciación y es necesaria para que la replicación comience en Saccharomyces cerevisiae , y tiene un papel esencial durante la elongación. Por lo tanto, Cdc45 tiene papeles centrales tanto en las fases de iniciación como de elongación de la replicación del ADN cromosómico. [52]
Cdc45 se asocia con la cromatina después del comienzo de la iniciación en la etapa G 1 tardía y durante la fase S del ciclo celular. Cdc45 se asocia físicamente con Mcm5 y muestra interacciones genéticas con cinco de los seis miembros de la familia de genes Mcm y el gen ORC2 . [53] [54] La carga de Cdc45 en la cromatina es fundamental para cargar otras proteínas de replicación, incluidas la ADN polimerasa α , la ADN polimerasa ε, la proteína de replicación A (RPA) y el antígeno nuclear de células proliferantes (PCNA) en la cromatina. [55] [56] [57] [58] Dentro de un sistema sin núcleo de Xenopus , se ha demostrado que Cdc45 es necesario para el desenrollado del ADN plasmídico. [58] El sistema sin núcleo de Xenopus también demuestra que el desenrollado del ADN y la unión estrecha de RPA a la cromatina solo ocurren en presencia de Cdc45. [55]
La unión de Cdc45 a la cromatina depende de la actividad de la cinasa Clb-Cdc28, así como de la actividad funcional de Cdc6 y Mcm2, lo que sugiere que Cdc45 se asocia con el pre-RC después de la activación de las cinasas dependientes de ciclina (CDK) de la fase S. Como lo indican el momento y la dependencia de CDK, la unión de Cdc45 a la cromatina es crucial para el compromiso con el inicio de la replicación del ADN. Durante la fase S, Cdc45 interactúa físicamente con las proteínas Mcm en la cromatina; sin embargo, la disociación de Cdc45 de la cromatina es más lenta que la de Mcm, lo que indica que las proteínas se liberan por diferentes mecanismos. [37]
Las seis proteínas de mantenimiento de minicromosomas y Cdc45 son esenciales durante la iniciación y la elongación para el movimiento de las horquillas de replicación y para el desenrollado del ADN. Los GINS son esenciales para la interacción de Mcm y Cdc45 en los orígenes de la replicación durante la iniciación y luego en las horquillas de replicación del ADN a medida que progresa el replisoma. [59] [60] El complejo GINS está compuesto por cuatro proteínas pequeñas Sld5 (Cdc105), Psf1 (Cdc101), Psf2 (Cdc102) y Psf3 (Cdc103), GINS representa 'go, ichi, ni, san' que significa '5, 1, 2, 3' en japonés. [61] Cdc45, Mcm2-7 y GINS juntos forman la helicasa CMG, [62] la helicasa replicativa del replisoma. Aunque el complejo Mcm2-7 por sí solo tiene una actividad de helicasa débil [63], Cdc45 y GINS son necesarios para una actividad de helicasa robusta [64] [65]
Mcm10 es esencial para la replicación cromosómica e interactúa con la helicasa de mantenimiento del minicromosoma 2-7 que está cargada en una forma inactiva en los orígenes de la replicación del ADN. [66] [67] Mcm10 también acompaña a la ADN polimerasa catalítica α y ayuda a estabilizar la polimerasa en las horquillas de replicación. [68] [69]
Al inicio de la fase S, el complejo prerreplicativo debe ser activado por dos quinasas específicas de la fase S para formar un complejo de iniciación en un origen de replicación. Una quinasa es la quinasa Cdc7-Dbf4 llamada quinasa dependiente de Dbf4 (DDK) y la otra es la quinasa dependiente de ciclina (CDK). [70] Los ensayos de unión a la cromatina de Cdc45 en levadura y Xenopus han demostrado que un evento posterior a la acción de CDK es la carga de Cdc45 en la cromatina. [71] [72] Se ha especulado que Cdc6 es un objetivo de la acción de CDK, debido a la asociación entre Cdc6 y CDK, y la fosforilación dependiente de CDK de Cdc6. Se ha considerado que la fosforilación dependiente de CDK de Cdc6 es necesaria para la entrada en la fase S. [73]
Tanto las subunidades catalíticas de DDK, Cdc7, como la proteína activadora, Dbf4, se conservan en eucariotas y son necesarias para el inicio de la fase S del ciclo celular. [74] [75] Tanto Dbf4 como Cdc7 son necesarias para la carga de Cdc45 en los orígenes de replicación de la cromatina. El objetivo de la unión de la quinasa DDK es la forma unida a la cromatina del complejo Mcm. [76] [77] Las estructuras cryoEM de alta resolución mostraron que la subunidad Dbf4 de DDK se extiende a lo largo de la interfaz del hexámero del MCM-DH unido al ADN, contactando Mcm2 de un hexámero y Mcm4/6 del hexámero opuesto. [48] [49] [50] Mcm2, Mcm4 y Mcm6 son todos sustratos de fosforilación por DDK [78] [74] pero solo el dominio rico en serina/treonina (NSD) N-terminal de Mcm4 es un objetivo esencial de DDK. [50] [51] La fosforilación del NSD conduce a la activación de la actividad de la helicasa Mcm.
Sld3, Sld2 y Dpb11 interactúan con muchas proteínas de replicación. Sld3 y Cdc45 forman un complejo que se asocia con el pre-RC en los orígenes tempranos de replicación incluso en la fase G11 y con los orígenes posteriores de replicación en la fase S de una manera mutuamente dependiente de Mcm. [79] [80] Dpb11 y Sld2 interactúan con la polimerasa ɛ y los experimentos de entrecruzamiento han indicado que Dpb11 y la polimerasa ɛ coprecipitan en la fase S y se asocian con los orígenes de replicación. [81] [82]
La Sld3 y la Sld2 son fosforiladas por la CDK, lo que permite que las dos proteínas replicativas se unan a la Dpb11. La Dpb11 tenía dos pares de dominios BRCA1 C Terminus (BRCT), que se conocen como dominios de unión a fosfopéptidos. [83] El par N-terminal de los dominios BRCT se une a la Sld3 fosforilada, y el par C-terminal se une a la Sld2 fosforilada. Ambas interacciones son esenciales para la activación dependiente de la CDK de la gemación del ADN en la levadura. [84]
Dpb11 también interactúa con GINS y participa en los pasos de iniciación y elongación de la replicación del ADN cromosómico. [60] [85] [86] GINS es una de las proteínas de replicación que se encuentran en las horquillas de replicación y forma un complejo con Cdc45 y Mcm.
Estas interacciones dependientes de la fosforilación entre Dpb11, Sld2 y Sld3 son esenciales para la activación dependiente de CDK de la replicación del ADN y, mediante el uso de reactivos de reticulación en algunos experimentos, se identificó un complejo frágil llamado complejo de precarga (pre-LC). Este complejo contiene Pol ɛ, GINS, Sld2 y Dpb11. Se ha descubierto que el pre-LC se forma antes de cualquier asociación con los orígenes de una manera dependiente de CDK y DDK, y la actividad de CDK regula el inicio de la replicación del ADN a través de la formación del pre-LC. [87]

La formación del complejo prerreplicativo (pre-RC) marca los sitios potenciales para el inicio de la replicación del ADN. En consonancia con el complejo de mantenimiento de minicromosomas que rodea el ADN bicatenario, la formación del pre-RC no conduce al desenrollado inmediato del ADN de origen ni al reclutamiento de las ADN polimerasas. En cambio, el pre-RC que se forma durante la fase G 1 del ciclo celular solo se activa para desenrollar el ADN e iniciar la replicación después de que las células pasan de la fase G 1 a la fase S del ciclo celular. [2]
Una vez que se forma el complejo de iniciación y las células pasan a la fase S, el complejo se convierte en un replisoma. El complejo replisoma eucariota es responsable de coordinar la replicación del ADN. La replicación en las cadenas líder y rezagada la realizan las ADN polimerasas ε y δ. Muchos factores del replisoma, incluidos Claspin, And1, el factor de replicación C, el cargador de pinzas y el complejo de protección de la horquilla, son responsables de regular las funciones de la polimerasa y coordinar la síntesis de ADN con el desenrollado de la cadena molde por el complejo Cdc45-Mcm-GINS. A medida que el ADN se desenrolla, el número de giros disminuye. Para compensar esto, el número de retorcimientos aumenta, introduciendo superenrollamientos positivos en el ADN. Estos superenrollamientos harían que la replicación del ADN se detuviera si no se eliminaran. Las topoisomerasas son responsables de eliminar estos superenrollamientos antes de la horquilla de replicación.
El replisoma es responsable de copiar todo el ADN genómico en cada célula proliferativa. Las reacciones de apareamiento de bases y formación de cadena, que forman la hélice hija, son catalizadas por las ADN polimerasas. [88] Estas enzimas se mueven a lo largo del ADN monocatenario y permiten la extensión de la hebra de ADN naciente "leyendo" la hebra molde y permitiendo la incorporación de las nucleobases de purina adecuadas , adenina y guanina , y nucleobases de pirimidina , timina y citosina . Los desoxirribonucleótidos libres activados existen en la célula como desoxirribonucleótidos trifosfatos (dNTP). Estos nucleótidos libres se agregan a un grupo 3'-hidroxilo expuesto en el último nucleótido incorporado. En esta reacción, se libera un pirofosfato del dNTP libre, generando energía para la reacción de polimerización y exponiendo el monofosfato 5', que luego se une covalentemente al oxígeno 3'. Además, los nucleótidos insertados incorrectamente pueden eliminarse y reemplazarse por los nucleótidos correctos en una reacción energéticamente favorable. Esta propiedad es vital para la corrección y reparación adecuadas de los errores que ocurren durante la replicación del ADN.
La horquilla de replicación es la unión entre las cadenas molde recién separadas, conocidas como las cadenas líder y rezagada, y el ADN bicatenario. Dado que el ADN dúplex es antiparalelo, la replicación del ADN se produce en direcciones opuestas entre las dos nuevas cadenas en la horquilla de replicación, pero todas las ADN polimerasas sintetizan ADN en la dirección 5' a 3' con respecto a la cadena recién sintetizada. Se requiere una mayor coordinación durante la replicación del ADN. Dos polimerasas replicativas sintetizan ADN en orientaciones opuestas. La polimerasa ε sintetiza ADN en la cadena de ADN "líder" de forma continua, ya que apunta en la misma dirección que el ADN que se desenrolla por el replisoma. Por el contrario, la polimerasa δ sintetiza ADN en la cadena "rezagada", que es la cadena molde de ADN opuesta, de forma fragmentada o discontinua.
Los tramos discontinuos de productos de replicación de ADN en la cadena rezagada se conocen como fragmentos de Okazaki y tienen una longitud de entre 100 y 200 bases en las horquillas de replicación eucariotas. La cadena rezagada suele contener tramos más largos de ADN monocatenario recubierto de proteínas de unión monocatenarias, que ayudan a estabilizar las plantillas monocatenarias al evitar la formación de una estructura secundaria. En los eucariotas, estas proteínas de unión monocatenarias son un complejo heterotrimérico conocido como proteína de replicación A (RPA). [89]
Cada fragmento de Okazaki está precedido por un cebador de ARN, que es desplazado por la procesión del siguiente fragmento de Okazaki durante la síntesis. La ARNasa H reconoce los híbridos de ADN: ARN que se crean mediante el uso de cebadores de ARN y es responsable de eliminarlos de la cadena replicada, dejando atrás una unión cebador: molde. La ADN polimerasa α reconoce estos sitios y alarga las roturas dejadas por la eliminación del cebador. En las células eucariotas, una pequeña cantidad del segmento de ADN inmediatamente aguas arriba del cebador de ARN también se desplaza, creando una estructura de colgajo. Luego, este colgajo es escindido por endonucleasas. En la horquilla de replicación, el hueco en el ADN después de la eliminación del colgajo es sellado por la ADN ligasa I , que repara las muescas que quedan entre el 3'-OH y el 5'fosfato de la cadena recién sintetizada. [90] Debido a la naturaleza relativamente corta del fragmento de Okazaki eucariota, la síntesis de replicación de ADN que ocurre de manera discontinua en la cadena rezagada es menos eficiente y requiere más tiempo que la síntesis de la cadena líder. La síntesis de ADN se completa una vez que se eliminan todos los cebadores de ARN y se reparan las mellas.

Durante la replicación del ADN, el replisoma desenrollará el ADN dúplex parental en una horquilla de replicación de plantilla de ADN de dos cadenas simples en una dirección de 5' a 3'. La cadena líder es la cadena de plantilla que se está replicando en la misma dirección que el movimiento de la horquilla de replicación. Esto permite que la cadena recién sintetizada complementaria a la cadena original se sintetice de 5' a 3' en la misma dirección que el movimiento de la horquilla de replicación. [91]
Una vez que una primasa añade un cebador de ARN al extremo 3' de la cadena principal, la síntesis de ADN continuará en una dirección 3' a 5' con respecto a la cadena principal sin interrupciones. La ADN polimerasa ε añadirá continuamente nucleótidos a la cadena molde, por lo que la síntesis de la cadena principal requiere solo un cebador y tiene una actividad de ADN polimerasa ininterrumpida. [92]
La replicación del ADN en la hebra retrasada es discontinua. En la síntesis de la hebra retrasada, el movimiento de la ADN polimerasa en la dirección opuesta a la de la horquilla de replicación requiere el uso de múltiples cebadores de ARN . La ADN polimerasa sintetizará fragmentos cortos de ADN llamados fragmentos de Okazaki que se agregan al extremo 3' del cebador. Estos fragmentos pueden tener entre 100 y 400 nucleótidos de longitud en eucariotas. [93]
Al final de la síntesis del fragmento de Okazaki, la ADN polimerasa δ se topa con el fragmento de Okazaki anterior y desplaza su extremo 5' que contiene el cebador de ARN y un pequeño segmento de ADN. Esto genera un colgajo de cadena sencilla de ARN-ADN, que debe escindirse, y la muesca entre los dos fragmentos de Okazaki debe sellarse mediante la ADN ligasa I. Este proceso se conoce como maduración del fragmento de Okazaki y se puede manejar de dos maneras: un mecanismo procesa colgajos cortos, mientras que el otro se ocupa de los colgajos largos. [94] La ADN polimerasa δ puede desplazar hasta 2 a 3 nucleótidos de ADN o ARN antes de su polimerización, generando un sustrato de "colgajo" corto para Fen1 , que puede eliminar nucleótidos del colgajo, un nucleótido a la vez.
Al repetir ciclos de este proceso, la ADN polimerasa δ y Fen1 pueden coordinar la eliminación de cebadores de ARN y dejar una muesca de ADN en la hebra rezagada. [95] Se ha propuesto que este proceso iterativo es preferible para la célula porque está estrechamente regulado y no genera colgajos grandes que necesitan ser extirpados. [96] En el caso de una actividad desregulada de Fen1/ADN polimerasa δ, la célula utiliza un mecanismo alternativo para generar y procesar colgajos largos utilizando Dna2, que tiene actividades tanto de helicasa como de nucleasa. [97] La actividad de nucleasa de Dna2 es necesaria para eliminar estos colgajos largos, dejando un colgajo más corto para ser procesado por Fen1. Los estudios de microscopía electrónica indican que la carga de nucleosomas en la hebra rezagada ocurre muy cerca del sitio de síntesis. [98] Por lo tanto, la maduración del fragmento de Okazaki es un proceso eficiente que ocurre inmediatamente después de que se sintetiza el ADN naciente.
Después de que la helicasa replicativa ha desenrollado el dúplex de ADN parental, exponiendo dos plantillas de ADN monocatenario, se necesitan polimerasas replicativas para generar dos copias del genoma parental. La función de la ADN polimerasa es altamente especializada y logra la replicación en plantillas específicas y en localizaciones estrechas. En la horquilla de replicación eucariota, hay tres complejos de polimerasas replicativas distintos que contribuyen a la replicación del ADN: polimerasa α, polimerasa δ y polimerasa ε. Estas tres polimerasas son esenciales para la viabilidad de la célula. [99]
Debido a que las ADN polimerasas requieren un cebador en el que comenzar la síntesis de ADN, la polimerasa α (Pol α) actúa como una primasa replicativa. La Pol α está asociada con una primasa de ARN y este complejo cumple la tarea de cebado sintetizando un cebador que contiene un tramo corto de 10 nucleótidos de ARN seguido de 10 a 20 bases de ADN. [3] Es importante destacar que esta acción de cebado ocurre al inicio de la replicación en los orígenes para comenzar la síntesis de la cadena líder y también en el extremo 5' de cada fragmento de Okazaki en la cadena rezagada.
Sin embargo, la Pol α no puede continuar la replicación del ADN y debe ser reemplazada por otra polimerasa para continuar la síntesis del ADN. [100] El cambio de polimerasa requiere cargadores de pinza y se ha demostrado que la replicación normal del ADN requiere las acciones coordinadas de las tres polimerasas del ADN: Pol α para la síntesis de cebado, Pol ε para la replicación de la cadena líder y Pol δ, que está constantemente cargada, para generar fragmentos de Okazaki durante la síntesis de la cadena rezagada. [101]
Las helicasas y polimerasas del ADN deben permanecer en estrecho contacto en la horquilla de replicación. Si el desenrollado se produce demasiado antes de la síntesis, se exponen grandes extensiones de ADN monocatenario. Esto puede activar la señalización de daños en el ADN o inducir procesos de reparación del ADN. Para evitar estos problemas, el replisoma eucariota contiene proteínas especializadas que están diseñadas para regular la actividad de la helicasa antes de la horquilla de replicación. Estas proteínas también proporcionan sitios de acoplamiento para la interacción física entre las helicasas y las polimerasas, lo que garantiza que el desenrollado del dúplex esté acoplado con la síntesis de ADN. [105]
Para que las ADN polimerasas funcionen, la hélice de ADN de doble cadena debe desenrollarse para exponer dos plantillas de ADN de cadena sencilla para la replicación. Las helicasas de ADN son responsables de desenrollar el ADN de doble cadena durante la replicación cromosómica. Las helicasas en células eucariotas son notablemente complejas. [106] El núcleo catalítico de la helicasa está compuesto por seis proteínas de mantenimiento de minicromosomas (Mcm2-7), que forman un anillo hexamérico . Lejos del ADN, las proteínas Mcm2-7 forman un único heterohexámero y se cargan en una forma inactiva en los orígenes de replicación del ADN como hexámeros dobles cabeza con cabeza alrededor del ADN de doble cadena. [106] [107] Las proteínas Mcm se reclutan a los orígenes de replicación y luego se redistribuyen por todo el ADN genómico durante la fase S, lo que indica su localización en la horquilla de replicación. [54]
La carga de las proteínas Mcm solo puede ocurrir durante la fase G 1 del ciclo celular, y el complejo cargado se activa luego durante la fase S mediante el reclutamiento de la proteína Cdc45 y el complejo GINS para formar la helicasa activa Cdc45–Mcm–GINS (CMG) en las horquillas de replicación del ADN. [62] [108] La actividad de Mcm es necesaria durante toda la fase S para la replicación del ADN. [38] [109] Una variedad de factores reguladores se reúnen alrededor de la helicasa CMG para producir el "complejo de progresión del replisoma" que se asocia con las polimerasas del ADN para formar el replisoma eucariota, cuya estructura aún está bastante mal definida en comparación con su contraparte bacteriana. [59] [110]
La helicasa CMG aislada y el complejo de progresión del replisoma contienen un único complejo de anillo de proteína Mcm, lo que sugiere que el doble hexámero cargado de las proteínas Mcm en los orígenes podría romperse en dos anillos hexaméricos individuales como parte del proceso de iniciación, y cada anillo del complejo de proteína Mcm forma el núcleo de una helicasa CMG en las dos horquillas de replicación establecidas a partir de cada origen. [59] [62] El complejo CMG completo es necesario para el desenrollado del ADN, y el complejo de CDC45-Mcm-GINS es la helicasa de ADN funcional en las células eucariotas. [64]
El complejo CMG interactúa con el replisoma a través de la interacción con las proteínas Ctf4 y And1. Las proteínas Ctf4/And1 interactúan tanto con el complejo CMG como con la ADN polimerasa α. [111] Ctf4 es un factor accesorio de la polimerasa α, que es necesario para el reclutamiento de la polimerasa α a los orígenes de replicación. [112]
Las proteínas Mrc1/Claspin acoplan la síntesis de la cadena líder con la actividad de la helicasa del complejo CMG. Mrc1 interactúa con la polimerasa ε así como con las proteínas Mcm. [113] La importancia de este vínculo directo entre la helicasa y la polimerasa de la cadena líder se ve subrayada por los resultados en células humanas cultivadas, donde Mrc1/Claspin es necesaria para la progresión eficiente de la horquilla de replicación. [114] Estos resultados sugieren que la replicación eficiente del ADN también requiere el acoplamiento de las helicasas y la síntesis de la cadena líder...
Las ADN polimerasas requieren factores adicionales para apoyar la replicación del ADN. Las ADN polimerasas tienen una estructura semicerrada en forma de "mano", que permite que la polimerasa se cargue en el ADN y comience la translocación. Esta estructura permite que la ADN polimerasa sostenga la plantilla de ADN monocatenario, incorpore dNTP en el sitio activo y libere el ADN bicatenario recién formado. Sin embargo, la estructura de las ADN polimerasas no permite una interacción estable continua con la plantilla de ADN. [1]
Para fortalecer la interacción entre la polimerasa y el ADN molde, las abrazaderas deslizantes de ADN se asocian con la polimerasa para promover la procesividad de la polimerasa replicativa. En eucariotas, la abrazadera deslizante es una estructura de anillo de homotrímero conocida como antígeno nuclear de células proliferantes (PCNA). El anillo de PCNA tiene polaridad con las superficies que interactúan con las ADN polimerasas y las une de forma segura al ADN molde. La estabilización dependiente de PCNA de las ADN polimerasas tiene un efecto significativo en la replicación del ADN porque los PCNA pueden mejorar la procesividad de la polimerasa hasta 1000 veces. [115] [116] El PCNA es un cofactor esencial y tiene la distinción de ser una de las plataformas de interacción más comunes en el replisoma para acomodar múltiples procesos en la horquilla de replicación, y por eso el PCNA también se considera un cofactor regulador para las ADN polimerasas. [117]
El PCNA rodea por completo la cadena de ADN molde y debe cargarse en el ADN en la horquilla de replicación. En la cadena líder, la carga del PCNA es un proceso poco frecuente, porque la replicación del ADN en la cadena líder es continua hasta que la replicación termina. Sin embargo, en la cadena rezagada, la ADN polimerasa δ debe cargarse continuamente al comienzo de cada fragmento de Okazaki. Esta iniciación constante de la síntesis del fragmento de Okazaki requiere la carga repetida de PCNA para una replicación eficiente del ADN.
La carga de PCNA se lleva a cabo mediante el complejo del factor de replicación C (RFC). El complejo RFC está compuesto por cinco ATPasas: Rfc1, Rfc2, Rfc3, Rfc4 y Rfc5. [118] RFC reconoce las uniones cebador-molde y carga PCNA en estos sitios. [119] [120] El homotrímero de PCNA es abierto por RFC mediante hidrólisis de ATP y luego se carga en el ADN en la orientación adecuada para facilitar su asociación con la polimerasa. [121] [122] Los cargadores de pinza también pueden descargar PCNA del ADN; un mecanismo necesario cuando se debe terminar la replicación. [122]
La replicación del ADN en la horquilla de replicación puede detenerse por una escasez de desoxinucleótidos trifosfatos (dNTP) o por daño del ADN, lo que resulta en estrés de replicación . [123] Esta detención de la replicación se describe como una horquilla de replicación estancada . Un complejo de protección de proteínas de la horquilla estabiliza la horquilla de replicación hasta que se puedan reparar el daño del ADN u otros problemas de replicación. [123] El estancamiento prolongado de la horquilla de replicación puede provocar más daño del ADN. Las señales de estancamiento se desactivan si se resuelven los problemas que causan la horquilla de replicación. [123]

La terminación de la replicación del ADN eucariota requiere diferentes procesos dependiendo de si los cromosomas son circulares o lineales. A diferencia de las moléculas lineales, los cromosomas circulares son capaces de replicar la molécula entera. Sin embargo, las dos moléculas de ADN permanecerán unidas. Este problema se soluciona mediante la descatenación de las dos moléculas de ADN por una topoisomerasa de tipo II . Las topoisomerasas de tipo II también se utilizan para separar las cadenas lineales a medida que se pliegan intrincadamente en un nucleosoma dentro de la célula.
Como se mencionó anteriormente, los cromosomas lineales enfrentan otro problema que no se ve en la replicación circular del ADN. Debido al hecho de que se requiere un cebador de ARN para el inicio de la síntesis de ADN, la hebra rezagada está en desventaja a la hora de replicar todo el cromosoma. Mientras que la hebra líder puede utilizar un único cebador de ARN para extender el extremo 5' de la hebra de ADN que se replica, varios cebadores de ARN son responsables de la síntesis de la hebra rezagada, creando fragmentos de Okazaki. Esto conduce a un problema debido al hecho de que la ADN polimerasa sólo puede añadir al extremo 3' de la hebra de ADN. La acción 3'-5' de la ADN polimerasa a lo largo de la hebra madre deja una región corta de ADN monocatenario (ssDNA) en el extremo 3' de la hebra madre cuando se han reparado los fragmentos de Okazaki. Dado que la replicación se produce en direcciones opuestas en los extremos opuestos de los cromosomas parentales, cada hebra es una hebra rezagada en un extremo. Con el tiempo, esto daría lugar a un acortamiento progresivo de ambos cromosomas hijos . Esto se conoce como el problema de replicación final. [1]
El problema de la replicación final es manejado en células eucariotas por regiones teloméricas y telomerasa . Los telómeros extienden el extremo 3' del cromosoma parental más allá del extremo 5' de la cadena hija. Esta estructura de ADN monocatenario puede actuar como un origen de replicación que recluta telomerasa. La telomerasa es una ADN polimerasa especializada que consta de múltiples subunidades proteicas y un componente de ARN. El componente de ARN de la telomerasa se une al extremo 3' monocatenario del ADN molde y contiene 1,5 copias de la secuencia telomérica. [93] La telomerasa contiene una subunidad proteica que es una transcriptasa inversa llamada transcriptasa inversa de la telomerasa o TERT. TERT sintetiza ADN hasta el final del ARN de la telomerasa molde y luego se desacopla. [93] Este proceso se puede repetir tantas veces como sea necesario con la extensión del extremo 3' de la molécula de ADN parental. Esta adición 3' proporciona una plantilla para la extensión del extremo 5' de la cadena hija mediante la síntesis de ADN de la cadena rezagada. La regulación de la actividad de la telomerasa está a cargo de las proteínas de unión a los telómeros.
La replicación del ADN procariota es bidireccional; dentro de un origen replicativo, se crean complejos de replisomas en cada extremo del origen de replicación y los replisomas se alejan entre sí desde el punto de partida inicial. En los procariotas, la replicación bidireccional se inicia en un origen replicativo en el cromosoma circular y termina en un sitio opuesto al inicio inicial del origen. [124] Estas regiones de terminación tienen secuencias de ADN conocidas como sitios Ter . Estos sitios Ter están unidos por la proteína Tus. El complejo Ter -Tus es capaz de detener la actividad de la helicasa, terminando la replicación. [125]
En las células eucariotas, la terminación de la replicación ocurre generalmente a través de la colisión de las dos horquillas replicativas entre dos orígenes de replicación activos. La ubicación de la colisión varía según el momento en que se activa el origen. De esta manera, si una horquilla de replicación se bloquea o colapsa en un sitio determinado, la replicación del sitio puede ser rescatada cuando un replisoma que viaja en la dirección opuesta completa la copia de la región. Existen barreras de la horquilla de replicación programadas (RFB) unidas por proteínas RFB en varias ubicaciones, a lo largo del genoma, que pueden terminar o pausar las horquillas de replicación, deteniendo la progresión del replisoma. [124]
Se ha descubierto que la replicación se produce de forma localizada en el núcleo celular. Contrariamente a la idea tradicional de que las horquillas de replicación se desplazan a lo largo del ADN estancado, surgió el concepto de fábricas de replicación , lo que significa que las horquillas de replicación se concentran en algunas regiones "factoriales" inmovilizadas por las que pasan las cadenas de ADN molde como si fueran cintas transportadoras. [126]
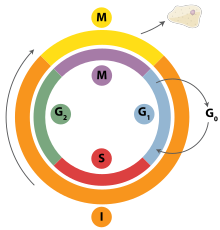
La replicación del ADN es un proceso estrechamente orquestado que se controla en el contexto del ciclo celular . El progreso a través del ciclo celular y, a su vez, la replicación del ADN están estrechamente regulados por la formación y activación de complejos prerreplicativos (pre-RC), lo que se logra mediante la activación e inactivación de las quinasas dependientes de ciclina (Cdks, CDK). En concreto, son las interacciones de las ciclinas y las quinasas dependientes de ciclina las responsables de la transición de la fase G 1 a la fase S.
Durante la fase G 1 del ciclo celular hay niveles bajos de actividad de CDK. Este bajo nivel de actividad de CDK permite la formación de nuevos complejos pre-RC pero no es suficiente para que la replicación del ADN sea iniciada por los pre-RC recién formados. Durante las fases restantes del ciclo celular hay niveles elevados de actividad de CDK. Este alto nivel de actividad de CDK es responsable de iniciar la replicación del ADN, así como de inhibir la formación de nuevos complejos pre-RC. [2] Una vez que se ha iniciado la replicación del ADN, el complejo pre-RC se descompone. Debido al hecho de que los niveles de CDK permanecen altos durante la fase S, G 2 y M del ciclo celular, no se pueden formar nuevos complejos pre-RC. Todo esto ayuda a garantizar que no pueda ocurrir ninguna iniciación hasta que se complete la división celular.
Además de las quinasas dependientes de ciclina, se cree que una nueva ronda de replicación se previene mediante la regulación negativa de Cdt1. Esto se logra mediante la degradación de Cdt1, así como mediante las acciones inhibidoras de una proteína conocida como geminina . La geminina se une firmemente a Cdt1 y se cree que es el principal inhibidor de la re-replicación. [2] La geminina aparece primero en la fase S y se degrada en la transición de metafase a anafase, posiblemente a través de la ubiquinación por el complejo promotor de anafase (APC). [127]
Existen varios puntos de control del ciclo celular a lo largo del ciclo celular que determinan si una célula progresará a través de la división por completo. Es importante destacar que en la replicación, el punto de control G 1 , o de restricción, determina si comenzará o no la replicación o si la célula pasará a una etapa de reposo conocida como G 0 . Las células en la etapa G 0 del ciclo celular no pueden iniciar una ronda de replicación porque no se expresan las proteínas de mantenimiento de los minicromosomas. La transición a la fase S indica que la replicación ha comenzado.
Para preservar la información genética durante la división celular, la replicación del ADN debe completarse con alta fidelidad. Para lograr esta tarea, las células eucariotas tienen proteínas en su lugar durante ciertos puntos del proceso de replicación que son capaces de detectar cualquier error durante la replicación del ADN y son capaces de preservar la integridad genómica. Estas proteínas de punto de control son capaces de detener el ciclo celular antes de entrar en mitosis para dar tiempo a la reparación del ADN. Las proteínas de punto de control también están involucradas en algunas vías de reparación del ADN, mientras que estabilizan la estructura de la horquilla de replicación para prevenir más daños. Estas proteínas de punto de control son esenciales para evitar la transmisión de mutaciones u otras aberraciones cromosómicas a la descendencia.
Las proteínas de punto de control eucariotas están bien conservadas e involucran a dos quinasas relacionadas con la fosfatidilinositol 3-quinasa (PIKK), ATR y ATM . Tanto ATR como ATM comparten una secuencia de fosforilación diana, el motivo SQ/TQ, pero sus funciones individuales en las células difieren. [128]
La ATR está involucrada en la detención del ciclo celular en respuesta a las roturas de la doble cadena de ADN. La ATR tiene un socio de punto de control obligado, la proteína que interactúa con la ATR (ATRIP), y juntas estas dos proteínas responden a tramos de ADN monocatenario que están recubiertos por la proteína de replicación A (RPA). [129] La formación de ADN monocatenario ocurre con frecuencia, más a menudo durante el estrés de replicación. La ATR-ATRIP es capaz de detener el ciclo celular para preservar la integridad del genoma. La ATR se encuentra en la cromatina durante la fase S, de manera similar a la RPA y la claspina. [130]
La generación de tractos de ADN monocatenario es importante para iniciar las vías de puntos de control aguas abajo del daño de replicación. Una vez que el ADN monocatenario se vuelve lo suficientemente largo, el ADN monocatenario recubierto con RPA puede reclutar ATR-ATRIP. [131] Para volverse completamente activa, la quinasa ATR depende de proteínas sensoras que detectan si las proteínas de puntos de control están localizadas en un sitio válido de estrés de replicación de ADN. La pinza heterotrimérica RAD9 - HUS1 - Rad1 (9-1-1) y su cargador de pinza RFC Rad17 pueden reconocer ADN con huecos o mellas. El cargador de pinza RFC Rad17 carga 9-1-1 en el ADN dañado. [132] La presencia de 9-1-1 en el ADN es suficiente para facilitar la interacción entre ATR-ATRIP y un grupo de proteínas denominadas mediadores de puntos de control, como TOPBP1 y Mrc1/ claspin . TOPBP1 interactúa con el componente fosforilado Rad9 de 9-1-1 y lo recluta, y se une a ATR-ATRIP, que fosforila Chk1. [133] Mrc1/Claspin también es necesario para la activación completa de ATR-ATRIP que fosforila Chk1, la principal quinasa efectora del punto de control descendente. [134] Claspin es un componente del replisoma y contiene un dominio para acoplarse con Chk1, lo que revela una función específica de Claspin durante la replicación del ADN: la promoción de la señalización del punto de control en el replisoma. [135]
La señalización de Chk1 es vital para detener el ciclo celular y evitar que las células entren en mitosis con una replicación de ADN incompleta o con daño en el ADN. La inhibición de Cdk dependiente de Chk1 es importante para la función del punto de control ATR-Chk1 y para detener el ciclo celular y permitir el tiempo suficiente para la finalización de los mecanismos de reparación del ADN, lo que a su vez evita la herencia del ADN dañado. Además, la inhibición de Cdk dependiente de Chk1 desempeña un papel fundamental en la inhibición de la activación del origen durante la fase S. Este mecanismo impide la síntesis continua de ADN y es necesario para la protección del genoma en presencia de estrés de replicación y posibles condiciones genotóxicas. [136] Por lo tanto, la actividad de ATR-Chk1 previene además posibles problemas de replicación a nivel de orígenes de replicación individuales al inhibir el inicio de la replicación en todo el genoma, hasta que se desactiva la cascada de señalización que mantiene la detención del ciclo celular.

El ADN eucariota debe estar muy compactado para caber en el espacio confinado del núcleo. Los cromosomas se empaquetan envolviendo 147 nucleótidos alrededor de un octámero de proteínas histonas , formando un nucleosoma . El octámero del nucleosoma incluye dos copias de cada histona H2A , H2B , H3 y H4 . Debido a la estrecha asociación de las proteínas histonas con el ADN, las células eucariotas tienen proteínas que están diseñadas para remodelar las histonas por delante de la horquilla de replicación, con el fin de permitir una progresión suave del replisoma. [137] También hay proteínas involucradas en el reensamblaje de las histonas detrás de la horquilla de replicación para restablecer la conformación del nucleosoma. [138]
Existen varias chaperonas de histonas que se sabe que participan en el ensamblaje de nucleosomas después de la replicación. Se ha descubierto que el complejo FACT interactúa con el complejo α-primasa de la ADN polimerasa, y las subunidades del complejo FACT interactúan genéticamente con factores de replicación. [139] [140] El complejo FACT es un heterodímero que no hidroliza ATP, pero es capaz de facilitar el "aflojamiento" de las histonas en los nucleosomas, pero aún no se sabe cómo el complejo FACT es capaz de aliviar la estrecha asociación de las histonas para la eliminación del ADN. [141]
Otra chaperona de histonas que se asocia con el replisoma es Asf1 , que interactúa con el complejo Mcm dependiente de los dímeros de histonas H3-H4. [142] Asf1 puede pasar el dímero H3-H4 recién sintetizado a los factores de deposición detrás de la horquilla de replicación y esta actividad hace que los dímeros de histonas H3-H4 estén disponibles en el sitio de deposición de histonas justo después de la replicación. [143] Asf1 (y su socio Rtt109) también se han implicado en la inhibición de la expresión génica de los genes replicados durante la fase S. [144]
El factor de ensamblaje de cromatina 1 ( CAF-1 ) chaperona heterotrimérica es una proteína de formación de cromatina que participa en el depósito de histonas en ambas cadenas de ADN recién replicadas para formar cromatina. [145] CAF-1 contiene un motivo de unión a PCNA, llamado PIP-box, que permite que CAF-1 se asocie con el replisoma a través de PCNA y pueda depositar dímeros de histona H3-H4 en el ADN recién sintetizado. [146] [147] La chaperona Rtt106 también participa en este proceso y se asocia con CAF-1 y dímeros H3-H4 durante la formación de la cromatina. [148] Estos procesos cargan histonas recién sintetizadas en el ADN.
Tras la deposición de las histonas H3-H4, los nucleosomas se forman por la asociación de la histona H2A-H2B. Se piensa que este proceso ocurre a través del complejo FACT, ya que este se asoció al replisoma y es capaz de unirse a la H2A-H2B libre, o existe la posibilidad de otra chaperona H2A-H2B, Nap1. [149] Estudios de microscopía electrónica muestran que esto ocurre muy rápidamente, ya que se pueden observar nucleosomas formando solo unos pocos cientos de pares de bases después de la horquilla de replicación. [150] Por lo tanto, todo el proceso de formación de nuevos nucleosomas ocurre justo después de la replicación debido al acoplamiento de las chaperonas de histonas al replisoma.
En comparación con la replicación del ADN procariota , concretamente en las bacterias, la finalización de la replicación del ADN eucariota es más compleja e implica múltiples orígenes de replicación y proteínas replicativas para lograrlo. El ADN procariota está dispuesto en forma circular y solo tiene un origen de replicación cuando comienza la replicación. Por el contrario, el ADN eucariota es lineal. Cuando se replica, hay hasta mil orígenes de replicación. [151]
El ADN eucariota es bidireccional. En este caso, el significado de la palabra bidireccional es diferente. El ADN eucariota lineal tiene muchos orígenes (llamados O) y extremos (llamados T). La "T" está presente a la derecha de la "O". Una "O" y una "T" juntas forman un replicón. Después de la formación del complejo de preiniciación, cuando un replicón comienza a elongarse, la iniciación comienza en el segundo replicón. Ahora bien, si el primer replicón se mueve en el sentido de las agujas del reloj, el segundo replicón se mueve en el sentido contrario a las agujas del reloj, hasta que se alcanza la "T" del primer replicón. En la "T", ambos replicones se fusionan para completar el proceso de replicación. Mientras tanto, el segundo replicón también se mueve hacia adelante, para encontrarse con el tercer replicón. Este movimiento en el sentido de las agujas del reloj y en el sentido contrario a las agujas del reloj de dos replicones se denomina replicación bidireccional.
La replicación del ADN eucariota requiere una coordinación precisa de todas las polimerasas del ADN y las proteínas asociadas para replicar el genoma completo cada vez que una célula se divide. Este proceso se logra a través de una serie de pasos de ensamblajes de proteínas en los orígenes de replicación, centrándose principalmente la regulación de la replicación del ADN en la asociación de la helicasa MCM con el ADN. Estos orígenes de replicación dirigen el número de complejos proteicos que se formarán para iniciar la replicación. En la replicación del ADN bacteriano, la regulación se centra en la unión de la proteína iniciadora DnaA al ADN, y el inicio de la replicación ocurre varias veces durante un ciclo celular. [93] Tanto el ADN procariota como el eucariota utilizan la unión de ATP y la hidrólisis para dirigir la carga de la helicasa y, en ambos casos, la helicasa se carga en la forma inactiva. Sin embargo, las helicasas eucariotas son hexámeros dobles que se cargan en el ADN bicatenario, mientras que las helicasas bacterianas son hexámeros simples cargados en el ADN monocatenario. [152]
La segregación de cromosomas es otra diferencia entre las células procariotas y eucariotas. Las células que se dividen rápidamente, como las bacterias, a menudo comienzan a segregar cromosomas que todavía están en proceso de replicación. En las células eucariotas, la segregación de cromosomas en las células hijas no se inicia hasta que se completa la replicación en todos los cromosomas. [93] Sin embargo, a pesar de estas diferencias, el proceso subyacente de replicación es similar tanto para el ADN procariota como para el eucariota.
Lista de las principales proteínas implicadas en la replicación del ADN eucariota: