Las ferredoxinas (del latín ferrum : hierro + redox , a menudo abreviado como "fd") son proteínas de hierro y azufre que median la transferencia de electrones en una variedad de reacciones metabólicas. El término "ferredoxina" fue acuñado por DC Wharton de DuPont Co. y aplicado a la "proteína de hierro" purificada por primera vez en 1962 por Mortenson, Valentine y Carnahan a partir de la bacteria anaeróbica Clostridium pasteurianum . [1] [2]
Otra proteína redox, aislada de los cloroplastos de espinaca , se denominó "ferredoxina del cloroplasto". [3] La ferredoxina del cloroplasto participa en las reacciones de fotofosforilación cíclica y no cíclica de la fotosíntesis . En la fotofosforilación no cíclica, la ferredoxina es el último aceptor de electrones, reduciendo así la enzima NADP + reductasa. Acepta electrones producidos a partir de la clorofila excitada por la luz solar y los transfiere a la enzima ferredoxina: NADP + oxidorreductasa EC 1.18.1.2.
Las ferredoxinas son pequeñas proteínas que contienen átomos de hierro y azufre organizados en grupos hierro-azufre . Estos “ condensadores ” biológicos pueden aceptar o descargar electrones, con el efecto de un cambio en el estado de oxidación de los átomos de hierro entre +2 y +3. De esta manera, la ferredoxina actúa como un agente de transferencia de electrones en reacciones biológicas redox .
Otros sistemas de transporte de electrones bioinorgánicos incluyen rubredoxinas , citocromos , proteínas azules de cobre y las proteínas Rieske estructuralmente relacionadas .
Las ferredoxinas se pueden clasificar según la naturaleza de sus grupos de hierro-azufre y por la similitud de secuencia.
Las ferredoxinas normalmente realizan una única transferencia de electrones.
Sin embargo, algunas ferredoxinas bacterianas (del tipo 2[4Fe4S]) tienen dos grupos de azufre y hierro y pueden llevar a cabo dos reacciones de transferencia de electrones. Dependiendo de la secuencia de la proteína, las dos transferencias pueden tener potenciales de reducción casi idénticos o pueden ser significativamente diferentes. [4] [5]
Las ferredoxinas son uno de los transportadores de electrones biológicos más reductores. Por lo general, tienen un potencial de punto medio de -420 mV. [6] El potencial de reducción de una sustancia en la célula diferirá de su potencial de punto medio dependiendo de las concentraciones de sus formas reducidas y oxidadas. Para una reacción de un electrón, el potencial cambia alrededor de 60 mV por cada cambio de potencia de diez en la relación de la concentración. Por ejemplo, si el grupo de ferredoxinas está reducido en alrededor del 95%, el potencial de reducción será de alrededor de -500 mV. [7] En comparación, otras reacciones biológicas en su mayoría tienen potenciales reductores menos: por ejemplo, el reductor biosintético primario de la célula, NADPH tiene un potencial redox celular de -370 mV ( E
0= -320mV).
Dependiendo de la secuencia de la proteína de soporte, las ferredoxinas tienen un potencial de reducción de alrededor de -500 mV [6] [8] a -340 mV. [9] Una sola célula puede tener múltiples tipos de ferredoxinas, donde cada tipo está ajustado para llevar a cabo de manera óptima diferentes reacciones. [10]
Las ferredoxinas altamente reductoras se reducen ya sea utilizando otro agente reductor fuerte o utilizando alguna fuente de energía para "impulsar" electrones desde fuentes menos reductoras hacia la ferredoxina. [11]
Las reacciones que reducen el Fd incluyen la oxidación de aldehídos a ácidos como la reacción de gliceraldehído a glicerato (-580 mV), la reacción de la monóxido de carbono deshidrogenasa (-520 mV) y las reacciones de 2-oxoácido:Fd oxidorreductasa (-500 mV) [12] [8] como la reacción llevada a cabo por la piruvato sintasa . [7]
La ferredoxina también se puede reducir utilizando NADH (-320 mV) o H
2(-414 mV), pero estos procesos están acoplados al consumo del potencial de membrana para impulsar el "impulso" de los electrones al estado de mayor energía. [6] El complejo Rnf es una proteína de membrana muy extendida en las bacterias que transfiere electrones de forma reversible entre NADH y ferredoxina mientras bombea Na+
o H+
iones a través de la membrana celular . El potencial quimiosmótico de la membrana se consume para impulsar la reducción desfavorable de Fd
bueypor NADH. Esta reacción es una fuente esencial de Fd-
rojoEn muchos organismos autótrofos , si la célula crece en sustratos que aportan Fd en exceso-
rojoEl complejo Rnf puede transferir estos electrones al NAD+
y almacenan la energía resultante en el potencial de membrana. [13] Las hidrogenasas convertidoras de energía (Ech) son una familia de enzimas que acoplan reversiblemente la transferencia de electrones entre Fd y H
2mientras bombea H+
iones a través de la membrana para equilibrar la diferencia de energía. [14]
La reducción desfavorable de Fd a partir de un donador de electrones menos reductor puede acoplarse simultáneamente con la reducción favorable de un agente oxidante a través de una reacción de bifurcación de electrones . [6] Un ejemplo de la reacción de bifurcación de electrones es la generación de Fd-
rojopara la fijación de nitrógeno en ciertos diazótrofos aeróbicos . Normalmente, en la fosforilación oxidativa, la transferencia de electrones de NADH a ubiquinona (Q) está acoplada a la carga de la fuerza motriz del protón. En Azotobacter, la energía liberada al transferir un electrón de NADH a Q se utiliza para impulsar simultáneamente la transferencia de un electrón de NADH a Fd. [15] [16]
Algunas ferredoxinas tienen un potencial redox suficientemente alto como para que puedan ser reducidas directamente por el NADPH. Una de estas ferredoxinas es la adrenoxina (-274 mV), que participa en la biosíntesis de muchos esteroides de mamíferos . [17] La ferredoxina Fd3 presente en las raíces de las plantas, que reduce el nitrato y el sulfito, tiene un potencial medio de -337 mV y también es reducida por el NADPH. [10]
Los miembros de la superfamilia ferredoxina 2Fe–2S ( InterPro : IPR036010 ) tienen una estructura central general que consiste en beta(2)-alfa-beta(2), que incluye putidaredoxina, terpredoxina y adrenodoxina. [18] [19] [20] [21] Son proteínas de alrededor de cien aminoácidos con cuatro residuos de cisteína conservados a los que se liga el grupo 2Fe–2S. Esta región conservada también se encuentra como un dominio en varias enzimas metabólicas y en proteínas multidominio, como la aldehído oxidorreductasa ( N -terminal), la xantina oxidasa ( N -terminal), la ftalato dioxigenasa reductasa ( C -terminal), la succinato deshidrogenasa proteína hierro-azufre ( N -terminal) y la metano monooxigenasa reductasa ( N -terminal).
Un grupo de ferredoxinas, originalmente encontradas en las membranas de los cloroplastos , ha sido denominado "tipo cloroplasto" o "tipo planta" ( InterPro : IPR010241 ). Su centro activo es un grupo [Fe2S2 ] , donde los átomos de hierro están coordinados tetraédricamente tanto por átomos de azufre inorgánico como por azufres de cuatro residuos de cisteína (Cys) conservados.
En los cloroplastos, las ferredoxinas Fe2S2 funcionan como transportadores de electrones en la cadena de transporte de electrones fotosintéticos y como donantes de electrones a varias proteínas celulares, como la glutamato sintasa, la nitrito reductasa, la sulfito reductasa y la ciclasa de la biosíntesis de clorofila . [22] Dado que la ciclasa es una enzima dependiente de la ferredoxina, esto puede proporcionar un mecanismo de coordinación entre la fotosíntesis y la necesidad de clorofila de los cloroplastos al vincular la biosíntesis de clorofila a la cadena de transporte de electrones fotosintéticos. En los sistemas de dioxigenasa bacteriana hidroxilantes, sirven como transportadores de transferencia de electrones intermedios entre las flavoproteínas reductasas y la oxigenasa.
La ferredoxina Fe 2 S 2 de Clostridium pasteurianum ( Cp 2FeFd; P07324 ) ha sido reconocida como una familia de proteínas distinta sobre la base de su secuencia de aminoácidos, propiedades espectroscópicas de su grupo hierro-azufre y la capacidad única de intercambio de ligandos de dos ligandos de cisteína al grupo [Fe 2 S 2 ]. Aunque el papel fisiológico de esta ferredoxina sigue sin estar claro, se ha revelado una interacción fuerte y específica de Cp 2FeFd con la proteína molibdeno-hierro de la nitrogenasa . Se han caracterizado ferredoxinas homólogas de Azotobacter vinelandii ( Av 2FeFdI; P82802 ) y Aquifex aeolicus ( Aa Fd; O66511 ). Se ha resuelto la estructura cristalina de Aa Fd. Aa Fd existe como un dímero. La estructura del monómero Aa Fd es diferente a la de otras ferredoxinas Fe2S2 . El pliegue pertenece a la clase α+β, con las primeras cuatro cadenas β y dos hélices α que adoptan una variante del pliegue de la tiorredoxina . [23] UniProt las clasifica como la familia de las "ferredoxinas de tipo 2Fe2S Shethna". [24]
La adrenodoxina (ferredoxina suprarrenal; InterPro : IPR001055 ), la putidaredoxina y la terpredoxina forman una familia de proteínas solubles Fe2S2 que actúan como transportadores de un solo electrón y que se encuentran principalmente en mitocondrias eucariotas y Pseudomonadota . La variante humana de la adrenodoxina se conoce como ferredoxina-1 y ferredoxina-2 . En los sistemas de monooxigenasa mitocondrial, la adrenodoxina transfiere un electrón de la NADPH:adrenodoxina reductasa al citocromo P450 unido a la membrana . En las bacterias, la putidaredoxina y la terpredoxina transfieren electrones entre las correspondientes ferredoxinas reductasas dependientes de NADH y las P450 solubles. [26] [27] No se conocen las funciones exactas de otros miembros de esta familia, aunque se ha demostrado que Escherichia coli Fdx está implicada en la biogénesis de los grupos Fe–S. [28] A pesar de la baja similitud de secuencia entre las ferredoxinas de tipo adrenodoxina y de tipo vegetal, las dos clases tienen una topología de plegamiento similar.
La ferredoxina-1 en humanos participa en la síntesis de hormonas tiroideas. También transfiere electrones de la adrenodoxina reductasa a la CYP11A1 , una enzima CYP450 responsable de la escisión de la cadena lateral del colesterol. La FDX-1 tiene la capacidad de unirse a metales y proteínas. [29] La ferredoxina-2 participa en la síntesis de hemo A y de proteínas de hierro y azufre. [30]
Las ferredoxinas [Fe 4 S 4 ] pueden subdividirse en ferredoxinas de bajo potencial (tipo bacteriano) y de alto potencial (HiPIP) .
Las ferredoxinas de alto y bajo potencial están relacionadas mediante el siguiente esquema redox:
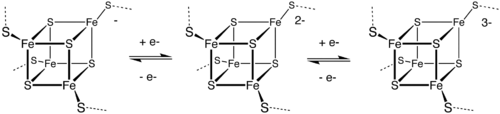
Los números de oxidación formales de los iones de hierro pueden ser [2Fe 3+ , 2Fe 2+ ] o [1Fe 3+ , 3Fe 2+ ] en ferredoxinas de bajo potencial. Los números de oxidación de los iones de hierro en ferredoxinas de alto potencial pueden ser [3Fe 3+ , 1Fe 2+ ] o [2Fe 3+ , 2Fe 2+ ].
Un grupo de ferredoxinas Fe 4 S 4 , originalmente encontradas en bacterias, se ha denominado "de tipo bacteriano". Las ferredoxinas de tipo bacteriano pueden a su vez subdividirse en otros grupos, en función de sus propiedades de secuencia. La mayoría contiene al menos un dominio conservado, incluidos cuatro residuos de cisteína que se unen a un grupo [Fe 4 S 4 ]. En la ferredoxina Fe 4 S 4 de Pyrococcus furiosus , uno de los residuos de Cys conservados está sustituido por ácido aspártico.
Durante la evolución de las ferredoxinas de tipo bacteriano, se produjeron eventos de duplicación, transposición y fusión de genes intrasecuenciales, lo que dio como resultado la aparición de proteínas con múltiples centros de hierro-azufre. En algunas ferredoxinas bacterianas, uno de los dominios duplicados ha perdido uno o más de los cuatro residuos de Cys conservados. Estos dominios han perdido su propiedad de unión de hierro-azufre o se unen a un grupo [Fe 3 S 4 ] en lugar de un grupo [Fe 4 S 4 ] [31] y son de tipo dicluster. [32]
Se conocen estructuras tridimensionales para varias ferredoxinas bacterianas monoagrupadas y diagrupadas. El pliegue pertenece a la clase α+β, con 2-7 hélices α y cuatro cadenas β que forman una estructura en forma de barril, y un bucle extruido que contiene tres ligandos Cys "proximales" del grupo hierro-azufre.
Las proteínas de hierro-azufre de alto potencial (HiPIP) forman una familia única de ferredoxinas Fe 4 S 4 que funcionan en cadenas de transporte de electrones anaeróbicas. Algunas HiPIP tienen un potencial redox más alto que cualquier otra proteína de hierro-azufre conocida (por ejemplo, la HiPIP de Rhodopila globiformis tiene un potencial redox de aproximadamente -450 mV). Hasta ahora se han caracterizado estructuralmente varias HiPIP, cuyos pliegues pertenecen a la clase α+β. Al igual que en otras ferredoxinas bacterianas, la unidad [Fe 4 S 4 ] forma un grupo de tipo cubano y está ligada a la proteína a través de cuatro residuos de Cys.