La clasificación de virus es el proceso de nombrar a los virus y colocarlos en un sistema taxonómico similar a los sistemas de clasificación utilizados para los organismos celulares .
Los virus se clasifican según características fenotípicas , como la morfología , el tipo de ácido nucleico , el modo de replicación, los organismos hospedadores y el tipo de enfermedad que causan. La clasificación taxonómica formal de los virus es responsabilidad del sistema del Comité Internacional de Taxonomía de Virus (ICTV), aunque el sistema de clasificación de Baltimore puede utilizarse para colocar a los virus en uno de siete grupos según su forma de síntesis de ARNm. El ICTV establece convenciones de nomenclatura específicas y otras pautas de clasificación.
En 2021, el ICTV modificó el Código internacional de clasificación y nomenclatura de virus (ICVCN) para exigir un formato binomial (género|| ||especie) para nombrar nuevas especies virales similar al utilizado para los organismos celulares; los nombres de las especies acuñados antes de 2021 se están convirtiendo gradualmente al nuevo formato, un proceso cuya finalización está prevista para fines de 2023. [ necesita actualización ]
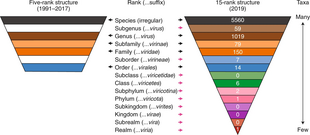
En 2022, la taxonomía ICTV enumeraba 11 273 especies de virus con nombre (incluidas algunas clasificadas como virus satélite y otras como viroides) en 2818 géneros, 264 familias, 72 órdenes, 40 clases, 17 filos, 9 reinos y 6 reinos. [1] Sin embargo, el número de virus con nombre supera considerablemente el número de especies de virus con nombre , ya que, a diferencia de los sistemas de clasificación utilizados en otras áreas de la biología, una "especie" de virus es un nombre colectivo para un grupo de virus (presumiblemente relacionados) que comparten ciertas características comunes (véase más abajo). Además, el uso del término "reino" en virología no equivale a su uso en otros grupos biológicos, donde refleja agrupaciones de alto nivel que separan tipos de organismos completamente diferentes (véase Reino (biología) ).
La definición formal y actualmente aceptada de "virus" fue aceptada por el Comité Ejecutivo del ICTV en noviembre de 2020 y ratificada en marzo de 2021, y es la siguiente: [2]
Los virus en sentido estricto se definen operativamente por el ICTV como un tipo de MGE que codifica al menos una proteína que es un componente principal del virión que encierra el ácido nucleico del MGE respectivo y, por lo tanto, el gen que codifica la proteína principal del virión en sí o MGE que se puede demostrar claramente que son miembros de una línea de descendencia evolutiva de dichas entidades principales que codifican proteínas del virión. Cualquier grupo monofilético de MGE que se origina a partir de un ancestro que codifica proteínas del virión debe clasificarse como un grupo de virus.
Las especies forman la base de cualquier sistema de clasificación biológica. Antes de 1982, se pensaba que los virus no podían adaptarse al concepto reproductivo de especie de Ernst Mayr y, por lo tanto, no eran susceptibles de dicho tratamiento. En 1982, el ICTV comenzó a definir una especie como "un conjunto de cepas" con cualidades de identificación únicas. En 1991, se adoptó el principio más específico de que una especie de virus es una clase politética de virus que constituye un linaje replicante y ocupa un nicho ecológico particular. [3]
En 2021 (la última edición del ICVCN), la definición de especie del ICTV establece: "Una especie es el nivel taxonómico más bajo en la jerarquía aprobada por el ICTV. Una especie es un grupo monofilético de MGE ( elementos genéticos móviles ) cuyas propiedades pueden distinguirse de las de otras especies mediante múltiples criterios", con el comentario "Los criterios por los que se distinguen las diferentes especies dentro de un género serán establecidos por el Grupo de Estudio apropiado. Estos criterios pueden incluir, entre otros, el rango de hospedadores naturales y experimentales, el tropismo celular y tisular, la patogenicidad, la especificidad del vector, la antigenicidad y el grado de parentesco de sus genomas o genes. Los criterios utilizados deben publicarse en la sección correspondiente del Informe del ICTV y revisarse periódicamente por el Grupo de Estudio apropiado". [4]
Existen muchos virus nombrados individualmente (a veces denominados "cepas de virus") por debajo del rango de especie de virus . El ICVCN da los ejemplos del virus del mosaico del caupí y el virus de la raya del maní, que están clasificados en la especie Virus del mosaico común del frijol , este último un miembro del género Potyvirus que a su debido tiempo recibirá un nombre binomial como Potyvirus [especie...] . Como otro ejemplo, el virus SARS-CoV-1 , que causa el síndrome respiratorio agudo severo ( SARS ) es diferente del virus SARS-CoV-2 , la causa de la pandemia de COVID-19 , pero ambos están clasificados dentro de la misma especie de virus, un miembro del género Betacoronavirus que actualmente se conoce como coronavirus relacionado con el síndrome respiratorio agudo severo que, según el mandato de 2021 del ICTV, también recibirá un nombre binomial a su debido tiempo. Como se establece en el ICVCN, sección 3.4, los nombres [y definiciones] de taxones por debajo del rango de especie no están regidos por el ICTV; "La denominación de dichas entidades no es responsabilidad del ICTV sino de grupos especializados internacionales. Es responsabilidad de los Grupos de Estudio del ICTV considerar cuál es la mejor manera de clasificar estas entidades en especies". [4] Utilizando el ejemplo dado anteriormente, el virus que causa la pandemia de COVID-19 recibió la designación "SARS-CoV-2" por parte del Grupo de Estudio Coronaviridae (CSG) del Comité Internacional de Taxonomía de Virus en 2020; en la misma publicación, este Grupo de Estudio recomendó una convención de denominación para aislamientos particulares de este virus "que se asemeje a los formatos utilizados para aislamientos de coronavirus aviares, filovirus y virus de la influenza" en el formato virus/huésped/ubicación/aislamiento/fecha, con un ejemplo citado como "SARS-CoV-2/humano/Wuhan/X1/2019". [5]
El Comité Internacional de Taxonomía de Virus comenzó a idear e implementar reglas para la denominación y clasificación de los virus a principios de la década de 1970, un esfuerzo que continúa hasta el presente. El ICTV es el único organismo encargado por la Unión Internacional de Sociedades de Microbiología con la tarea de desarrollar, refinar y mantener una taxonomía universal de virus, siguiendo los métodos establecidos en el Código Internacional de Clasificación y Nomenclatura de Virus. [4] [6] El sistema comparte muchas características con el sistema de clasificación de organismos celulares , como la estructura del taxón . Sin embargo, existen algunas diferencias, como el uso universal de cursiva para todos los nombres taxonómicos, a diferencia del Código Internacional de Nomenclatura para algas, hongos y plantas y el Código Internacional de Nomenclatura Zoológica .
La clasificación viral comienza a nivel de reino y continúa de la siguiente manera, con los sufijos taxonómicos entre paréntesis: [4]
En paralelo al sistema de nomenclatura binomial adoptado en las especies celulares, el ICTV ha ordenado recientemente (2021) que las nuevas especies de virus se denominen utilizando un formato binomial ( especie de género , p. ej. , Betacoronavirus panicum ), y que los nombres de especies de virus preexistentes se reemplacen progresivamente por nuevos nombres en formato binomial. [7] Una revisión de mediados de 2023 del estado de este cambio afirmó: "... un gran número de propuestas [relativas a la nomenclatura de los virus, presentadas al Comité Ejecutivo (CE) del ICTV para su consideración] renombraron especies existentes para cumplir con el formato de nomenclatura binomial recientemente ordenado. Como resultado, 8.982 de las 11.273 especies actuales (80%) ahora tienen nombres binomiales. El proceso concluirá en 2023, y las 2.291 especies restantes serán renombradas". [8]
A partir de 2021, se utilizan todos los niveles de taxones excepto el subreino, el subreino y la subclase. Se reconocen seis reinos, una clase incertae sedis , 22 familias incertae sedis y dos géneros incertae sedis : [9]
Reinos :
Clases de inciertae sedis :
Familias de Incertae sedis :
Géneros Incertae sedis :
Se ha sugerido que la similitud en el ensamblaje y la estructura del virión observada para ciertos grupos virales que infectan huéspedes de diferentes dominios de la vida (por ejemplo, tectivirus bacterianos y adenovirus eucariotas o Caudovirales procariotas y herpesvirus eucariotas) refleja una relación evolutiva entre estos virus. [10] Por lo tanto, se ha sugerido que la relación estructural entre virus se utilice como base para definir taxones de nivel superior (linajes virales basados en la estructura) que podrían complementar el esquema de clasificación ICTV de 2010. [11]
El ICTV ha ido añadiendo gradualmente muchos taxones de nivel superior utilizando relaciones en los pliegues de proteínas. Los cuatro reinos definidos en la versión de 2019 se definen por la presencia de una proteína de una determinada familia estructural. [12]

La clasificación de Baltimore (definida por primera vez en 1971) es un sistema de clasificación que coloca a los virus en uno de siete grupos dependiendo de una combinación de su ácido nucleico ( ADN o ARN ), cadena (monocatenaria o bicatenaria), sentido y método de replicación . [13] Nombrados en honor a David Baltimore , un biólogo ganador del Premio Nobel , estos grupos se designan con números romanos . Otras clasificaciones están determinadas por la enfermedad causada por el virus o su morfología, ninguna de las cuales es satisfactoria debido a que diferentes virus causan la misma enfermedad o se ven muy similares. Además, las estructuras virales a menudo son difíciles de determinar bajo el microscopio. Clasificar los virus según su genoma significa que todos los de una categoría dada se comportarán de manera similar, lo que ofrece alguna indicación de cómo proceder con futuras investigaciones. Los virus se pueden colocar en uno de los siete grupos siguientes: [14]

Los virus con un genoma de ADN , a excepción de los virus de transcripción inversa de ADN, son miembros de tres de los cuatro reinos virales reconocidos : Duplodnaviria , Monodnaviria y Varidnaviria . Pero el orden incertae sedis Ligamenvirales , y muchas otras familias y géneros incertae sedis , también se utilizan para clasificar los virus de ADN. Los dominios Duplodnaviria y Varidnaviria consisten en virus de ADN bicatenario; otros virus de ADN bicatenario son incertae sedis . El dominio Monodnaviria consiste en virus de ADN monocatenario que generalmente codifican una endonucleasa HUH ; otros virus de ADN monocatenario son incertae sedis . [15]
Todos los virus que tienen un genoma de ARN , y que codifican una ARN polimerasa dependiente de ARN (RdRp), son miembros del reino Orthornavirae , dentro del reino Riboviria . [16]
Todos los virus que codifican una transcriptasa inversa (también conocida como RT o polimerasa de ADN dependiente de ARN) son miembros de la clase Revtraviricetes , dentro del filo Arterviricota , reino Pararnavirae y reino Riboviria . La clase Blubervirales contiene la familia única Hepadnaviridae de virus de transcripción inversa (RT) de ADN; todos los demás virus RT son miembros de la clase Ortervirales . [17]
Holmes (1948) utilizó una taxonomía linneana con nomenclatura binomial para clasificar los virus en tres grupos dentro de un orden, Virales. Se ubican de la siguiente manera:
El sistema no fue aceptado por otros debido a que no tenía en cuenta las similitudes morfológicas. [18]
Los agentes infecciosos son más pequeños que los virus y tienen solo algunas de sus propiedades. [19] [20] Desde 2015, el ICTV ha permitido clasificarlos de manera similar a los virus. [21]
Los virus satélite dependen de la coinfección de una célula huésped con un virus auxiliar para su multiplicación productiva. Sus ácidos nucleicos tienen secuencias de nucleótidos sustancialmente distintas de las de su virus auxiliar o del huésped. Cuando un agente subviral satélite codifica la proteína de la cubierta en la que está encapsulado, se lo denomina virus satélite.
Los ácidos nucleicos satélite se parecen a los ácidos nucleicos satélite en que se replican con la ayuda de virus auxiliares. Sin embargo, se diferencian en que pueden codificar funciones que pueden contribuir al éxito de sus virus auxiliares; si bien a veces se los considera elementos genómicos de sus virus auxiliares, no siempre se encuentran dentro de ellos. [19]
Las partículas interferentes defectuosas son virus defectuosos que han perdido su capacidad de replicarse, excepto en presencia de un virus auxiliar, que normalmente es el virus parental. También pueden interferir con el virus auxiliar.
Los viriformes son una categoría polifilética de elementos virales endógenos . En algún momento de su evolución, fueron "domesticados" por su anfitrión como parte clave del ciclo de vida del anfitrión. El ejemplo prototípico son los miembros de los (también polifiléticos) Polydnaviriformidae , que son utilizados por las avispas para enviar fragmentos de ADN que debilitan la inmunidad a la presa empaquetándolos en partículas similares a viriones . Otros miembros son los llamados agentes de transferencia de genes (GTA) que se encuentran entre los procariotas. Las partículas de GTA se parecen a los fagos con cola , pero son más pequeñas y llevan principalmente fragmentos aleatorios de ADN del anfitrión. Los GTA son producidos por el anfitrión en momentos de estrés; la liberación de GTA mata a la célula huésped, pero permite que fragmentos de su material genético vivan en otras bacterias, generalmente de la misma especie. [25] Los tres clados conocidos de GTA, Rhodogtaviriformidae , Bartogtaviriformidae y Brachygtaviriformidae , surgieron independientemente de diferentes partes del árbol genealógico de Caudoviricetes . [26]
{{cite web}}: CS1 maint: varios nombres: lista de autores ( enlace )