La estructura biomolecular es la forma tridimensional intrincada y plegada que forma una molécula de proteína , ADN o ARN , y que es importante para su función. La estructura de estas moléculas puede considerarse en cualquiera de varias escalas de longitud que van desde el nivel de átomos individuales hasta las relaciones entre subunidades proteicas completas . Esta útil distinción entre escalas se expresa a menudo como una descomposición de la estructura molecular en cuatro niveles: primario, secundario, terciario y cuaternario. El andamiaje para esta organización multiescala de la molécula surge en el nivel secundario, donde los elementos estructurales fundamentales son los diversos enlaces de hidrógeno de la molécula . Esto conduce a varios dominios reconocibles de la estructura de la proteína y la estructura del ácido nucleico , incluidas características de la estructura secundaria como hélices alfa y láminas beta para proteínas, y bucles de horquilla , protuberancias y bucles internos para ácidos nucleicos. Los términos estructura primaria , secundaria , terciaria y cuaternaria fueron introducidos por Kaj Ulrik Linderstrøm-Lang en sus Lane Medical Lectures de 1951 en la Universidad de Stanford .
La estructura primaria de un biopolímero es la especificación exacta de su composición atómica y los enlaces químicos que conectan esos átomos (incluida la estereoquímica ). Para un biopolímero típico no ramificado ni reticulado (como una molécula de una proteína intracelular típica , o de ADN o ARN ), la estructura primaria es equivalente a especificar la secuencia de sus subunidades monoméricas , como aminoácidos o nucleótidos .
La estructura primaria de una proteína se informa comenzando desde el extremo N amino hasta el extremo C carboxilo , mientras que la estructura primaria de la molécula de ADN o ARN se conoce como la secuencia de ácido nucleico informada desde el extremo 5' hasta el extremo 3' . La secuencia de ácido nucleico se refiere a la secuencia exacta de nucleótidos que componen la molécula completa. A menudo, la estructura primaria codifica motivos de secuencia que son de importancia funcional. Algunos ejemplos de tales motivos son: las cajas C/D [1] y H/ACA [2] de los snoRNAs , el sitio de unión LSm encontrado en los ARN espliceosomales como U1 , U2 , U4 , U5 , U6 , U12 y U3 , la secuencia Shine-Dalgarno , [3] la secuencia de consenso de Kozak [4] y el terminador de la ARN polimerasa III . [5]
La estructura secundaria de una proteína es el patrón de enlaces de hidrógeno en un biopolímero. Estos determinan la forma tridimensional general de los segmentos locales de los biopolímeros, pero no describen la estructura global de posiciones atómicas específicas en el espacio tridimensional, que se consideran estructura terciaria . La estructura secundaria se define formalmente por los enlaces de hidrógeno del biopolímero, como se observa en una estructura de resolución atómica. En las proteínas, la estructura secundaria se define por patrones de enlaces de hidrógeno entre los grupos amina y carboxilo de la cadena principal (los enlaces de hidrógeno cadena lateral-cadena principal y cadena lateral-cadena lateral son irrelevantes), donde se utiliza la definición DSSP de un enlace de hidrógeno.
La estructura secundaria de un ácido nucleico está definida por el enlace de hidrógeno entre las bases nitrogenadas.
Sin embargo, en el caso de las proteínas, el enlace de hidrógeno está correlacionado con otras características estructurales, lo que ha dado lugar a definiciones menos formales de la estructura secundaria. Por ejemplo, las hélices pueden adoptar ángulos diedros de la cadena principal en algunas regiones del diagrama de Ramachandran ; por lo tanto, un segmento de residuos con tales ángulos diedros a menudo se denomina hélice , independientemente de si tiene los enlaces de hidrógeno correctos. Se han propuesto muchas otras definiciones menos formales, que a menudo aplican conceptos de la geometría diferencial de curvas, como curvatura y torsión . Los biólogos estructurales que resuelven una nueva estructura de resolución atómica a veces asignan su estructura secundaria a simple vista y registran sus asignaciones en el archivo del Banco de Datos de Proteínas (PDB) correspondiente.
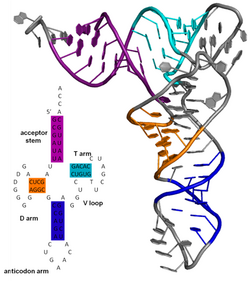
La estructura secundaria de una molécula de ácido nucleico se refiere a las interacciones de apareamiento de bases dentro de una molécula o conjunto de moléculas que interactúan. La estructura secundaria de los ARN biológicos a menudo se puede descomponer de forma única en tallos y bucles. A menudo, estos elementos o combinaciones de ellos se pueden clasificar aún más, por ejemplo, tetraloops , pseudonudos y tallo-bucles . Hay muchos elementos de estructura secundaria de importancia funcional para el ARN biológico. Los ejemplos famosos incluyen los tallo-bucles terminadores independientes de Rho y el trébol del ARN de transferencia (ARNt). Existe una pequeña industria de investigadores que intenta determinar la estructura secundaria de las moléculas de ARN. Los enfoques incluyen métodos tanto experimentales como computacionales (consulte también la Lista de software de predicción de la estructura del ARN ).
La estructura terciaria de una proteína o cualquier otra macromolécula es su estructura tridimensional, definida por las coordenadas atómicas. [6] Las proteínas y los ácidos nucleicos se pliegan en estructuras tridimensionales complejas que dan lugar a las funciones de las moléculas. Si bien dichas estructuras son diversas y complejas, a menudo están compuestas por motivos y dominios de estructura terciaria reconocibles y recurrentes que sirven como bloques de construcción molecular. Se considera que la estructura terciaria está determinada en gran medida por la estructura primaria de la biomolécula (su secuencia de aminoácidos o nucleótidos ).
La estructura cuaternaria de la proteína [a] se refiere al número y disposición de múltiples moléculas de proteína en un complejo de múltiples subunidades.
En el caso de los ácidos nucleicos, el término es menos común, pero puede referirse a la organización de nivel superior del ADN en la cromatina , [7] incluidas sus interacciones con las histonas , o a las interacciones entre unidades de ARN separadas en el ribosoma [8] [9] o el espliceosoma .
Los virus , en general, pueden considerarse como máquinas moleculares. El bacteriófago T4 es un virus particularmente bien estudiado y su estructura cuaternaria de proteínas está relativamente bien definida. [10] Un estudio de Floor (1970) [11] mostró que, durante la construcción in vivo del virus por proteínas morfogenéticas específicas , estas proteínas deben producirse en proporciones equilibradas para que se produzca el ensamblaje adecuado del virus. La insuficiencia (debido a la mutación ) en la producción de una proteína morfogenética particular (por ejemplo, una proteína crítica de la fibra de la cola), puede conducir a la producción de virus descendientes casi todos los cuales tienen muy poco del componente proteico particular para funcionar correctamente, es decir, para infectar células huésped. [11] Sin embargo, una segunda mutación que reduce otro componente morfogenético (por ejemplo, en la placa base o la cabeza del fago) podría en algunos casos restablecer un equilibrio de modo que una mayor proporción de las partículas de virus producidas sean capaces de funcionar. [11] De este modo, se descubrió que una mutación que reduce la expresión de un gen, cuyo producto se emplea en la morfogénesis, puede ser suprimida parcialmente por una mutación que reduce la expresión de un segundo gen morfogenético, lo que da como resultado una producción más equilibrada de los productos génicos del virus. El concepto de que, in vivo , es necesaria una disponibilidad equilibrada de componentes para una morfogénesis molecular adecuada puede tener una aplicabilidad general para comprender el ensamblaje de las máquinas moleculares de las proteínas.
El sondeo de estructura es el proceso mediante el cual se utilizan técnicas bioquímicas para determinar la estructura biomolecular. [12] Este análisis se puede utilizar para definir los patrones que se pueden utilizar para inferir la estructura molecular, el análisis experimental de la estructura y la función molecular y una mayor comprensión del desarrollo de moléculas más pequeñas para futuras investigaciones biológicas. [13] El análisis de sondeo de estructura se puede realizar a través de muchos métodos diferentes, que incluyen el sondeo químico, el sondeo de radicales hidroxilo, el mapeo de interferencia de análogos de nucleótidos (NAIM) y el sondeo en línea. [12]
Las estructuras de proteínas y ácidos nucleicos se pueden determinar utilizando espectroscopia de resonancia magnética nuclear ( RMN ) o cristalografía de rayos X o criomicroscopía electrónica de partícula única ( cryoEM ). Los primeros informes publicados para ADN (por Rosalind Franklin y Raymond Gosling en 1953) de patrones de difracción de rayos X de A-ADN —y también B-ADN— utilizaron análisis basados en transformadas de funciones de Patterson que proporcionaron solo una cantidad limitada de información estructural para fibras orientadas de ADN aisladas de timo de ternera . [14] [15] Luego, Wilkins et al. propusieron un análisis alternativo en 1953 para la difracción de rayos X de B-ADN y los patrones de dispersión de fibras de ADN hidratadas y orientadas a bacterias y cabezas de esperma de trucha en términos de cuadrados de funciones de Bessel . [16] Aunque la forma B-ADN es la más común en las condiciones que se encuentran en las células, [17] no es una conformación bien definida sino una familia o un conjunto difuso de conformaciones de ADN que ocurren en los altos niveles de hidratación presentes en una amplia variedad de células vivas. [18] Sus patrones de difracción y dispersión de rayos X correspondientes son característicos de los paracristales moleculares con un grado significativo de desorden (más del 20%), [19] [20] y la estructura no es manejable utilizando solo el análisis estándar.
Por el contrario, el análisis estándar, que involucra solo transformadas de Fourier de funciones de Bessel [21] y modelos moleculares de ADN , todavía se utiliza rutinariamente para analizar patrones de difracción de rayos X de A-ADN y Z-ADN. [22]

La predicción de la estructura biomolecular es la predicción de la estructura tridimensional de una proteína a partir de su secuencia de aminoácidos , o de un ácido nucleico a partir de su secuencia de nucleobases (bases). En otras palabras, es la predicción de la estructura secundaria y terciaria a partir de su estructura primaria. La predicción de la estructura es la inversa del diseño biomolecular, como en el diseño racional , el diseño de proteínas , el diseño de ácidos nucleicos y la ingeniería biomolecular .
La predicción de la estructura de las proteínas es uno de los objetivos más importantes que persiguen la bioinformática y la química teórica . La predicción de la estructura de las proteínas es de gran importancia en medicina (por ejemplo, en el diseño de fármacos ) y biotecnología (por ejemplo, en el diseño de nuevas enzimas ). Cada dos años, el rendimiento de los métodos actuales se evalúa en el experimento de Evaluación crítica de la predicción de la estructura de las proteínas ( CASP ).
También se ha realizado una cantidad significativa de investigación bioinformática dirigida al problema de la predicción de la estructura del ARN. Un problema común para los investigadores que trabajan con ARN es determinar la estructura tridimensional de la molécula dada solo la secuencia de ácido nucleico. Sin embargo, en el caso del ARN, gran parte de la estructura final está determinada por la estructura secundaria o las interacciones de apareamiento de bases intramoleculares de la molécula. Esto se demuestra por la alta conservación de los apareamientos de bases en diversas especies.
La estructura secundaria de las moléculas pequeñas de ácidos nucleicos está determinada en gran medida por interacciones locales fuertes, como los enlaces de hidrógeno y el apilamiento de bases . La suma de la energía libre para dichas interacciones, generalmente utilizando un método del vecino más cercano , proporciona una aproximación de la estabilidad de la estructura dada. [23] La forma más directa de encontrar la estructura de energía libre más baja sería generar todas las estructuras posibles y calcular la energía libre para ellas, pero el número de estructuras posibles para una secuencia aumenta exponencialmente con la longitud de la molécula. [24] Para moléculas más largas, el número de estructuras secundarias posibles es enorme. [23]
Los métodos de covariación de secuencias se basan en la existencia de un conjunto de datos compuesto por múltiples secuencias de ARN homólogas con secuencias relacionadas pero diferentes. Estos métodos analizan la covariación de los sitios de bases individuales en la evolución ; el mantenimiento en dos sitios ampliamente separados de un par de nucleótidos de apareamiento de bases indica la presencia de un enlace de hidrógeno estructuralmente requerido entre esas posiciones. Se ha demostrado que el problema general de la predicción de pseudonudos es NP-completo . [25]
El diseño biomolecular puede considerarse lo inverso de la predicción de la estructura. En la predicción de la estructura, la estructura se determina a partir de una secuencia conocida, mientras que en el diseño de proteínas o ácidos nucleicos se genera una secuencia que formará una estructura deseada.
Otras biomoléculas, como los polisacáridos , los polifenoles y los lípidos , también pueden tener una estructura de orden superior de consecuencia biológica.