La genética de poblaciones es una disciplina científica que contribuye al estudio de las migraciones históricas y evolutivas humanas. La investigación de dos marcadores uniparentales dentro de nuestro genoma, el cromosoma Y (ADN-Y) y el ADN mitocondrial (ADNmt), así como el ADN autosómico , proporciona información particularmente útil. Los datos del ADN-Y y del ADN autosómico sugieren que los croatas son en su mayoría descendientes de los eslavos del período de migración medieval , según el ADNmt tienen una diversidad genética que encaja en un paisaje genético materno europeo más amplio y, en general, tienen una uniformidad con otros eslavos del sur del territorio de la antigua Yugoslavia .
En Croacia se encuentran numerosos yacimientos del Paleolítico , la mayoría de ellos pertenecientes al Musteriense , en el Paleolítico Medio . En el Neolítico, en el sudeste de Europa se fundaron importantes culturas como Vinča , Varna y Starčevo . En la Edad del Bronce se produjo una simbiosis entre los protoindoeuropeos de la cultura kurgana y las poblaciones autóctonas, lo que dio lugar a la formación, entre otros, de los protoilirios . Poco a poco se fueron mezclando y fueron asimilados por los romanos , los celtas , los ostrogodos y, finalmente, los primeros eslavos a partir de finales del siglo VI.

En el estudio de 2014, de los tres perfiles de SNP generados con éxito de muestras de la cultura neolítica Starčevo de Vinkovci , dos pertenecían al haplogrupo de ADN-Y G2a-P15 y uno a I2a1-P37.2, lo que podría indicar a G2a como posibles representantes de la propagación de la agricultura desde el Cercano Oriente a Europa, mientras que I2a como sustrato mesolítico en Europa. [1] En el estudio de 2018, 10 de las 17 muestras de Croacia tuvieron una secuenciación de ADN-Y exitosa; dos muestras del Neolítico cardinal croata (6005-5786 a. C.) de la cueva de Zemunica pertenecían a C1a2 y E1b1b1a1b1, la cultura Starčevo del Neolítico temprano (5837-5659 a. C.) de Beli Manastir -Popova zemlja a C, la cultura Impressa croata del Neolítico temprano (5641-5560 a. C.) de Kargadur a G2a2a1, dos muestras de la cultura Sopot del Neolítico medio (5207-4546 a. C.) de Osijek a G2a2a1 y J2a1, Sopot del Neolítico tardío (4790-4558 a. C.) de Beli Manastir-Popova zemlja a I, dos muestras de la cultura Vučedol (2884-2582 a. C.) de Beli Manastir-Popova zemlja y El Tell de Vučedol pertenecía a R1b1a1a2a2 y G2a2a1a2a, y la muestra de la Edad del Bronce Temprano-Medio (1631-1521 a. C.) de Veliki Vanik pertenecía a J2b2a. [2]
En 2019 se analizaron autosómicamente tres individuos del siglo V con deformación craneal artificial de Osijek, probablemente de origen huno o germánico. Según el análisis de componentes principales y los métodos de mezcla, uno de ellos tenía ascendencia del este/noreste asiático, otro europeo/caucásico y el tercero, de Oriente Próximo/norteafricano. [3]
En 2021, se realizó un análisis de todo el genoma de 38 de los 41 individuos de la masacre de hace 6200 años en el pueblo de Potočani en el este de Croacia. [4] [5] Pertenecían a la cultura Lasinja de la Edad del Cobre Medio, y autosómicamente el 70% de ellos no estaban en parentesco cercano, lo que implica "una comunidad compuesta por muchos grupos familiares". Según Admixture y PCA, reveló "que los individuos analizados están ligeramente desplazados del grupo neolítico de Anatolia en la dirección de los cazadores-recolectores de Europa occidental, similar a otros agricultores europeos del Neolítico medio a tardío antes de la llegada de la ascendencia esteparia [occidental] ", ya que en promedio tenían aproximadamente un 91% de ascendencia relacionada con los agricultores neolíticos de Anatolia (ANF) y un 9% de cazadores-recolectores occidentales (WHG), siendo muy distantes de los croatas modernos. Entre ellos se encontraban "linajes paternos típicos de las poblaciones neolíticas de los Balcanes (once G2a2, dos I2a1a(xI2a1a2), un I2a2, C y C1a2-V20)". [6]
En el mismo año se analizaron 19 muestras adicionales del Neolítico Medio de la cultura de Sopot, 1 de la Edad del Cobre y 1 de la Edad Romana de Beli Manastir-Popova zemlja, así como 7 muestras de la Edad del Bronce de la cultura de Cerámica Incrustada Transdanubia de Jagodnjak -Krčevine en Croacia Oriental. Según Admixture, las muestras del Neolítico croata tienen un 97,6-100% de ascendencia ANF, las muestras de la Edad del Cobre tienen un 71% de ascendencia ANF y un 29% de ascendencia WSH, mientras que las muestras de la Edad del Bronce tienen un 47% de ascendencia ANF, un 20% WHG y un 33% WSG. Los haplogrupos de ADN-Y observados en el Neolítico/Período del Cobre fueron dos G2a2a-PF3147, un G2a2b2a1a1-PF3345, un C1a2b-Z38888, dos I2a2a-M223 y un J-M304; en el Período del Bronce solo hubo cuatro G2a2a1a2a2a1-Z31430 y un G2a2a1-PF3148. El único individuo del período romano del 260 al 402 d. C., "que puede o no ser representativo de la población más amplia en ese período de tiempo", tenía una combinación genética autosómica muy similar a los actuales europeos del sudeste (incluidos los croatas), "lo que agrega un punto de datos importante al cambio sustancial en la ascendencia que ocurrió en la región entre la Edad del Bronce y la actualidad". Llevaba el haplogrupo Y-ADN escita- euroasiático R1a1a1b2a2-Z2124 > R1a1a1b2a2b1-F1345 (subclado R-F1019* [7] ). [8] Entre 2018 y 2022 estudios analizaron muchas muestras relacionadas con la cultura Cetina , los protoilirios y tribus ilirias específicas ( Iapydes y Liburni ), encontrando que su linaje paterno pertenecía casi exclusivamente al haplogrupo J2b-L283. [2] [9] [10]
Un estudio arqueogenético de 2022 publicado en Science comparó muestras de población de los Balcanes antiguos, medievales y modernos y descubrió que las migraciones eslavas medievales "afectaron profundamente a la región", lo que resultó en la reducción de la ascendencia neolítica de Anatolia en el sudeste de Europa. Las poblaciones balcánicas preeslavas tienen la mayor parte del componente neolítico de Anatolia de ascendencia, mientras que los eslavos actuales fuera de los Balcanes tienen la menor cantidad, "con la gente actual del sudeste de Europa", incluidos los croatas, "en un punto intermedio entre los dos extremos", siendo los griegos y los albaneses los que más, mientras que los croatas y los húngaros los que menos. [11] Un estudio arqueogenético de 2023 publicado en Cell confirmó que la difusión de la lengua y la identidad eslavas se debió a grandes movimientos de personas, tanto hombres como mujeres, con ascendencia específica de Europa del Este y que "más de la mitad de la ascendencia de la mayoría de los pueblos de los Balcanes actuales proviene de las migraciones eslavas, con alrededor de un tercio de ascendencia eslava incluso en países como Grecia, donde hoy no se hablan lenguas eslavas". [12] [13] La población del período preeslavo de Croacia y la región tenía los haplogrupos de ADN-Y E-V13, R1b, J2b, J2a, G2a, mientras que con la migración eslava llegaron R1a-Z282 e I2a-L621, entre otros. [13]
Genéticamente , en la línea cromosómica Y paterna y en estudios publicados entre 2003 y 2008, una mayoría (>75%) de los croatas varones de Croacia pertenece a uno de los tres principales haplogrupos europeos de ADN-Y : I2 (32% [14] [15] [16] -34% [17] ), R1a (27% [17] -34% [14] [15] [16] ) y R1b (12% [17] -15% [14] [15] [16] ), mientras que una minoría (>25%) pertenece a los haplogrupos E (9% [17] ), I1 (5% [15] -9% [17] ), J (4% [17] ), N (2% [17] ) y G (1% [17] ). Según estudios recientes y más amplios publicados entre 2012 y 2022, una mayoría (65%) de los hombres croatas de Croacia pertenece a los haplogrupos I2 (39%-40%) y R1a (22%-24%), mientras que una minoría (35%) pertenece a los haplogrupos E (10%), R1b (6%-7%), J (6%-7%), I1 (5-8%), G (2%), Q (0-1,93%), H (0-1,8%), T (0,6%), N (0-0,6%) y L (0,2%). [18] [19]

El haplogrupo I entre los croatas de Croacia representa alrededor del 43,5%, [18] mientras que en Šokci , un subgrupo de croatas del este de Croacia, representa entre el 44% y el 50% del mismo haplogrupo. [20] [21] Se divide en dos subdivisiones principales: el subclado I2 (37,7%), típico de las poblaciones de los Balcanes y Europa del Este, y el I1 (5,8%), típico de las poblaciones de Escandinavia. [17] Del subclado I2, el más predominante es I2a1a (I-P37.2) es decir, su linaje de subclado I-M423 (18.400-14.000 YBP) > I-Y3104 (14.000-11.400 YBP) > I2a1a2b-L621 (11.400-6.500 YBP) > I-CTS10936 (6.500-5.600 YBP) > I-S19848 (5.600 YBP) > I-CTS4002 (5.600-5.100 YBP) > I-CTS10228 (5.100-3.400 YBP) > I-Y3120 (3.400-2.100 YBP) > I-S17250 (2.100-1.850 YBP) > I-PH908 (1.850-1.700 YBP), [22] que es típico de las poblaciones eslavas del sur del sudeste de Europa , siendo más alta en Bosnia-Herzegovina (>50%). [16] En Croacia, la frecuencia más alta se observa en Dalmacia , alcanzando su punto máximo en las ciudades de Split (50,48%), Dubrovnik (53%) y Zadar (60%), [23] [19] así como las islas del sur del Adriático de Vis (44,6%), [23] Brač y Korčula (~55%), y Hvar (52,88-65%). [17] [19] Se encuentra alrededor del promedio en las ciudades de Pula (31,31%) y Osijek (27,6-40,91%), [17] [19] algunas islas del sur de Mljet (33,3%), Lastovo (33,3%) y las islas del Adriático medio de Pašman (37,2%) y Pag (36,4%). [23] La frecuencia es más baja en la ciudad de Varaždin (18%) en el noroeste de Croacia, [19] en la región montañosa occidental de Žumberak (18,2%), [23] y en las islas del centro-norte de Cres (3%), Dugi otok (11,4%), Ugljan (24,6%) y Krk (27%). [14] [23] La frecuencia más alta se encuentra en los croatas de Bosnia y Herzegovina (63,8-73,3%). [16] [17] Según los 8 marcadores STR, las distancias genéticas más cercanas son las poblaciones de los países vecinos, pero también, dependiendo del método, Ucrania, Bielorrusia, Polonia y Rusia. [24]La frecuencia muy alta del subclado I-P37.2 en los Balcanes occidentales disminuye en todas las direcciones. [25] [23] La población con el haplogrupo I migró a Europa desde Oriente Medio, hace aproximadamente 25.000-13.000 años. Representa la población paleolítica y mesolítica de cazadores-recolectores en Europa. Sin embargo, en comparación con investigaciones anteriores que sostenían un origen autóctono prehistórico del haplogrupo contemporáneo I2 en Croacia y los Balcanes, [nb 1] Battaglia et al. (2009) ya observaron la mayor varianza del haplogrupo en Ucrania , y Zupan et al. (2013) señalaron que sugiere que llegó con la migración eslava desde la tierra natal que estaba en la actual Ucrania. [30] La reciente investigación de OM Utevska (2017), concluyó que los haplotipos del haplogrupo STR tienen la mayor diversidad en Ucrania, con el resultado del marcador STR ancestral "DYS448=20" que comprende el grupo " Dnieper - Cárpatos ", mientras que el resultado derivado más joven "DYS448=19" comprende el "grupo de los Balcanes" que predomina entre los eslavos del sur. [31] Este "grupo de los Balcanes" también tiene la mayor varianza en Ucrania, lo que indica que la frecuencia muy alta en los Balcanes occidentales se debe a un efecto fundador . [31] Utevska calculó que la divergencia del grupo STR y su expansión secundaria desde el curso medio del río Dnieper o desde los Cárpatos orientales hacia la península de los Balcanes ocurrió aproximadamente hace 2.860 ± 730 años, relacionándolo con los tiempos anteriores a los eslavos, pero mucho después del declive de la cultura Tripolye . [31] Sin embargo, los cálculos basados en STR dan fechas sobreestimadas, [32] [33] y más específicamente, el grupo está representado por un solo SNP, I-PH908, conocido como I2a1a2b1a1a1c en el árbol filogenético ISOGG (2019), y según YFull YTree se formó y tuvo TMRCA aproximadamente 1.850-1.700 YBP. [22] Aunque es dominante entre los pueblos eslavos modernos en el territorio de las antiguas provincias balcánicas del Imperio Romano , hasta ahora no se encontró entre las muestras del período romano y está casi ausente en la población contemporánea de Italia . [34] Según Pamjav et al. (2019) y Fóthi et al. (2020), la distribución de subclados ancestrales como el de I-CTS10228 entre los portadores contemporáneos indica una rápida expansión desde el sureste de Polonia., está principalmente relacionada con los eslavos y su migración medieval, y la "mayor explosión demográfica ocurrió en los Balcanes". [34] [35] Según Olalde et al. (2023) se confirmó que I2-L621 estuvo ausente antes del período medieval en los Balcanes y llegó con los eslavos. [13]
R1a1a1-M17 (22,1%-25,6%) es el segundo haplogrupo más predominante. [18] El haplogrupo R-M17 en Croacia se divide principalmente en dos subclados, R-M558 que es predominante (19,2%) y R-M458 (4,9%), mientras que R-Z282 es raro (1,2%). [23] Tiene la frecuencia más alta en la región norte (29,1%) y central (23,6%), y casi la misma frecuencia en la región este (18,6%), sur (19,1%) y oeste (20%) de Croacia. [18] La frecuencia local más alta de R1a1a1-M17 se observó en los croatas de Varaždin (38%) y Osijek (26-39%), [17] [19] Žumberak (34,1), [23] y en las islas centro-norteñas de Dugi Otok (34,1%), Krk (37%), Pašman (38%) y Cres (56,6%), [14] [23] siendo similar a los valores de los otros eslavos, como eslovenos, checos y eslovacos. La frecuencia es menor en Šokci de Croacia oriental (16%), [20] [21] en la ciudad de Dubrovnik (13,4%) y Split (19%) en Dalmacia, así como en las islas meridionales de Hvar (8-10,58%) y Vis (17%). [14] [23] [19] En los croatas de Bosnia, la frecuencia es similar a la de otros eslavos del sur (12%). [17] [36] Considerando los subclados, el único caso atípico es la isla de Cres, que tuvo un porcentaje casi igual de R1a-M558 (29,3%) y R1a-M458 (27,3%). [23] Según las distancias genéticas de 8 marcadores STR, las más cercanas son las poblaciones de los países cercanos, pero también, según el método, Bielorrusia, Eslovaquia, Polonia y Rusia. [24] El subclado R-M558 es más frecuente entre los eslavos orientales de Europa del Este y la región del Volga-Ural, mientras que el R-M458 entre los eslavos occidentales de Europa Central y Oriental. Ambos están presentes en "frecuencias informativas en poblaciones de los Balcanes con herencia eslava conocida". [37] El subclado R-M558 CTS1211 también se encontró entre los conquistadores húngaros, lo que indica mezcla y asimilación de los eslavos entre los húngaros. [38]
La frecuencia más alta del haplogrupo Haplogroup R1b (7,9%-9,1%), que en Croacia se divide en varios subclados (principalmente R-L23 y su subclado R-U152 ), se encuentra en la región norte (10,9%) y central (11,8%) de Croacia, [18] mientras que localmente en los croatas de la isla de Krk (16,2%) y Dugi Otok (25%), [14] [23] y Žumberak (11,3%), [23] mientras que en las islas del sur, ciudad de Dubrovnik (3,9%) y en los croatas de Bosnia está casi ausente (1-6%), [14] [17] [23] [19] o como en Osijek no se encontró. [17] Estos dos haplogrupos (R1a y R1b) están relacionados con la migración de los protoindoeuropeos desde el área euroasiática hace unos 5.000 años, [25] y R1a está relacionado particularmente con la migración de la población eslava. [16] [23] Su frecuencia muestra una gradación norte-sur y una distribución de frecuencia opuesta al haplogrupo I-P37.2, [25] y la frecuencia más alta se observa en el norte, centro y este de Croacia. [14] [17] [18]
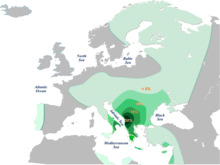
Del haplogrupo E (9,8%-10,6%) entre los croatas, el más frecuente es el subclado E1b1b1a1b-V13 (6,7%), mientras que E1b1b1a3-M149 y E1b1b1c-M123 también se encontraron en pequeñas cantidades (1,1%). [17] E-V13 es típico de las poblaciones del sudeste de Europa, alcanzando su pico entre los albanokosovares (44%), y también es alto entre los macedonios, griegos, rumanos, búlgaros y serbios. [16] La frecuencia más alta en el continente croata se ha encontrado en Varaždin (16%) y Žumberak (18,2%) [23] [19] en las islas centrales Dugi Otok (15,9%) y Ugljan (13,2%), así como en las islas meridionales Vis (23,4%) y Mljet (15,4%). [23] En las islas del norte de Cres (3%) [23] y Krk (6,8%) fue similar a otras islas del sur (3,7-4,3%). [14] En los croatas de Bosnia la frecuencia fue la misma que entre los croatas de Croacia (8,9%). [17] Los subclados de J1 son raros en Croacia, mientras que J2 es más alto en los croatas de Croacia, alcanzando su punto máximo en los croatas de Osijek (10,2%) y las islas centrales de Ugljan (10,2%) y Pašman (16,6%), así como la isla norte de Krk (10,8%) y Cres (14,1%), [14] [17] [23] que en los croatas de Bosnia (ambos 1,1%). [17] El subclado G2a-P15 tanto en croatas como en croatas de Bosnia se encuentra en números bajos (1,1%), [17] pero alcanza un pico local en la ciudad nororiental de Osijek (13,8%), [17] y las islas meridionales de Mljet (15,4%), Korčula (10,4%), Brač (6%) así como la isla norteña de Cres (7%). [14] [23] El haplogrupo E y J están relacionados con la migración neolítica post-LGM de una población de Anatolia que trajo consigo la domesticación de animales y plantas salvajes. Específicamente, el subclado del haplogrupo E probablemente surgió localmente en los Balcanes no antes de hace 8.000-10.000 años. Estos haplogrupos muestran una gradación sur-norte. [25] El haplogrupo G podría haber estado presente en Europa durante el LGM o la población con algunos de sus subclados que llegaron con los primeros agricultores. [25]
Los subclados del haplogrupo N son raros en Croacia (0-2,2%). [17] [23] [19] Es muy frecuente en el Lejano Oriente , como Siberia y China, mientras que en Europa en los finlandeses (60%) y en los países bálticos (45%). De manera inusual para las poblaciones europeas, otro haplogrupo Q de Asia central-Siberia se encontró en frecuencias inusualmente altas debido al efecto fundador en las islas de Hvar (7,69-14%), Lastovo (8,3%) y Korčula (6%). [14] [23] [19] También se encontró en un individuo de Herzegovina. [16] En estudios posteriores, se confirmó que un croata de Bosnia y Herzegovina pertenecía al subclado Q-L472>Z2902>B285>B29 que se encuentra en Asia Central y Oriental, [39] mientras que un croata de Croacia, otro de la isla de Hvar y un croata de origen judío en Polonia pertenecían al subclado Q-L245>Y2998>Y2209 que se encuentra principalmente en Oriente Medio y entre los judíos asquenazíes . [40]
La región de la actual Croacia era parte de una región balcánica más amplia que puede haber servido como uno de los varios refugios durante el LGM, una región de origen para la recolonización de Europa durante el período postglacial y el Holoceno (hace 10.000 años). [17] [25] La costa oriental del Adriático estaba mucho más al sur. [15] Las partes norte y oeste de ese mar eran estepas y llanuras, mientras que las islas croatas modernas (ricas en sitios arqueológicos del Paleolítico) eran colinas y montañas. [15] [17] La región tuvo un papel específico en la estructuración de la herencia genética paterna europea, y particularmente entre los eslavos, caracterizada por el predominio de R1a e I, y la escasez de linajes E. [16] La diversidad genética de la población insular contemporánea se caracteriza por un fuerte aislamiento y endogamia . [41]
En la tabla siguiente se cita el estudio más extenso (2012) hasta ahora sobre la población de Croacia. Es una base de datos de ADN de referencia nacional de 17 sistemas de loci que adquirieron haplotipos Y- STR que se predijeron en haplogrupos Y -SNP estimados (más del 90% de probabilidad) . [18] Las subpoblaciones se dividieron en cinco regiones cuyas subpoblaciones mostraron una fuerte similitud y homogeneidad de la contribución genética paterna, todas agrupadas, con excepción de la subpoblación del sur de Croacia que mostró una leve diferencia. Además de un alto grado de homogeneidad general, existen similitudes de gradiente con un grupo de Europa central (germánico, eslavo, húngaro) y un grupo de Europa meridional (bosnio, serbio, búlgaro, macedonio, rumano, albanés, sur de Italia), que van de norte a sur (y de este a oeste). [18] De los países vecinos y los pueblos étnicos más cercanos a ellos, Bosnia y Herzegovina y los bosnios . [42] [43] [44]
En el estudio de ADN-Y y ADNmt de 2014, una muestra mesolítica datada entre 6080 y 6020 a. C. de Vela Spila, cerca de Vela Luka, en la isla de Korčula, pertenecía al haplogrupo de ADNmt U5b2a5, común en las comunidades de cazadores-recolectores, mientras que a otras once muestras neolíticas de la cultura Starčevo, datadas entre 6000 y 5400 a. C. de Vinkovci , se les asignaron los haplogrupos J1c, K1a, T2b, HV0, K, V, V6, que revelan una diversidad de ADNmt similar y una ascendencia compartida en las primeras poblaciones agrícolas de la cuenca de Panonia y las poblaciones del LBK centroeuropeo , acompañadas de una reducción del sustrato de ADNmt mesolítico. [1] Los resultados preliminares del estudio de ADNmt de 2016, que incluirá aproximadamente 30 muestras del Neolítico y 5 muestras de la Edad de Bronce Temprana a Tardía, en 5 huesos petrosos croatas antiguos (3 Cardial Impresso neolíticos de la cueva Zemunica cerca de Bisko , 1 cultura Danilo del Neolítico medio de la cueva Zidana cerca de Staničići Žumberački, 1 Edad de Bronce media/tardía de la cueva Jazinka cerca de Nečven ) indicaron los haplogrupos de ADNmt K2 y K1b1a, H1e/H41, H1b para muestras neolíticas similares a los agricultores europeos tempranos (EEF) y los modernos sardos y europeos del sur, mientras que el haplogrupo HV o H4 para la muestra de la Edad de Bronce similar a la población croata y balcánica actual, pero sin evidencia clara de conexión con la migración indoeuropea. [47] El estudio de 2018 que incluyó 17 muestras de Croacia; Mesolítico (7308–7027 a. C.) de Vela Spila a U5b2b, tres muestras del Neolítico cardinal de Croacia (6005–5751 a. C.) de la cueva de Zemunica a H1, K1b1a y N1a1, Starčevo del Neolítico temprano (5837–5659 a. C.) de Beli Manastir-Popova zemlja a U8b1b1, dos muestras de la Impressa de Croacia del Neolítico temprano (5670–5560 a. C.) de Kargadur a H5a y H7c, dos muestras del Neolítico medio de Sopot (5207–4546 a. C.) de Osijek a U5a1a2 y H10, dos muestras del Neolítico tardío de Sopot (4790–4536 a. C.) de Beli Manastir-Popova zemlja a U5b2b y N1a1, Eneolítico (3710–3360 a. C.) desde Radovanci hasta J1c2, tres muestras de Vučedol (3000–2582 a. C.) de Beli Manastir-Popova zemlja y Vucedol Tell hasta T2e, T2c2 y U4a, Edad del Bronce Temprano-Medio (1631–1521 a. C.) desde Veliki Vanik hasta I1a1, y la muestra de Edad del Bronce Tardío (805–761 a. C.) de la cueva de Jazinka pertenecían a HV0e. [2]El estudio de 2021 incluyó 38 muestras neolíticas (4200 a. C.) de Potočani en el este de Croacia y pertenecían a H, H4a1, H5b, H7, H13b1, H26, H42, HV, J1, K1a1, K1a3a, K1a4a1, K1b1b1, N1a1a1, N1a1a1a2, N1a1a1a3, T1a2, T2b, T2b23, T2f, U2, U5b1d1 y X2b, "lo que sugiere que las víctimas de Potočani pertenecían a una gran comunidad con un grupo diverso de linajes femeninos". [6] En el mismo año, en un estudio de 28 muestras desde el Neolítico hasta la época romana en Croacia oriental, se encontraron H, HV9, J2b1a5, K1a, K1a1, K1a1a, K1a4, K1a5, K1b1b1, K2a, K2b1, N1a1a1, N1a1a1a2, N1a1a1a3, T2b, T2b11, T2b21, T2b3, T2c1d1, T2f2, U2e1a1, U5a1g, U5b1b1a, U5b2b, U8b1a1. [8]
El estudio de ADNmt de 2011 sobre 27 restos esqueléticos medievales tempranos en Naklice cerca de Omiš en el sur de Dalmacia mostró que el 67% pertenecía al haplogrupo H, el 18% al J, el 11% al U5 y el 4% al HV. [48] El estudio de ADNmt de 2015 sobre restos esqueléticos medievales en Šopot (siglos XIV-XV) y Ostrovica (siglo IX) en el norte de Dalmacia confirmó que los perfiles heredados por la línea materna no diferían ni entre los yacimientos de Ostrovica y Šopot ni entre las poblaciones medievales y modernas, mostrando la misma prevalencia de haplogrupos (H, J, U) tanto en las poblaciones medievales como en las contemporáneas. [49] El estudio de 2014 de un esqueleto masculino encontrado en Split del Período Romano Tardío (fechado en 1760±80 YBP) mostró que pertenecía al haplogrupo H. [50]

Genéticamente, en la línea de ADN mitocondrial materno , una mayoría (>65%) de croatas de Croacia (continental y costa) pertenecen a tres de los once principales haplogrupos europeos de ADNmt: H (25,5-45%), U (18,85-22%), J (7,5-11%), mientras que una gran minoría (>35%) pertenece a muchos otros haplogrupos más pequeños. [51] [52]
En todos los estudios, el haplogrupo H es el haplogrupo materno más frecuente (45% en 2014) en el continente y la costa de Croacia respectivamente, pero en el estudio más reciente de 2020 tiene frecuencias más bajas del 25,5% debido a diferencias de nomenclatura principalmente de linajes R/R0 (7,5%). [52] La frecuencia más alta en Croacia se observó en la población de la isla Korčula (60,2%), Susak (66%) y Mljet (73,5%), [51] [53] mientras que la frecuencia más baja en las islas Cres (27,7%), [54] y Hvar (27,8%). [55] Es el haplogrupo europeo dominante. [51] La elevada frecuencia del subhaplogrupo H1b en Mljet (30,9%), por lo demás poco común en otros estudios, es un ejemplo típico de un efecto fundador: migración desde la región costera más cercana y expansión microevolutiva en la isla. [51]
El haplogrupo U (18,85% en 2014, [56] 22% en 2020 [52] ) está representado principalmente por su subclado U5 , que es el segundo haplogrupo más frecuente, con un 11,6% en el continente y un 10,4% en la costa, con frecuencias similares en las islas de Brač (8,6%), Krk (11,3%) y Hvar (14,8%), mientras que la más baja en Korčula (2%). [55] En general, el haplogrupo U, incluidos sus subclados como U5, es el más frecuente en la ciudad de Dubrovnik (19,9%) y las islas Lastovo (23,5%) y Cres (29,4%). [54] [51] Es el haplogrupo europeo más antiguo (edad de coalescencia alrededor de 50.000 YBP) y su subclado U5 constituye la mayor parte de la diversidad de haplogrupos en Europa. La alta frecuencia de U4 en Lastovo (11,8%) indica efecto fundador. [51]
El haplogrupo J es el tercer haplogrupo más frecuente (7,5% en 2020 [52] ), con un 11,9% en el continente pero solo un 3,1% en la costa, sin embargo, las islas tuvieron frecuencias más altas que la población costera Korčula (6,1%), Brač (8,6%), Krk (9%), Hvar (9,3%), [55] pico en Žumberak (12,34%) [56] y Lastovo (19,6%), [51] mientras que en Cres está casi totalmente ausente. [54]
El haplogrupo T es el tercer o cuarto haplogrupo más frecuente (10% en 2020 [52] ). Su subclade T2 tiene una frecuencia similar de 3,1-5,8% tanto en la población costera y continental como en la insular, con un pico excepcional en la isla de Hvar (12%), [55] [54] y Susak (33%), [53] sin embargo, el haplogrupo T general tiene una frecuencia menor en Mljet (1,5%), Lastovo (3,9%) y Dubrovnik (2,3). [51]
El haplogrupo K (7,5% en 2020 [52] ) tiene una frecuencia media del 3,6% en el continente y del 6,3% en la costa, está ausente en Lastovo [51] y tiene la frecuencia más baja en las islas Cres (3,36%) [54] y Hvar (3,7%), mientras que la más alta en la isla Brač (9,5%). [55]
El haplogrupo V (bajo HV > HV0) es un clado hermano menor del haplogrupo H, [51] y tiene casi la misma frecuencia mínima y máxima (4-5,7%) tanto en poblaciones continentales como insulares, con excepción de Korčula (8,2%), [55] así como una frecuencia menor en Mljet (1,5%), Lastovo (2%) y Dubrovnik (3,3%). [51]
La frecuencia del haplogrupo W en la población continental y costera está entre el 2,2 y el 4,2%, mientras que entre las poblaciones insulares es del 1,9-3,1%, con excepción de Krk (7,5%), [55] y Cres (12,6%). [54] En las islas Mljet y Lastovo está entre el 4,4 y el 5,9%, mientras que en Dubrovnik está casi ausente (0,6%). [51]
Otros haplogrupos de ADNmt con picos locales notables son: subclados HV con frecuencias bajas en el continente y la costa (0,4-2,1%) pero promedio (4,1-4,6) en las islas, y altas en Dubrovnik (7,7%) [51] y Brač (10,5%). [55] El haplogrupo N1a en Cres (9,24%) es el hallazgo más septentrional hasta ahora de esta rama en Europa, y los haplotipos indican un efecto fundador relativamente reciente. [54] Es un haplogrupo característico de los primeros agricultores. [1] El haplogrupo F que está casi ausente (solo 0,2-0,4% en el continente), pero alcanza un pico de 8,3% en Hvar. [55] [56] El haplogrupo I en Krk (11,3%), cuyos subhaplogrupos se separaron alrededor del LGM. [57] El haplogrupo X varía entre 0,63 y 3,17 %, y pertenece principalmente al subclado X2 > X2b, [56] y una investigación reciente de Cres y Rab posiblemente encontró un linaje X3 "nuevo y específico de la isla" que "se formó dentro de la población croata". [58]
Durante décadas, las poblaciones insulares croatas han sido estudiadas debido a su aislamiento, lo que permite rastrear procesos microevolutivos y comprender fuerzas evolutivas, como la deriva genética (expresión genética específica), el efecto fundador y los cuellos de botella poblacionales (reducción del tamaño de la población) que dieron forma a la población contemporánea. Los resultados hasta ahora indican que el flujo genético y la afluencia de mujeres a las islas fueron limitados. [51] También se puede considerar un aislamiento genético moderado para la población continental de la región montañosa de Žumberak porque tenían una afinidad suelta con la región de origen propuesta por Uskoks (bosnios, herzegovinos, croatas del sur) o con sus vecinos más cercanos actuales (croatas del norte y eslovenos). [56] En el ejemplo de la población de la isla de Krk, el análisis de ADNmt de alta resolución mostró evidencia de que los asentamientos de Omišalj , Vrbnik y Dobrinj están relacionados en un grupo conjunto de asentamientos eslavos tempranos, mientras que las regiones de Poljica y Dubašnica son un grupo separado fundado por inmigrantes eslavos y valacos o morlacos del interior de Velebit que llegaron en el siglo XV. [57] En el ejemplo de la población de la isla de Mljet se pueden percibir eventos demográficos e históricos como el uso de la isla como estación de cuarentena, mientras que a lo largo de Vis y Lastovo se practica la consanguinidad y la endogamia debido a la falta de diversidad genética, siendo adecuada para la investigación genético-epidemiológica. [51]
En el análisis de mtADN de 2004, un grupo estaba formado por poblaciones de las islas Hvar, Krk y Brač, y un segundo grupo incluía el continente croata y la costa croata, mientras que la isla de Korčula se distinguió debido a una frecuencia excepcionalmente alta del haplogrupo H. [55] En el análisis de PCA interpoblacional de mtADN de 2009 de los subhaplogrupos, las poblaciones insulares de Krk, Ugljan , Korčula, Brač y Hvar se agruparon, lo que implica que tenían linajes maternos cercanos, con Vis cerca de ellos, pero Rab (U4, H6, J1c) y especialmente Cres (prevalencia U2, W, N1a) tenían posiciones periféricas separadas tanto del grupo como entre sí, y confirmaron "que la deriva genética, especialmente el efecto fundador, ha jugado un papel importante en la configuración de la composición genética de la población aislada de la isla de Cres". [54] En el estudio de 2017, el mayor caso atípico debido al aislamiento, la endogamia y el índice de diversidad genética informado más bajo fue Susak. [53] En el análisis PCA de mtADN de 2014, las poblaciones del este y sur de Croacia se agruparon junto con Bosnia y Herzegovina, mientras que el oeste y norte de Croacia con Eslovenia. Como la población eslovena no forma parte del grupo del sudeste de Europa, se considera un posible aporte de diferentes olas migratorias de eslavos en la Edad Media . [56]

Según la encuesta de IBD autosómica de 2013 "de ascendencia genealógica reciente durante los últimos 3000 años a escala continental", los hablantes de la lengua serbocroata comparten un número muy alto de ancestros comunes que datan del período de migración hace aproximadamente 1500 años con Polonia y el grupo de Rumania-Bulgaria, entre otros en Europa del Este. Se concluye que es causado por la expansión huna y eslava, que fue una "población relativamente pequeña que se expandió sobre una gran área geográfica", particularmente "la expansión de las poblaciones eslavas en regiones de baja densidad de población a partir del siglo VI" y que es "altamente coincidente con la distribución moderna de las lenguas eslavas". [60] Según Kushniarevich et al., el estudio de Hellenthal et al. El análisis de IBD de 2014 también encontró "eventos de mezcla multidireccional entre los europeos del este (tanto eslavos como no eslavos), que datan de alrededor de 1000-1600 YBP", lo que coincide con "el marco temporal propuesto para la expansión eslava". [61] El análisis de IBD de 2015 encontró que los eslavos del sur tienen una proximidad menor a los griegos que con los eslavos del este y del oeste, y que hay "patrones uniformes de intercambio de IBD entre los eslavos del este y el oeste - poblaciones 'intereslavas' ( húngaros , rumanos y gagauz ) - y los eslavos del sur, es decir, en un área de supuestos movimientos históricos de personas, incluidos los eslavos". El ligero pico de segmentos de IBD compartidos entre los eslavos del sur y del este y el oeste sugiere una "ascendencia compartida en tiempo eslavo". [61] La comparación del análisis de IBD de 2014 de las poblaciones de los Balcanes occidentales y de Oriente Medio también encontró un flujo genético insignificante entre los siglos XVI y XIX durante la islamización de los Balcanes. [36] En el análisis de 2022, los individuos croatas, bosnios y herzegovinos y serbios formaron el grupo "Balcanes noroccidentales", que tenía menos ascendencia del sur de Europa ("griega") que el grupo "Balcanes nororientales", formado por individuos rumanos y búlgaros. El grupo "Balcanes noroccidentales" también tenía "segmentos de IBD dentro del grupo ligeramente más largos que los de los Balcanes nororientales, lo que coincide con un Ne consistentemente más bajo y un ROH elevado , lo que sugiere una población más pequeña que el noreste de los Balcanes". [59]
Según un análisis autosómico de 2014 de los Balcanes occidentales, la población croata muestra uniformidad genética con otras poblaciones eslavas del sur. Los croatas y bosnios estaban más cerca de las poblaciones de Europa del Este y se superponían en gran medida con los húngaros de Europa Central. [36] En el análisis de 2015, formaron un grupo eslavo del sur occidental con los bosnios y eslovenos en comparación con el grupo oriental formado por macedonios y búlgaros con los serbios en el medio. El grupo occidental tiene una inclinación hacia los húngaros, checos y eslovacos, mientras que el grupo oriental hacia los rumanos y en cierta medida los griegos. [61] El componente genético ancestral modelado de los baltoeslavos entre los eslavos del sur estaba entre el 55 y el 70%. [61] En el análisis de 2018 de la población eslovena , la población croata nuevamente se agrupó con eslovenos, húngaros y estaba cerca de los checos. La población de Croacia comparte en su mayoría una ascendencia común con los europeos orientales, occidentales y meridionales, y casi no tiene relación con poblaciones aisladas como los sardos y los vascos . [36] [62]
Según el estudio arqueogenético de 2023 basado en modelos autosómicos qpAdm, los croatas actuales tienen un 66,5 % de ascendencia eslava medieval temprana de Europa central y oriental, un 31,2 % de ascendencia romana local de Croacia y Serbia y un 2,4 % de ascendencia de Anatolia occidental de la era imperial romana. [13]
Según estudios de marcadores STR autosómicos de 2005 y 2006, las islas más aisladas fueron Korčula, [63] y Susak. [41] Un estudio de secuenciación del exoma completo de 2016 de 176 individuos de la isla de Vis confirmó el estado de aislamiento de la población de la isla y reveló el "patrón de mutaciones de pérdida de función, que se asemeja a los rastros de la evolución adaptativa". [64] El estudio del marcador STR autosómico de 2022 sobre 2877 individuos no relacionados de subpoblaciones continentales (ciudades de Zagreb, Pazin, Delnice, Zabok y Donji Miholjac, y la región de Baranja) e insulares (Krk, Cres, tres islas de Dalmacia del Norte Ugljan-Pašman-Dugi Otok, Brač, Hvar, Korčula y Vis) encontró una mayor diferenciación genética (0,005) en comparación con el sudeste de Europa (0,002), lo que indica "un cierto grado de aislamiento genético, probablemente debido a la influencia de la endogamia dentro de las poblaciones insulares rurales". La población croata es la más cercana a Bosnia y Herzegovina, Hungría y Eslovenia. Hay cuatro grupos principales visibles dentro de los croatas muestreados, el primero incluye las islas continentales-Brač y Krk-Dalmacia del Norte, el segundo Hvar-Korčula, el tercero Cres y el cuarto Vis, los dos últimos con las distancias más altas de los demás. [65]
Din-N es más antiguo que Din-S. N=al norte del Danubio y S=al sur del río Danubio... 8 de mayo de 2007: Dinárico I1b1 y DYS 448. DYS448 19 para S y 20 para N.
Si bien las razones de la diferencia entre las tasas genealógicas y evolutivas de Y-STR se entienden en parte, sigue sin estar claro qué tasa utilizar. Muchos han aplicado la tasa evolutiva, aunque unos pocos han utilizado las tasas genealógicas, o ambas. Los genealogistas genéticos generalmente aplican la tasa genealógica y critican los estudios genéticos de población por informar (en su opinión) edades tres veces sobreestimadas ... La edad de cada haplogrupo también se calculó utilizando la tasa genealógica STR y la tasa evolutiva STR. Los intervalos de confianza para las dos edades basadas en STR (no se muestran en el gráfico) no se superponen. Por ejemplo, la edad genealógica de I2a-L621 (2200 ± 500 años) alcanza la edad de la envoltura (de 2600 a 3100 años), mientras que la edad evolutiva se encuentra mucho más allá (9900 ± 2700 años). El patrón observado (Fig. 2a) difiere claramente para haplogrupos de diferentes clases de edad. Para edades inferiores a 7000 años, la tasa genealógica de STR proporciona resultados consistentes con las edades "reales" o que las subestiman ligeramente, mientras que la tasa evolutiva da como resultado sobrestimaciones tres veces superiores. Para edades entre aproximadamente 7000 y 15 000 años, ninguna de las tasas de STR proporciona resultados correctos. Para haplogrupos de más de 15 000 años, la tasa evolutiva estima correctamente o sobreestima la edad "real".
Observamos 16 loci de 640 muestras I2a-L621 en la base de datos del proyecto I2a de FTDNA y encontramos que 7 individuos estaban a 2 pasos genéticos de las muestras de Karos, de los cuales 1 era húngaro de Kunszentmárton, 2 eran ucranianos, 1 era lituano, 1 era bielorruso, 1 era ruso y 1 era alemán de Polonia. Según el análisis de SNP, el grupo CTS10228 tiene una antigüedad de 2200 ± 300 años. La expansión demográfica del grupo puede haber comenzado en el sudeste de Polonia en esa época, ya que los portadores del subgrupo más antiguo se encuentran allí en la actualidad. El grupo no puede vincularse únicamente a los eslavos, porque el período protoeslavo fue posterior, alrededor del 300-500 d. C. La edad basada en SNP de la rama CTS10228 de Europa del Este es de 2200 ± 300 años. Los portadores del subgrupo más antiguo viven en el sudeste de Polonia, y es probable que la rápida expansión demográfica que llevó el marcador a otras regiones de Europa comenzara allí. La mayor explosión demográfica se produjo en los Balcanes, donde el subgrupo es dominante en el 50,5% de los croatas, el 30,1% de los serbios, el 31,4% de los montenegrinos y en aproximadamente el 20% de los albaneses y griegos. Como resultado, este subgrupo a menudo se llama dinárico. Es interesante que, si bien es dominante entre los pueblos balcánicos modernos, este subgrupo aún no ha estado presente durante el período romano, ya que también está casi ausente en Italia (ver Recurso en línea 5; ESM_5). ... Su haplogrupo genético, I2a-CTS10228, está muy extendido entre los eslavos, pero solo está presente en el 7% de los pueblos caucásicos, concretamente entre los karachái ... Como tal, parece que el haplogrupo I2a-CTS10228 en el linaje paterno de los líderes karos surge de una rama específica en el Cáucaso Norte que data de aproximadamente 400-500 d. C. Sus descendientes modernos viven entre los karachái, los húngaros y varias otras nacionalidades circundantes.
Az I2-CTS10228 (köznevén "dinári-kárpáti") alcsoport legkorábbi közös őse 2200 évvel ezelőttre tehető, így esetében nem arról van szó, hogy a mezolit népesség Kelet-Európában ilyen mértékben fennmaradt , hanem arról, hogy egy, a mezolit csoportoktól származó szűk család az európai vaskorban sikeresen integrálódott egy olyan társadalomba, amely hamarosan erőteljes demográfiai expanzióba kezdett. Ez is mutatja, hogy nem Feltétlenül népek, mintsem családok sikerével, nemzetségek elterjedésével is számolnunk kell, és ezt a jelenlegi etnikai identitással összefüggésbe hozni lehetetlen. A csoport elterjedése alapján valószínűsíthető, hogy a szláv népek migrációjában vett részt, így válva az R1a-t követően a második legdominánsabb csoporttá a mai Kelet-Európában. Nyugat-Európából viszont teljes mértékben hiányzik, kivéve a kora középkorban szláv nyelvet beszélő keletnémet területeket.