Los coanoflagelados son un grupo de eucariotas flagelados coloniales y unicelulares de vida libre considerados los parientes vivos más cercanos de los animales . Los coanoflagelados son flagelados con collar, que tienen un collar en forma de embudo de microvellosidades interconectadas en la base de un flagelo . Los coanoflagelados son capaces de reproducción tanto asexual como sexual. [9] Tienen una morfología celular distintiva caracterizada por un cuerpo celular ovoide o esférico de 3 a 10 μm de diámetro con un solo flagelo apical rodeado por un collar de 30 a 40 microvellosidades (ver figura). El movimiento del flagelo crea corrientes de agua que pueden impulsar a los coanoflagelados que nadan libremente a través de la columna de agua y atrapar bacterias y detritos contra el collar de microvellosidades, donde estos alimentos son engullidos. Esta alimentación proporciona un vínculo crítico dentro del ciclo global del carbono , vinculando los niveles tróficos . Además de sus funciones ecológicas críticas, los coanoflagelados son de particular interés para los biólogos evolutivos que estudian los orígenes de la multicelularidad en los animales. Como los parientes vivos más cercanos de los animales, los coanoflagelados sirven como un modelo útil para las reconstrucciones del último ancestro unicelular de los animales. Según un estudio de 2021, los craspédidos del grupo corona (y quizás los coanoflagelados del grupo corona si Acanthoecida surgió dentro de Craspedida [10] ) aparecieron hace 422,78 millones de años, [11] aunque un estudio previo de 2017 recuperó la divergencia de los coanoflagelados del grupo corona (craspédidos) en 786,62 millones de años. [12]
Choanoflagellate es una palabra híbrida del griego χοάνη khoánē que significa " embudo " (debido a la forma del collar) y la palabra latina flagellum (de ahí el término inglés flagellum ). [ cita requerida ]

Cada coanoflagelado tiene un solo flagelo , rodeado por un anillo de protuberancias llenas de actina llamadas microvellosidades , que forman un collar cilíndrico o cónico ( choanos en griego). El movimiento del flagelo atrae agua a través del collar, y las bacterias y los detritos son capturados por las microvellosidades e ingeridos. [13] Las corrientes de agua generadas por el flagelo también empujan a las células que nadan libremente, como en el esperma animal . En contraste, la mayoría de los demás flagelados son arrastrados por sus flagelos. [ cita requerida ]
Además del único flagelo apical rodeado de microvellosidades llenas de actina que caracteriza a los coanoflagelados, la organización interna de los orgánulos en el citoplasma es constante. [14] Un cuerpo basal flagelar se encuentra en la base del flagelo apical, y un segundo cuerpo basal no flagelar descansa en ángulo recto con respecto a la base flagelar. El núcleo ocupa una posición apical-central en la célula, y las vacuolas alimenticias se ubican en la región basal del citoplasma. [14] [15] Además, el cuerpo celular de muchos coanoflagelados está rodeado por una matriz extracelular o periplasto distintivo . Estas cubiertas celulares varían mucho en estructura y composición y son utilizadas por los taxónomos para fines de clasificación. Muchos coanoflagelados construyen "casas" complejas en forma de canasta, llamadas lorica , a partir de varias tiras de sílice cementadas entre sí. [14] Se desconoce la importancia funcional del periplasto, pero en los organismos sésiles se cree que ayuda a la adhesión al sustrato. En los organismos planctónicos, se especula que el periplasto aumenta la resistencia, contrarrestando así la fuerza generada por el flagelo y aumentando la eficiencia de la alimentación. [16]
Los coanoflagelados pueden nadar libremente en la columna de agua o ser sésiles , adhiriéndose al sustrato directamente o a través del periplasto o un pedicelo delgado. [17] Aunque se piensa que los coanoflagelados son estrictamente de vida libre y heterótrofos , varios parientes de los coanoflagelados, como los miembros de Ichthyosporea o Mesomycetozoa , siguen un estilo de vida parasitario o patógeno . [18] Las historias de vida de los coanoflagelados son poco conocidas. Se piensa que muchas especies son solitarias; sin embargo, la colonialidad parece haber surgido de forma independiente varias veces dentro del grupo, y las especies coloniales aún conservan una etapa solitaria. [17]

Se conocen más de 125 especies existentes de coanoflagelados [13] , distribuidas globalmente en ambientes marinos , salobres y de agua dulce desde el Ártico hasta los trópicos, ocupando zonas pelágicas y bentónicas . Aunque la mayoría de los muestreos de coanoflagelados se han producido entre 0 y 25 m (0 y 82 pies), se han recuperado a una profundidad de hasta 300 m (980 pies) en aguas abiertas [19] y 100 m (330 pies) bajo las capas de hielo de la Antártida. [20] Se plantea la hipótesis de que muchas especies son cosmopolitas a escala global [p. ej., se ha informado de Diaphanoeca grandis en América del Norte , Europa y Australia (OBIS)], mientras que se informa que otras especies tienen distribuciones regionales restringidas. [21] Las especies de coanoflagelados codistribuidas pueden ocupar microambientes bastante diferentes, pero en general, los factores que influyen en la distribución y dispersión de los coanoflagelados aún deben dilucidarse. [ cita requerida ]
Varias especies , como las del género Proterospongia , forman colonias simples , [13] grupos planctónicos que se asemejan a un racimo de uvas en miniatura en el que cada célula de la colonia está flagelada o grupos de células en un solo tallo. [14] [22] En octubre de 2019, los científicos descubrieron un nuevo comportamiento de banda de los coanoflagelados: aparentemente pueden coordinarse para responder a la luz. [23]
Los coanoflagelados se alimentan de bacterias y vinculan formas de carbono que de otro modo serían inaccesibles a organismos que se encuentran más arriba en la cadena trófica. [24] Incluso hoy en día, son importantes en el ciclo del carbono y en la red alimentaria microbiana . [13] Hay algunas pruebas de que los coanoflagelados también se alimentan de virus. [25]

Los coanoflagelados crecen vegetativamente, y varias especies experimentan fisión longitudinal; [15] sin embargo, el ciclo de vida reproductivo de los coanoflagelados aún no se ha dilucidado. Un artículo publicado en agosto de 2017 mostró que los cambios ambientales, incluida la presencia de ciertas bacterias, desencadenan la enjambre y la posterior reproducción sexual de los coanoflagelados. [9] Se desconoce el nivel de ploidía ; [27] sin embargo, el descubrimiento de retrotransposones y genes clave involucrados en la meiosis [28] sugirió anteriormente que usaban la reproducción sexual como parte de su ciclo de vida. Algunos coanoflagelados pueden experimentar enquistamiento, que implica la retracción del flagelo y el collar y el encapsulamiento en una pared fibrilar densa en electrones. Al transferirlos a un medio fresco, se produce el desenquistamiento; aunque aún queda por observar directamente. [29]
Se ha informado de evidencia de reproducción sexual en la especie de coanoflagelado Salpingoeca rosetta . [30] [31] También se ha informado de evidencia de la presencia de genes meióticos conservados en los coanoflagelados Monosiga brevicollis y Monosiga ovata . [32]
Los coanoflagelados acantócidos producen una estructura extracelular en forma de cesta conocida como lorica. La lorica está compuesta de tiras costales individuales, hechas de un biocompuesto de sílice y proteína. Cada tira costal se forma dentro de la célula del coanoflagelado y luego se secreta a la superficie celular. En los coanoflagelados nudiformes, el ensamblaje de la lorica se lleva a cabo utilizando una serie de tentáculos una vez que se han producido suficientes tiras costales para formar una lorica completa. En los coanoflagelados tectiformes, las tiras costales se acumulan en una disposición fija debajo del collar. Durante la división celular, la nueva célula toma estas tiras costales como parte de la citocinesis y ensambla su propia lorica utilizando solo estas tiras producidas previamente. [33]
La biosilicificación de los coanoflagelados requiere la concentración de ácido silícico dentro de la célula. Esto se lleva a cabo por las proteínas transportadoras de silicio (SiT). El análisis de las SiT de los coanoflagelados muestra que son similares a los transportadores de silicio de tipo SiT de las diatomeas y otras estramenopilas formadoras de sílice . La familia de genes SiT muestra poca o ninguna homología con otros genes, incluso con genes en coanoflagelados no silíceos o estramenopilas. Esto sugiere que la familia de genes SiT evolucionó a través de un evento de transferencia lateral de genes entre Acanthoecids y Stramenopiles. Este es un caso notable de transferencia horizontal de genes entre dos grupos eucariotas distantemente relacionados, y ha proporcionado pistas sobre la bioquímica y las interacciones silicio-proteína de la familia única de genes SiT. [34]
Félix Dujardin , un biólogo francés interesado en la evolución de los protozoos, registró las similitudes morfológicas de los coanoflagelados y los coanocitos de esponja y propuso la posibilidad de una relación cercana ya en 1841. [16] Durante la última década, esta relación hipotética entre los coanoflagelados y los animales ha sido confirmada por análisis independientes de múltiples secuencias genéticas no vinculadas: 18S rDNA, genes codificantes de proteínas nucleares y genomas mitocondriales (Steenkamp, et al., 2006; Burger, et al., 2003; [18] Wainright, et al., 1993). Es importante destacar que las comparaciones de las secuencias del genoma mitocondrial de un coanoflagelado y tres esponjas confirman la ubicación de los coanoflagelados como un grupo externo a Metazoa (animales, también conocidos como Animalia ) y niegan la posibilidad de que los coanoflagelados evolucionaran a partir de los metazoos (Lavrov, et al., 2005). Finalmente, un estudio de 2001 de los genes expresados en coanoflagelados ha revelado que los coanoflagelados sintetizan homólogos de los genes de adhesión y señalización celular de los metazoos. [35] La secuenciación del genoma muestra que, entre los organismos vivos, los coanoflagelados están más estrechamente relacionados con los animales. [13] Debido a que los coanoflagelados y los metazoos están estrechamente relacionados, las comparaciones entre los dos grupos prometen proporcionar conocimientos sobre la biología de su último ancestro común y los primeros eventos en la evolución de los metazoos . Los coanocitos (también conocidos como "células con collar") de las esponjas (consideradas entre los metazoos más basales) tienen la misma estructura básica que los coanoflagelados. Las células con collar se encuentran en otros grupos animales, como los gusanos cinta , [36] lo que sugiere que esta fue la morfología de su último ancestro común. El último ancestro común de los animales y los coanoflagelados era unicelular, tal vez formando colonias simples; en contraste, el último ancestro común de todos los animales eumetazoos fue un organismo multicelular, con tejidos diferenciados, un "plan corporal" definido y desarrollo embrionario (incluida la gastrulación). [13] El momento de la división de estos linajes es difícil de precisar, pero probablemente fue a fines del Precámbrico, hace más de 600 millones de años . [13]
Relaciones externas de los Choanoflagellatea. [37]
Los coanoflagelados se incluyeron en Chrysophyceae hasta Hibberd, 1975. [38] La reciente reconstrucción filogenética molecular de las relaciones internas de los coanoflagelados permite la polarización de la evolución de los caracteres dentro del clado. Se utilizaron grandes fragmentos de los genes codificantes del ARN ribosómico nuclear SSU y LSU , la alfa tubulina y la proteína de choque térmico 90 para resolver las relaciones internas y la polaridad de los caracteres dentro de los coanoflagelados. [22] Cada uno de los cuatro genes mostró resultados similares de forma independiente y el análisis del conjunto de datos combinados ( concatenados ) junto con secuencias de otras especies estrechamente relacionadas ( animales y hongos ) demuestra que los coanoflagelados están fuertemente respaldados como monofiléticos y confirman su posición como el pariente vivo unicelular conocido más cercano de los animales.
Anteriormente, Choanoflagellida se dividió en estas tres familias en función de la composición y estructura de su periplasto: Codonosigidae, Salpingoecidae y Acanthoecidae. Los miembros de la familia Codonosigidae parecen carecer de periplasto cuando se examinan con microscopio óptico, pero pueden tener una fina capa exterior visible solo con microscopio electrónico . La familia Salpingoecidae consiste en especies cuyas células están encerradas en una teca firme que es visible tanto con microscopio óptico como electrónico. La teca es una cubierta secretada compuesta predominantemente de celulosa u otros polisacáridos . [39] Ahora se sabe que estas divisiones son parafiléticas , con una evolución convergente de estas formas generalizada. La tercera familia de coanoflagelados, Acanthoecidae, ha sido apoyada como un grupo monofilético. Este clado posee una sinapomorfía de las células que se encuentran dentro de una lorica con forma de canasta, lo que proporciona el nombre alternativo de "coanoflagelados loricados". La lorica de Acanthoecid se compone de una serie de tiras costales silíceas dispuestas en un patrón de lorica específico de la especie". [14] [16]
El árbol de coanoflagelados basado en la filogenia molecular se divide en tres clados bien sustentados . [22] El clado 1 y el clado 2 consisten cada uno de una combinación de especies tradicionalmente atribuidas a Codonosigidae y Salpingoecidae, mientras que el clado 3 comprende especies del grupo clasificado taxonómicamente como Acanthoecidae. [22] El mapeo de rasgos de carácter en esta filogenia indica que el último ancestro común de los coanoflagelados fue un organismo marino con un ciclo de vida diferenciado con etapas sedentarias y móviles . [22]

Coanoflagelados; [8]



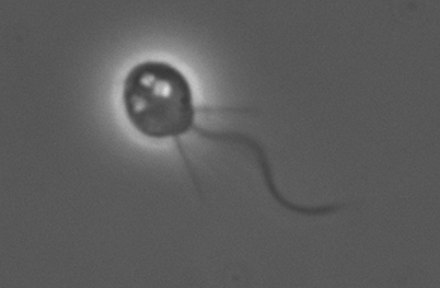

El genoma de Monosiga brevicollis , con 41,6 millones de pares de bases, [13] es similar en tamaño al de los hongos filamentosos y otros eucariotas unicelulares de vida libre, pero mucho más pequeño que el de los animales típicos. [13] En 2010, un estudio filogenómico reveló que varios genes de algas están presentes en el genoma de Monosiga brevicollis . Esto podría deberse al hecho de que, en la historia evolutiva temprana, los coanoflagelados consumían algas como alimento a través de la fagocitosis . [40] Carr et al. (2010) [32] examinaron el genoma de M. brevicollis en busca de genes de meiosis eucariotas conocidos . De los 19 genes meióticos eucariotas conocidos analizados (incluidos 8 que no funcionan en ningún otro proceso que la meiosis), 18 se identificaron en M. brevicollis . La presencia de genes meióticos, incluidos genes específicos de la meiosis, indica que la meiosis y, por implicación, el sexo , están presentes dentro de los coanoflagelados.
El genoma de Salpingoeca rosetta tiene un tamaño de 55 megabases. [41] En el genoma están presentes homólogos de los genes de adhesión celular, neuropéptidos y metabolismo de glucoesfingolípidos. S. rosetta tiene un ciclo de vida sexual y transiciones entre etapas haploides y diploides . [31] En respuesta a la limitación de nutrientes, los cultivos haploides de S. rosetta se vuelven diploides. Este cambio de ploidía coincide con el apareamiento durante el cual las células pequeñas y flageladas se fusionan con células flageladas más grandes. También hay evidencia de apareamiento histórico y recombinación en S. rosetta .
La bacteria marina Vibrio fischeri induce la reproducción sexual de S. rosetta . [30] Una sola proteína de V. fischeri , EroS, recapitula completamente la actividad afrodisíaca de V. fisheri viva .
En 2019 se secuenciaron los genomas amplificados de células individuales de cuatro coanoflagelados marinos no cultivados, denominados provisionalmente UC1–UC4. Los genomas de UC1 y UC4 están relativamente completos. [42]
En 2006 se publicó un conjunto de datos EST de Monosiga ovata . [43] El principal hallazgo de este transcriptoma fue el dominio Hoglet del coanoflagelado y arrojó luz sobre el papel de la redistribución de dominios en la evolución de la vía de señalización Hedgehog . M. ovata tiene al menos cuatro genes meióticos eucariotas. [32]
El transcriptoma de Stephanoeca diplocostata fue publicado en 2013. Este primer transcriptoma de un coanoflagelado loricado [34] condujo al descubrimiento de los transportadores de silicio de los coanoflagelados. Posteriormente, se identificaron genes similares en una segunda especie de loricado, Diaphanoeca grandis . El análisis de estos genes encontró que los transportadores de silicio de los coanoflagelados muestran homología con los transportadores de silicio de tipo SIT de las diatomeas y han evolucionado a través de la transferencia horizontal de genes .
En 2018 se publicaron 19 transcriptomas adicionales y se encontró una gran cantidad de familias de genes que antes se creía que eran exclusivos de los animales. [44]