

Una cápside es la cubierta proteica de un virus , que encierra su material genético . Consiste en varias subunidades estructurales oligoméricas (repetidas) hechas de proteínas llamadas protómeros . Las subunidades morfológicas tridimensionales observables, que pueden corresponder o no a proteínas individuales, se denominan capsómeros . Las proteínas que forman la cápside se denominan proteínas de la cápside o proteínas de la cubierta viral ( VCP ). La cápside y el genoma interno se denominan nucleocápside .
Las cápsides se clasifican en líneas generales según su estructura. La mayoría de los virus tienen cápsides con estructura helicoidal o icosaédrica [2] [3] . Algunos virus, como los bacteriófagos , han desarrollado estructuras más complicadas debido a limitaciones de elasticidad y electrostática. [4] La forma icosaédrica, que tiene 20 caras triangulares equiláteras, se aproxima a una esfera , mientras que la forma helicoidal se asemeja a la forma de un resorte , ocupando el espacio de un cilindro pero sin ser un cilindro en sí misma. [5] Las caras de la cápside pueden constar de una o más proteínas. Por ejemplo, la cápside del virus de la fiebre aftosa tiene caras que constan de tres proteínas llamadas VP1–3. [6]
Algunos virus tienen envoltura , lo que significa que la cápside está recubierta por una membrana lipídica conocida como envoltura viral . La envoltura es adquirida por la cápside de una membrana intracelular en el huésped del virus; algunos ejemplos incluyen la membrana nuclear interna, la membrana de Golgi y la membrana externa de la célula . [7]
Una vez que el virus ha infectado una célula y comienza a replicarse, se sintetizan nuevas subunidades de la cápside utilizando el mecanismo de biosíntesis de proteínas de la célula. En algunos virus, incluidos aquellos con cápsides helicoidales y especialmente aquellos con genomas de ARN, las proteínas de la cápside se ensamblan con sus genomas. En otros virus, especialmente los virus más complejos con genomas de ADN de doble cadena, las proteínas de la cápside se ensamblan en procápsides precursoras vacías que incluyen una estructura de portal especializada en un vértice. A través de este portal, el ADN viral se transloca hacia la cápside. [8]
Los análisis estructurales de las arquitecturas de las proteínas principales de la cápside (MCP) se han utilizado para clasificar los virus en linajes. Por ejemplo, el bacteriófago PRD1, el virus de las algas Paramecium bursaria Chlorella virus-1 (PBCV-1), el mimivirus y el adenovirus de los mamíferos se han colocado en el mismo linaje, mientras que los bacteriófagos de ADN bicatenario con cola ( Caudovirales ) y los herpesvirus pertenecen a un segundo linaje. [9] [10] [11] [12]


La estructura icosaédrica es extremadamente común entre los virus. El icosaedro consta de 20 caras triangulares delimitadas por 12 vértices quíntuples y consta de 60 unidades asimétricas. Por lo tanto, un virus icosaédrico está formado por 60N subunidades proteicas. El número y la disposición de los capsómeros en una cápside icosaédrica se pueden clasificar utilizando el "principio de cuasi-equivalencia" propuesto por Donald Caspar y Aaron Klug . [13] Al igual que los poliedros de Goldberg , una estructura icosaédrica puede considerarse como construida a partir de pentámeros y hexámeros. Las estructuras pueden indexarse mediante dos números enteros h y k , con y ; se puede pensar que la estructura da h pasos desde el borde de un pentámero, gira 60 grados en sentido antihorario y luego da k pasos para llegar al siguiente pentámero. El número de triangulación T para la cápside se define como:
En este esquema, las cápsides icosaédricas contienen 12 pentámeros más 10( T − 1) hexámeros. [14] [15] El número T es representativo del tamaño y la complejidad de las cápsides. [16] Se pueden encontrar ejemplos geométricos para muchos valores de h , k y T en Lista de poliedros geodésicos y Poliedros de Goldberg .
Existen muchas excepciones a esta regla: por ejemplo, los poliomavirus y los papilomavirus tienen pentámeros en lugar de hexámeros en posiciones hexavalentes en una red cuasi T = 7. Los miembros del linaje de virus de ARN bicatenario, incluidos los reovirus , rotavirus y bacteriófagos φ6, tienen cápsides formadas por 120 copias de proteína de cápside, correspondientes a una cápside T = 2, o posiblemente una cápside T = 1 con un dímero en la unidad asimétrica. De manera similar, muchos virus pequeños tienen una cápside pseudo T = 3 (o P = 3), que está organizada de acuerdo con una red T = 3, pero con polipéptidos distintos que ocupan las tres posiciones cuasi equivalentes [17].
Los números T se pueden representar de diferentes maneras, por ejemplo, T = 1 solo se puede representar como un icosaedro o un dodecaedro y, dependiendo del tipo de cuasisimetría, T = 3 se puede presentar como un dodecaedro truncado , un icosidodecaedro o un icosaedro truncado y sus respectivos duales un triakisicosaedro , un triacontaedro rómbico o un pentakisdodecaedro . [18] [ aclaración necesaria ]
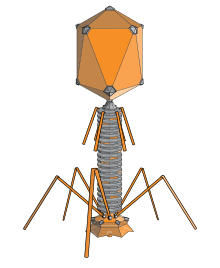
Un icosaedro alargado es una forma común para las cabezas de los bacteriófagos. Dicha estructura está compuesta por un cilindro con un casquete en cada extremo. El cilindro está compuesto por 10 caras triangulares alargadas. El número Q (o T mid ), que puede ser cualquier entero positivo, [19] especifica el número de triángulos, compuestos de subunidades asimétricas, que forman los 10 triángulos del cilindro. Los casquetes se clasifican por el número T (o T end ). [20]
La bacteria E. coli es el huésped del bacteriófago T4 , que tiene una estructura de cabeza alargada. La proteína gp31 codificada por el bacteriófago parece ser funcionalmente homóloga a la proteína chaperona GroES de E. coli y capaz de sustituirla en el ensamblaje de los viriones del bacteriófago T4 durante la infección. [21] Al igual que GroES, gp31 forma un complejo estable con la chaperonina GroEL que es absolutamente necesario para el plegamiento y ensamblaje in vivo de la proteína gp23 de la cápside principal del bacteriófago T4. [21]

Muchos virus vegetales filamentosos y con forma de bastón tienen cápsides con simetría helicoidal . [22] La estructura helicoidal puede describirse como un conjunto de n hélices moleculares unidimensionales relacionadas por una simetría axial de n pliegues. [23] La transformación helicoidal se clasifica en dos categorías: sistemas helicoidales unidimensionales y bidimensionales. [23] La creación de una estructura helicoidal completa se basa en un conjunto de matrices traslacionales y rotacionales que están codificadas en el banco de datos de proteínas. [23] La simetría helicoidal está dada por la fórmula P = μ x ρ , donde μ es el número de unidades estructurales por vuelta de la hélice, ρ es la elevación axial por unidad y P es el paso de la hélice. Se dice que la estructura es abierta debido a la característica de que cualquier volumen puede ser encerrado variando la longitud de la hélice. [24] El virus helicoidal más comprendido es el virus del mosaico del tabaco. [22] El virus es una molécula única de ARN de cadena (+). Cada proteína de la cubierta en el interior de la hélice une tres nucleótidos del genoma de ARN. Los virus de la influenza A se diferencian por estar compuestos por múltiples ribonucleoproteínas, la proteína NP viral organiza el ARN en una estructura helicoidal. El tamaño también es diferente; el virus del mosaico del tabaco tiene 16,33 subunidades proteicas por vuelta helicoidal, [22] mientras que el virus de la influenza A tiene un bucle de cola de 28 aminoácidos. [25]
Las funciones de la cápside son:
El virus debe ensamblar una cubierta proteica estable y protectora para proteger el genoma de agentes químicos y físicos letales, como los extremos de pH o temperatura y las enzimas proteolíticas y nucleolíticas . En el caso de los virus sin envoltura, la propia cápside puede estar involucrada en la interacción con los receptores de la célula huésped, lo que lleva a la penetración de la membrana de la célula huésped y la internalización de la cápside. La entrega del genoma se produce mediante la posterior desprotección o desmontaje de la cápside y la liberación del genoma en el citoplasma, o mediante la expulsión del genoma a través de una estructura portal especializada directamente en el núcleo de la célula huésped.
Se ha sugerido que muchas proteínas de la cápside viral han evolucionado en múltiples ocasiones a partir de proteínas celulares funcionalmente diversas. [26] El reclutamiento de proteínas celulares parece haber ocurrido en diferentes etapas de la evolución, de modo que algunas proteínas celulares fueron capturadas y refuncionalizadas antes de la divergencia de los organismos celulares en los tres dominios contemporáneos de la vida, mientras que otras fueron secuestradas relativamente recientemente. Como resultado, algunas proteínas de la cápside están ampliamente distribuidas en virus que infectan organismos distantemente relacionados (por ejemplo, proteínas de la cápside con el pliegue de rollo de gelatina ), mientras que otras están restringidas a un grupo particular de virus (por ejemplo, proteínas de la cápside de alfavirus). [26] [27]
Un modelo computacional (2015) ha demostrado que las cápsides pueden haberse originado antes que los virus y que sirvieron como medio de transferencia horizontal entre comunidades replicadoras ya que estas comunidades no podrían sobrevivir si el número de genes parásitos aumentaba, siendo ciertos genes los responsables de la formación de estas estructuras y los que favorecían la supervivencia de las comunidades autorreplicantes. [28] El desplazamiento de estos genes ancestrales entre organismos celulares podría favorecer la aparición de nuevos virus durante la evolución. [27]