El SARS-CoV-2 , el agente causal de la COVID-19 , se introdujo por primera vez a los humanos a través de una zoonosis (transmisión de un patógeno a un humano desde un animal), y un evento de contagio zoonótico es el origen de la COVID-19 que la comunidad científica considera más plausible. [a] Los coronavirus humanos , incluido el SARS-CoV-2, son enfermedades zoonóticas que a menudo se adquieren a través de una infección de contagio de animales. [2]
La aparición previa del SARS-CoV-1 y el MERS-CoV mostró que los betacoronavirus representan un riesgo de aparición de enfermedades que amenazan a los humanos. [3] [4] La mayor conciencia debido al brote de SARS de 2002-2004 motivó la investigación sobre el potencial de otros brotes de coronavirus y los reservorios animales que podrían conducir a ellos. [5] Se sabe que los murciélagos, en particular, albergan poblaciones persistentes de coronavirus y, en condiciones de infección persistente, los coronavirus tienden a acumular mutaciones que permiten que sus dominios de unión al receptor interactúen con ortólogos de especies cruzadas de los receptores objetivo . [6] Con base en estudios serológicos y moleculares, los murciélagos de herradura chinos fueron identificados como el reservorio más probable para el SARS-CoV-1. [7] Los murciélagos también fueron un reservorio probable para el betacoronavirus relacionado MERS-CoV , aunque la evidencia de esto es menos concluyente que el papel de los camellos como reservorio para el MERS. [8] En 2010, los experimentos in vitro habían confirmado que las modificaciones en el dominio de unión del receptor de la proteína de la espícula podrían permitir la infección humana por varios coronavirus relacionados con el SARS. [9] Los virólogos Rachel Graham y Ralph S. Baric escribieron en ese momento que "la cuestión de la aparición de otro coronavirus humano patógeno a partir de reservorios de murciélagos podría expresarse de manera más apropiada como 'cuándo' que como 'si'". [10] [ se necesita una mejor fuente ]
El origen probable del SARS-CoV-2 de los murciélagos se alinea con los orígenes de otros coronavirus de su género Betacoronavirus , subgénero Sarbecovirus . [11] [12] [13] [14] Varias especies de murciélagos tienen mecanismos celulares especiales para resistir las citocinas proinflamatorias asociadas con la virulencia del Betacoronavirus , [15] por ejemplo, las proteínas de pico en los coronavirus relacionados con el SARS coevolucionan con los receptores ACE2 de murciélago en una carrera armamentista evolutiva. [16]

Los murciélagos, junto con sus virus, tienen grandes rangos geográficos superpuestos en el sudeste asiático, [17] y hay una concentración y diversidad particularmente grandes de coronavirus relacionados con los murciélagos en el sur y suroeste de China. [15] Los virus conocidos más similares al SARS-CoV-2 incluyen los coronavirus de murciélago RpYN06 con un 94,5% de identidad, [15] y RmYN02 con un 93% de identidad . [18] RaTG13 no fue el progenitor directo del SARS-CoV-2. [19] Temmam et al. no encontró evidencia serológica de exposición a BANAL-52 entre los manipuladores de murciélagos y los recolectores de guano en el área de Laos donde se muestreó. [20] Lytras et al. escribió que "el SARS-CoV-2 se puede rastrear inequívocamente hasta los murciélagos de herradura". [21] Estimaron que los ancestros comunes más recientes del SARS-CoV-2 , RmYN02 y RaTG13, divergieron hace 40 y 50 años, respectivamente. [22]
El dominio de unión al receptor de la proteína de la espícula del SARS-CoV-2 tiene una inserción de aminoácidos entre sus subunidades S1 y S2. [23] Entre los sarbecovirus, solo el SARS-CoV-2 y el RmYN02 tienen dicha inserción, lo que sugiere diferencias en las especies reservorio, los huéspedes intermediarios o la vía evolutiva. [18] El motivo de unión al receptor es la porción del SARS-CoV-2 que más se aleja de RaTG13. [24] El dominio de unión al receptor del SARS-CoV-2 es más similar al de los coronavirus de pangolín . [18] Los virus, incluido el BANAL-52 aislado de murciélagos en Laos, mostraron una alta similitud con el dominio de unión al receptor del SARS-CoV-2 en los residuos de aminoácidos, pero menos del 76,4 % de identidad de nucleótidos en toda la proteína de la espícula. [2] La unión observada del ácido N-acetilneuramínico por el NTD [25] de la proteína de la espiga y la pérdida de esa unión a través de la mutación del bolsillo de unión de azúcar correspondiente en variantes emergentes de interés ha sugerido un papel potencial para la unión transitoria de azúcar en la zoonosis del SARS-CoV-2, en consonancia con propuestas evolutivas anteriores. [26]
Dentro del género Betacoronavirus , los sitios de escisión de furina son comunes en los subgéneros Merbecovirus y Embecovirus . [27] Los sitios de escisión de furina han evolucionado independientemente seis veces en diferentes clados de Betacoronavirus. [27] Los sitios de escisión de furina también evolucionaron en Alphacoronavirus y Gammacoronavirus independientemente de Betacoronavirus. [27]

La escisión de furina contribuye en gran medida a la transmisibilidad y patogenicidad del SARS-CoV-2 en humanos. [28] Las variantes del SARS-CoV-2 que carecen del sitio de escisión de furina son transmisibles entre humanos, pero de manera mucho menos efectiva. [29] El sitio de escisión de furina a veces se describe como "polibásico" debido a su motivo particular de aminoácidos básicos . [28] El SARS-CoV-2 comparte la identidad de aminoácidos con un sitio de escisión de furina de la subunidad α del ENaC humano . [30] [31] [b] El ENaC humano es idéntico solo al de unos pocos grandes simios y Pipistrellus kuhli . [32]
El SARS-CoV-2 también se distingue entre los coronavirus humanos por tener un único gen ORF8 intacto en lugar de subunidades "a" y "b". [1]
El SARS-CoV-2 tiene un rango de hospedadores más amplio en comparación con el SARS-CoV-1 y el MERS-CoV. [33] [34] El SARS-CoV-2 (junto con el SARS-CoV-1 y el MERS-CoV) son virus generalistas, no específicamente adaptados a los humanos, lo que significa que tienen potencial para propagarse a muchas especies y establecer nuevos reservorios naturales después de cambios evolutivos adaptativos. [35]
Dentro de un solo huésped, surgen diversas variaciones de un solo nucleótido a través de mutaciones aleatorias y deriva genética que dan lugar a cuasiespecies virales . [36] El SARS-CoV-2 muta a un ritmo más lento que el típico de los virus de ARN. [36] El principal impulsor de mutación derivado del huésped es la edición por proteínas APOBEC . [36] La selección negativa por procesos inmunes del huésped causa una evolución convergente hacia el escape inmunológico. [36] La persistencia de la infección está correlacionada con la diversidad de cuasiespecies, pero se desconoce la dirección de la causalidad para esto. [36]
La susceptibilidad real del huésped puede diferir significativamente de las predicciones in silico . [35] [37] El proceso de adaptación del huésped se ha estudiado en ratones humanizados , así como mediante la generación de cepas virales adaptadas a ratones a través de pases en serie . [38] Los ratones de tipo salvaje solo son débilmente susceptibles a la cepa original del SARS-CoV-2. [38]
En comparación con otros virus de ARN monocatenario , los coronavirus tienen una mayor tendencia a sufrir recombinación genética , lo que les permite intercambiar material genético con parientes cercanos que coinfectan el mismo organismo. [39] Se cree que el origen del SARS-CoV-1 involucra múltiples eventos de recombinación. [40] La recombinación entre cepas del SARS-CoV-1 es común. [41] Las filogenias inferidas de los coronavirus relacionados con el SARS2 podrían explicarse por la recombinación. [42] Se ha informado de la recombinación entre varias variantes preocupantes del SARS-CoV-2. [43]

Lytras et al. identificaron el marco de lectura abierto de la espícula del SARS-CoV-2 como un punto crítico de recombinación. [44] Especulan que el genoma del SARS-CoV-2 puede involucrar eventos de recombinación repetidos que sobreimprimen regiones que ya eran productos de la recombinación. [45] Temmam et al. escribieron que debido a la difusión limitada de los virus de murciélago en mamíferos, la coinfección requerida para la recombinación en mamíferos era poco probable. [46] Por lo tanto, consideraron más probable que el sitio de escisión de furina surgiera en un reservorio de murciélago antes del desbordamiento.
Después de la transmisión entre especies de un virus, se espera una rápida evolución y selección positiva . [47] Varios estudios encontraron solo signos débiles de evolución adaptativa al principio de la pandemia de COVID-19. [c] Kang et al. escribieron que el SARS-CoV-2 había exhibido relativamente poca variación genética para 2021. [47] Tai et al. escribieron que la expansión de la población en lugar de la selección positiva explicó el espectro de frecuencia de mutación durante la pandemia temprana. [49] Cagliani et al. escribieron que el genoma del SARS-CoV-2 en general muestra evidencia de una selección purificadora "fuerte a moderada" . [50] Los marcos de lectura abiertos accesorios , especialmente ORF8, mostraron una selección débil a neutral. La falta general de selección positiva durante el brote temprano de SARS-CoV-2 contrastó con el curso evolutivo del SARS-CoV-1. [51]
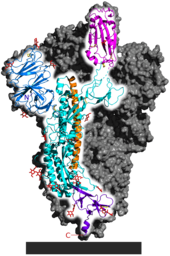
Sin embargo, se encontró evidencia sólida de selección positiva en la subunidad S1 de la proteína de pico, que contiene el dominio de unión al receptor. [24] [52]
La inestabilidad del genoma en la proteína de la espiga es típica de los coronavirus en general, lo que favorece la producción de numerosas variantes de la proteína de la espiga. [43] El dominio de unión al receptor es un factor significativo en el tropismo del huésped , o la variedad de especies que un virus puede infectar. [53] [54] La adaptación del coronavirus a un nuevo huésped a menudo requiere mutaciones en el dominio de unión al receptor. [55] Kang et al. identificaron un polimorfismo de un solo nucleótido relativo a RaTG13 en la proteína de la espiga, consistente entre más de 180.000 muestras de SARS-CoV-2, que afecta la glicosilación del dominio de unión al receptor. [56] Utilizando un sistema de genética inversa para generar un mutante de tipo ancestral, confirmaron que la supuesta forma ancestral de este SNP era mucho menos transmisible en células humanas. [57]
La mayoría de los sarbecovirus no dependen de ACE2 para entrar en la célula. [58] Guo et al. dividen los sarbecovirus en cuatro clados, siendo el primero los virus respiratorios que utilizan ACE2, incluidos el SARS-CoV-1, el SARS-CoV-2 y el WIV1. En relación con el clado 1, los clados 3 y 4 tienen una deleción de un residuo en el dominio de unión al receptor y una capacidad disminuida para utilizar ACE2. El clado 2 tiene dos deleciones y no interactúa con ACE2. Los clados 2 a 4 son más difíciles de aislar o propagar en cultivos celulares y, en consecuencia, han sido menos estudiados. La infección independiente de ACE2 por sarbecovirus depende de altos niveles de tripsina , una proteína digestiva, en el entorno del huésped. La tripsina puede compensar otras proteasas del huésped faltantes o compatibles. La ubicuidad de la furina en comparación con la tripsina permitiría la ganancia de un sitio de escisión de furina para expandir el tropismo tisular viral. [59] Guo et al. Se identificaron sarbecovirus de clado 1 y 2 en muestras fecales de Rhinolophus , lo que sugiere que ambos tipos se replican naturalmente en el tracto digestivo de los murciélagos. [58] Los murciélagos con infecciones similares al SARS en la naturaleza no mostraron replicación viral en gotitas respiratorias. [60] La transmisión fecal-oral es una ruta alternativa para algunos virus respiratorios. [58] No se ha informado de una mayor incidencia de sarbecovirus en trabajadores que entran en contacto directo con guano. [13] Temmam et al. encontraron que BANAL-236, un virus relacionado con el SARS-CoV-2 aislado de murciélagos en Laos, actúa como un virus entérico en macacos . [61]
A diferencia del SARS-CoV-1, el brote inicial de SARS-CoV-2 se informó en una sola ciudad. [62] La vigilancia epidemiológica china no informó ningún otro brote de neumonía en el otoño de 2019. [63] Graham y Baric escribieron que en el caso del SARS-CoV-1, las poblaciones de virus circularon y se adaptaron a las civetas y los humanos en el transcurso de dos años antes del brote reconocido. [60]
Es posible la transmisión del SARS-CoV-2 de humanos a animales, conocida como zoonosis inversa o antroponosis. [64] Se ha informado de una infección zoonótica inversa o experimental con SARS-CoV-2 en 31 especies animales. [65] No se cree que la vida silvestre desempeñe un papel significativo en la circulación continua del SARS-CoV-2 entre humanos. [66]
.jpg/440px-DSC_0377_(8413679878).jpg)
Se encontraron variantes del SARS-CoV-2 adaptadas al visón circulando simultáneamente entre humanos y visones de granja. [2] Los visones son el único animal en Europa o América del Norte que ha experimentado brotes generalizados de SARS-CoV-2. La transmisión de humanos a visones ha ocurrido varias veces, y en la mayoría de los casos no resultó en un brote sostenido en visones. [51] Se observó evidencia sólida de selección positiva en visones después de la propagación, concentrada en el motivo de unión al receptor. [67]
Se ha documentado la transmisión a los humanos en el caso de visones, hámsteres y gatos. [66] Se sospecha la transmisión a los humanos a través de ciervos. [66] Se ha confirmado la transmisión del SARS-CoV-2 entre gatos domésticos . [68]
Li et al. escribieron que el SARS-CoV-2 puede tener menos capacidad que el SARS-CoV-1 para pasar de humanos a animales. [54]
En el brote de SARS-CoV-1, las civetas palmeras, los perros mapaches, los tejones hurones, los zorros rojos, los gatos domésticos y las ratas de los arrozales fueron posibles vectores. [7] Graham y Baric escribieron que las infecciones humanas y de civeta probablemente se originaron de un progenitor común desconocido. [69] Patrick Berche escribió que las apariciones de SARS-CoV-1 y MERS-CoV parecían ser procesos secuenciales que involucraban huéspedes intermediarios, coinfecciones y recombinación. [70] En contraste con la rápida identificación de huéspedes animales para SARS-CoV-1 y MERS-CoV, no se ha encontrado una fuente animal directa para SARS-CoV-2. [71] Holmes et al. escribieron que la falta de huésped intermediario probablemente se debe a que hasta ahora no se ha probado el animal correcto. [19] Frutos et al. Propusieron que, en lugar de un evento de contagio discreto, el SARS-CoV-2 surgió de acuerdo con un modelo de circulación, que implica una transferencia horizontal repetida entre humanos, murciélagos y otros mamíferos sin establecer reservorios significativos en ninguno de ellos hasta la pandemia. [72]

Los pangolines han sido considerados un posible reservorio del SARS-CoV-2 . [73] Los pangolines a veces se venden en mercados húmedos en China, donde se consideran un manjar culinario y un componente de la medicina tradicional. [13] La mayor similitud de secuencia con el dominio de unión del receptor de la espícula del SARS-CoV-2 se encontró en un coronavirus que infecta a los pangolines de Sunda en la provincia de Guangdong . [33] Los pangolines son frecuentemente contrabandeados a China. [45] Lytras et al. escribieron que, en consonancia con la falta de infecciones reportadas de pangolines en Malasia, es probable que se infectaran después de ser traficados a China. [45]
El dominio de unión del receptor del SARS-CoV-2 comparte más sustituciones sinónimas con un pangolín-CoV que con RaTG13. [74] Sin embargo, el potencial de unión del SARS-CoV-2 a la ECA2 del pangolín es muy bajo. [37] Se ha planteado la hipótesis de que el dominio de unión del receptor del SARS-CoV-2 se origina a partir de la recombinación de la porción relevante del pangolín-CoV con un virus similar a RaTG13. [75]
Los ratones ciervos son muy susceptibles al SARS-CoV-2, lo que los convierte en posibles reservorios o huéspedes intermediarios. [76] Los perros mapaches podrían ser capaces de transmitir el SARS-CoV-2 a otros animales en condiciones similares a las de una granja. [68] Se ha demostrado la transmisión entre perros mapaches en condiciones de laboratorio. [65] Los ciervos de cola blanca son un vector potencial del SARS-CoV-2. [68]
Se ha propuesto que el contagio se produjo en uno o más mercados húmedos de China. [77] Los animales de caza silvestres y semisilvestres se comercializan y consumen habitualmente en China, y esta práctica se ha extendido en las últimas décadas. El estrecho contacto entre animales en condiciones insalubres crea el potencial para que las enfermedades prosperen. En el brote de 2002 en Guangdong, la mayoría de los animales vivos en los mercados mostraron evidencia serológica de exposición al SARS-CoV-1. [70]

Muchos de los primeros casos se asociaron con el mercado de mariscos de Huanan en Wuhan. [21] La primera infección documentada por COVID-19 fue en un trabajador de un puesto de mariscos en el mercado de Huanan. [78] No se han publicado las secuencias de ese paciente; sin embargo, se detectó SARS-CoV-2 perteneciente al linaje B/L en muestras ambientales del puesto del paciente. [78] No se informó de la presencia de murciélagos o pangolines en el mercado entre mayo de 2017 y noviembre de 2019. Los perros mapaches, que se supone que son susceptibles al SARS-CoV-2, estaban en el mercado. [79] Una encuesta realizada por He et al. en 2021 identificó 102 virus que afectan a mamíferos en animales de caza chinos, 65 de los cuales se identificaron entonces por primera vez. [80] No encontraron ningún virus similar al SARS, pero sí encontraron evidencia de la transmisión de un virus similar al MERS de murciélagos a erizos. [80]
Entre el 1 de enero y el 30 de marzo, epidemiólogos del CDC de China recogieron 457 muestras de materia animal, incluidos cadáveres y heces, y recogieron 923 muestras ambientales. Todas las muestras animales dieron negativo para SARS-CoV-2. [81] Se encontró SARS-CoV-2 en 73 muestras ambientales. Se aisló el virus vivo de tres muestras, dos de las cuales provenían de puestos pertenecientes a pacientes conocidos. [81] No se encontró ninguna asociación significativa entre el título del virus ambiental y el tipo de producto vendido en puestos particulares. [82] Se identificó SARS-CoV-2 en los cuatro pozos de alcantarillado del mercado. El drenaje superficial del área circundante se concentró debajo del mercado.
Una muestra ambiental contenía linaje A/S SARS-CoV-2, mientras que todas las demás pertenecían al linaje B/L. [83] La muestra que contenía el linaje A/S también contenía evidencia de humanos y ganado, pero ningún animal salvaje. [84] En general, la vida silvestre, incluidos los perros mapaches, se detectó en niveles muy bajos y en su mayoría se asoció con muestras negativas. [85] Liu et al. escribieron que no había suficiente información para determinar el origen del virus. [85] En particular, escribieron que la evidencia no prueba la presencia de un perro mapache infectado o la ocurrencia de múltiples derrames zoonóticos en el mercado como propusieron Pekar et al.
La Organización Mundial de la Salud define las variantes preocupantes como una variante con evidencia de aumento de transmisibilidad, gravedad o escape inmunológico. [86] Todas las variantes preocupantes hasta ahora evolucionaron independientemente de la cepa original, en lugar de unas de otras. [66]
La evolución de la variante ómicron parece haber sido más rápida que la de otras variantes. [55] Las teorías sobre el origen de la variante ómicron incluyen la circulación a largo plazo entre humanos fuera de las áreas donde se realizó vigilancia genética, la mutación en un individuo inmunodeprimido o la adaptación en una especie animal después de una zoonosis inversa. [87] [88] Se propone que la variante ómicron se originó en ratones. [41] [88]