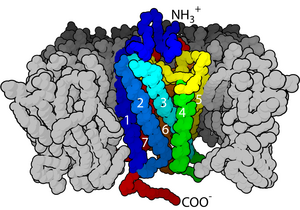
Los receptores de la superficie celular ( receptores de membrana , receptores transmembrana ) son receptores que están incrustados en la membrana plasmática de las células . [1] Actúan en la señalización celular al recibir (unirse a) moléculas extracelulares . Son proteínas integrales de membrana especializadas que permiten la comunicación entre la célula y el espacio extracelular . Las moléculas extracelulares pueden ser hormonas , neurotransmisores , citoquinas , factores de crecimiento , moléculas de adhesión celular o nutrientes ; reaccionan con el receptor para inducir cambios en el metabolismo y la actividad de una célula. En el proceso de transducción de señales , la unión del ligando afecta un cambio químico en cascada a través de la membrana celular.
Muchos receptores de membrana son proteínas transmembrana . Las hay de varios tipos, incluidas las glicoproteínas y las lipoproteínas . [2] Se conocen cientos de receptores diferentes y muchos más aún deben estudiarse. [3] [4] Los receptores transmembrana generalmente se clasifican según su estructura terciaria (tridimensional). Si se desconoce la estructura tridimensional, se pueden clasificar según la topología de la membrana . En los receptores más simples, las cadenas polipeptídicas cruzan la bicapa lipídica una vez, mientras que otras, como los receptores acoplados a proteína G , cruzan hasta siete veces. Cada membrana celular puede tener varios tipos de receptores de membrana, con diferentes distribuciones superficiales. Un único receptor también puede distribuirse de manera diferente en diferentes posiciones de la membrana, según el tipo de membrana y la función celular. Los receptores suelen estar agrupados en la superficie de la membrana, en lugar de estar distribuidos uniformemente. [5] [6]
Se han propuesto dos modelos para explicar el mecanismo de acción de los receptores transmembrana.

Los receptores transmembrana de la membrana plasmática normalmente se pueden dividir en tres partes.
El dominio extracelular está justo fuera de la célula u orgánulo . Si la cadena polipeptídica cruza la bicapa varias veces, el dominio externo comprende bucles entrelazados a través de la membrana. Por definición, la función principal de un receptor es reconocer y responder a un tipo de ligando. Por ejemplo, un neurotransmisor , una hormona o iones atómicos pueden unirse al dominio extracelular como un ligando acoplado a un receptor. Klotho es una enzima que hace que un receptor reconozca el ligando ( FGF23 ).
Las dos clases más abundantes de receptores transmembrana son los GPCR y las proteínas transmembrana de paso único . [8] [9] En algunos receptores, como el receptor nicotínico de acetilcolina , el dominio transmembrana forma un poro proteico a través de la membrana o alrededor del canal iónico . Tras la activación de un dominio extracelular mediante la unión del ligando apropiado, el poro se vuelve accesible a los iones, que luego se difunden. En otros receptores, los dominios transmembrana sufren un cambio conformacional al unirse, lo que afecta las condiciones intracelulares. En algunos receptores, como los miembros de la superfamilia 7TM , el dominio transmembrana incluye un bolsillo de unión a ligando.
El dominio intracelular (o citoplasmático ) del receptor interactúa con el interior de la célula u orgánulo, transmitiendo la señal. Hay dos caminos fundamentales para esta interacción:

Los procesos de transducción de señales a través de receptores de membrana implican reacciones externas, en las que el ligando se une a un receptor de membrana, y reacciones internas, en las que se desencadena una respuesta intracelular. [10] [11]
La transducción de señales a través de receptores de membrana requiere cuatro partes:

Los receptores de membrana se dividen principalmente por estructura y función en 3 clases: el receptor ligado a canales iónicos ; El receptor ligado a enzimas ; y El receptor acoplado a proteína G.
Durante el evento de transducción de señales en una neurona, el neurotransmisor se une al receptor y altera la conformación de la proteína. Esto abre el canal iónico, permitiendo que los iones extracelulares entren en la célula. Se altera la permeabilidad iónica de la membrana plasmática, lo que transforma la señal química extracelular en una señal eléctrica intracelular que altera la excitabilidad celular . [12]
El receptor de acetilcolina es un receptor unido a un canal catiónico. La proteína consta de cuatro subunidades: subunidades alfa (α), beta (β), gamma (γ) y delta (δ). Hay dos subunidades α, con un sitio de unión a acetilcolina cada una. Este receptor puede existir en tres conformaciones. El estado cerrado y desocupado es la conformación de la proteína nativa. Cuando dos moléculas de acetilcolina se unen a los sitios de unión de las subunidades α, la conformación del receptor se altera y se abre la puerta, lo que permite la entrada de muchos iones y moléculas pequeñas. Sin embargo, este estado abierto y ocupado sólo dura un período menor y luego la puerta se cierra, convirtiéndose en el estado cerrado y ocupado. Las dos moléculas de acetilcolina pronto se disociarán del receptor, devolviéndolo al estado original cerrado y desocupado. [13] [14]

En 2009, se conocen seis tipos de receptores ligados a enzimas : receptores tirosina quinasas ; Receptores asociados a tirosina quinasa; Tirosina fosfatasas similares a receptores ; Receptor de serina / treonina quinasas ; Receptores de guanilil ciclasas y receptores asociados a histidina quinasa . Los receptores tirosina quinasas tienen la mayor población y la aplicación más amplia. La mayoría de estas moléculas son receptores de factores de crecimiento como el factor de crecimiento epidérmico (EGF), el factor de crecimiento derivado de plaquetas (PDGF), el factor de crecimiento de fibroblastos (FGF), el factor de crecimiento de hepatocitos (HGF), el factor de crecimiento nervioso (NGF) y hormonas. como la insulina . La mayoría de estos receptores se dimerizarán después de unirse con sus ligandos, para activar más transducciones de señales. Por ejemplo, después de que el receptor del factor de crecimiento epidérmico (EGF) se une a su ligando EGF, los dos receptores se dimerizan y luego sufren la fosforilación de los residuos de tirosina en la porción enzimática de cada molécula receptora. Esto activará la tirosina quinasa y catalizará más reacciones intracelulares.
Los receptores acoplados a proteína G comprenden una gran familia de proteínas de receptores transmembrana. Se encuentran únicamente en eucariotas . [15] Los ligandos que se unen y activan estos receptores incluyen: compuestos fotosensibles, olores , feromonas , hormonas y neurotransmisores . Estos varían en tamaño desde moléculas pequeñas hasta péptidos y proteínas grandes . Los receptores acoplados a la proteína G están implicados en muchas enfermedades y, por tanto, son el objetivo de muchos fármacos modernos. [dieciséis]
Hay dos vías principales de transducción de señales que involucran a los receptores acoplados a proteína G: la vía de señalización del AMPc y la vía de señalización del fosfatidilinositol . [17] Ambos están mediados por la activación de la proteína G. La proteína G es una proteína trimérica, con tres subunidades designadas como α, β y γ. En respuesta a la activación del receptor, la subunidad α libera difosfato de guanosina (GDP) unido, que es desplazado por trifosfato de guanosina (GTP), activando así la subunidad α, que luego se disocia de las subunidades β y γ. La subunidad α activada puede afectar aún más a las proteínas de señalización intracelular o apuntar directamente a proteínas funcionales.
Si los receptores de membrana están desnaturalizados o deficientes, la transducción de señales puede verse obstaculizada y provocar enfermedades. Algunas enfermedades son causadas por trastornos de la función del receptor de membrana. Esto se debe a una deficiencia o degradación del receptor mediante cambios en los genes que codifican y regulan la proteína del receptor. El receptor de membrana TM4SF5 influye en la migración de células hepáticas y hepatoma . [18] Además, el receptor cortical NMDA influye en la fluidez de la membrana y está alterado en la enfermedad de Alzheimer. [19] Cuando la célula es infectada por un virus sin envoltura, el virus primero se une a receptores de membrana específicos y luego pasa él mismo o un componente subviral al lado citoplasmático de la membrana celular. En el caso del poliovirus , se sabe in vitro que las interacciones con los receptores provocan reordenamientos conformacionales que liberan una proteína del virión llamada VP4. El extremo N de VP4 está miristilado y, por tanto, hidrófobo ( ácido mirístico =CH 3 (CH 2 ) 12 COOH). Se propone que los cambios conformacionales inducidos por la unión al receptor dan como resultado la unión del ácido mirístico a VP4 y la formación de un canal para el ARN.

A través de métodos como la cristalografía de rayos X y la espectroscopia de RMN , la información sobre las estructuras 3D de las moléculas objetivo ha aumentado dramáticamente, al igual que la información estructural sobre los ligandos. "Esto impulsa el rápido desarrollo del diseño de fármacos basado en estructuras ". Algunos de estos nuevos fármacos se dirigen a los receptores de membrana. Los enfoques actuales para el diseño de fármacos basado en estructuras se pueden dividir en dos categorías. La primera categoría trata de determinar ligandos para un receptor determinado. Esto generalmente se logra mediante consultas a bases de datos, simulaciones biofísicas y la construcción de bibliotecas químicas. En cada caso, se analiza una gran cantidad de moléculas de ligando potenciales para encontrar aquellas que encajen en el bolsillo de unión del receptor. Este enfoque suele denominarse diseño de fármacos basado en ligandos. La ventaja clave de buscar en una base de datos es que ahorra tiempo y energía para obtener nuevos compuestos eficaces. Otro enfoque de diseño de fármacos basado en estructuras consiste en mapear combinatoriamente ligandos, lo que se conoce como diseño de fármacos basado en receptores. En este caso, las moléculas de ligando se diseñan dentro de las limitaciones de un bolsillo de unión ensamblando piezas pequeñas de manera gradual. Estas piezas pueden ser átomos o moléculas. La ventaja clave de este método es que se pueden descubrir nuevas estructuras. [20] [21] [22]