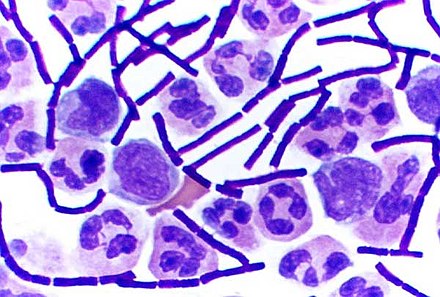

En bacteriología , las bacterias grampositivas son bacterias que dan un resultado positivo en la prueba de tinción de Gram , que se utiliza tradicionalmente para clasificar rápidamente las bacterias en dos amplias categorías según su tipo de pared celular .
Los microbiólogos utilizan la tinción de Gram para clasificar las bacterias en dos categorías principales, Gram positivas (+) y Gram negativas (-). Las bacterias Gram positivas tienen una capa gruesa de peptidoglicano dentro de la pared celular y las bacterias Gram negativas tienen una capa delgada de peptidoglicano.
Las bacterias grampositivas captan la tinción violeta cristal utilizada en la prueba y luego parecen de color púrpura cuando se observan a través de un microscopio óptico . Esto se debe a que la gruesa capa de peptidoglicano en la pared celular bacteriana retiene el tinte después de que se elimina del resto de la muestra, en la etapa de decoloración de la prueba.
Por el contrario, las bacterias gramnegativas no pueden retener la tinción violeta después del paso de decoloración; El alcohol utilizado en esta etapa degrada la membrana externa de las células gramnegativas, haciendo que la pared celular sea más porosa e incapaz de retener la tinción violeta cristal. Su capa de peptidoglicano es mucho más delgada y está intercalada entre una membrana celular interna y una membrana externa bacteriana , lo que hace que absorban la contratinción ( safranina o fucsina ) y aparezcan de color rojo o rosado.
A pesar de su capa de peptidoglicano más gruesa, las bacterias grampositivas son más receptivas a ciertos antibióticos dirigidos a la pared celular que las bacterias gramnegativas, debido a la ausencia de la membrana externa. [1]


En general, las siguientes características están presentes en las bacterias grampositivas: [2]
Sólo algunas especies tienen una cápsula , normalmente formada por polisacáridos . Además, sólo algunas especies son flageladas , y cuando los tienen , sólo tienen dos anillos basales del cuerpo para sostenerlos, mientras que las gramnegativas tienen cuatro. Tanto las bacterias grampositivas como las gramnegativas suelen tener una capa superficial llamada capa S. En las bacterias grampositivas, la capa S está unida a la capa de peptidoglicano. La capa S de las bacterias gramnegativas está unida directamente a la membrana externa . Específica de las bacterias grampositivas es la presencia de ácidos teicoicos en la pared celular. Algunos de ellos son ácidos lipoteicoicos, que tienen un componente lipídico en la membrana celular que puede ayudar a anclar el peptidoglicano. [ cita necesaria ]
Junto con la forma de las células , la tinción de Gram es un método rápido utilizado para diferenciar especies bacterianas. Dicha tinción, junto con las pruebas de requerimiento de crecimiento y susceptibilidad a los antibióticos, y otras pruebas macroscópicas y fisiológicas, forman una base para la clasificación y subdivisión práctica de las bacterias (p. ej., véanse la figura y las versiones anteriores a 1990 del Manual de bacteriología sistemática de Bergey ).

Históricamente , el reino Monera se dividió en cuatro divisiones basadas principalmente en la tinción de Gram: Bacillota (positiva en tinción), Gracilicutes (negativa en tinción), Mollicutes (neutral en tinción) y Mendocutes (variable en tinción). [3] Basado en estudios filogenéticos del ARN ribosómico 16S del fallecido microbiólogo Carl Woese y colaboradores y colegas de la Universidad de Illinois , se cuestionó la monofilia de las bacterias grampositivas, [4] con importantes implicaciones para el estudio terapéutico y general de estos organismos. Basándose en estudios moleculares de las secuencias 16S, Woese reconoció doce filos bacterianos . Dos de ellos eran grampositivos y se dividieron según la proporción del contenido de guanina y citosina en su ADN . El filo de alto G+C estaba formado por Actinobacteria , y el filo de bajo G+C contenía Firmicutes . [4] Los Actinomycetota incluyen los géneros Corynebacterium , Mycobacterium , Nocardia y Streptomyces . La Bacillota (baja G + C) tiene un contenido de GC del 45 al 60%, pero es más bajo que el de Actinomycetota. [2]

Aunque tradicionalmente las bacterias se dividen en dos grupos principales, grampositivas y gramnegativas, según su propiedad de retención en la tinción de Gram, este sistema de clasificación es ambiguo ya que se refiere a tres aspectos distintos (resultado de la tinción, organización de la envoltura, grupo taxonómico), que no necesariamente se fusionan para algunas especies bacterianas. [5] [6] [7] [8] La respuesta de tinción de grampositivas y gramnegativas tampoco es una característica confiable ya que estos dos tipos de bacterias no forman grupos filogenéticamente coherentes. [5] Sin embargo, aunque la respuesta a la tinción de Gram es un criterio empírico, su base radica en las marcadas diferencias en la ultraestructura y composición química de la pared celular bacteriana, marcada por la ausencia o presencia de una membrana lipídica externa. [5] [9]
Todas las bacterias grampositivas están limitadas por una membrana lipídica de una sola unidad y, en general, contienen una capa gruesa (20 a 80 nm) de peptidoglicano responsable de retener la tinción de Gram. Varias otras bacterias, que están limitadas por una sola membrana, pero se tiñen como gramnegativas debido a la falta de la capa de peptidoglicano, como en los micoplasmas , o a su incapacidad para retener la tinción de Gram debido a la composición de su pared celular, también muestran estrecha relación con las bacterias grampositivas. Para las células bacterianas unidas por una membrana celular única, se ha propuesto el término bacteria monodermo . [5] [9]
A diferencia de las bacterias grampositivas, todas las bacterias gramnegativas típicas están limitadas por una membrana citoplasmática y una membrana celular externa; contienen sólo una fina capa de peptidoglicano (2 a 3 nm) entre estas membranas. La presencia de membranas celulares interna y externa define un nuevo compartimento en estas células: el espacio periplásmico o compartimento periplásmico. Estas bacterias han sido designadas como bacterias diderm . [5] [9] La distinción entre las bacterias monodermo y didermo está respaldada por indeles característicos conservados en una serie de proteínas importantes (a saber, DnaK, GroEL). [5] [6] [9] [10] De estos dos grupos de bacterias estructuralmente distintos, se indica que los monodermos son ancestrales. Basándose en una serie de observaciones, entre ellas que las bacterias grampositivas son las principales productoras de antibióticos y que, en general, las bacterias gramnegativas son resistentes a ellos, se ha propuesto que la membrana celular externa de las bacterias gramnegativas (diderms ) ha evolucionado como un mecanismo protector contra la presión de selección de antibióticos . [5] [6] [9] [10] Algunas bacterias, como Deinococcus , que se tiñen como grampositivas debido a la presencia de una capa gruesa de peptidoglicano y también poseen una membrana celular externa, se sugieren como intermediarias en la transición entre monodermo ( bacterias grampositivas) y diderm (gramnegativas). [5] [10] Las bacterias diderm también se pueden diferenciar aún más entre diderms simples que carecen de lipopolisacárido, las bacterias diderm arquetípicas donde la membrana celular externa contiene lipopolisacárido y las bacterias diderm donde la membrana celular externa está formada por ácido micólico . [7] [10] [11]
En general, las bacterias grampositivas son monodermas y tienen una única bicapa lipídica , mientras que las bacterias gramnegativas son didermas y tienen dos bicapas. Las excepciones incluyen:
Algunas especies de Bacillota no son grampositivas. La clase Negativicutes, que incluye Selenomonas , son diderm y se tiñen gramnegativos. [11] Además, se ha descubierto que varios taxones bacterianos (a saber, Negativicutes , Fusobacteriota , Synergistota y Elusimicrobiota ) que forman parte del filo Bacillota o se ramifican en sus proximidades poseen una estructura celular diderma. [8] [10] [11] Sin embargo, una firma indel conservada (CSI) en la proteína HSP60 ( GroEL ) distingue todos los filos tradicionales de bacterias gramnegativas (p. ej., Pseudomonadota , Aquificota , Chlamydiota , Bacteroidota , Chlorobiota , " Cianobacterias " , Fibrobacterota , Verrucomicrobiota , Planctomycetota , Spirochaetota , Acidobacteriota , etc.) de estas otras bacterias diderm atípicas, así como otros filos de bacterias monoderm (p. ej., Actinomycetota , Bacillota , Thermotogota , Chloroflexota , etc.). [10] La presencia de este CSI en todas las especies secuenciadas de filos de bacterias gramnegativas que contienen LPS ( lipopolisacárido ) convencionales proporciona evidencia de que estos filos de bacterias forman un clado monofilético y que no hay pérdida de la membrana externa de ninguna especie de este grupo. ha ocurrido. [10]

En el sentido clásico, seis géneros grampositivos suelen ser patógenos en humanos. Dos de ellos, Streptococcus y Staphylococcus , son cocos (con forma de esfera). Los organismos restantes son bacilos (con forma de bastón) y pueden subdividirse según su capacidad para formar esporas . Los formadores que no son esporas son Corynebacterium y Listeria (un cocobacilo), mientras que Bacillus y Clostridium producen esporas. [16] Las bacterias formadoras de esporas se pueden dividir nuevamente según su respiración : Bacillus es un anaerobio facultativo , mientras que Clostridium es un anaerobio obligado . [17] Además, Rathybacter , Leifsonia y Clavibacter son tres géneros grampositivos que causan enfermedades en las plantas. Las bacterias grampositivas son capaces de provocar infecciones graves y, en ocasiones, mortales en los recién nacidos. [18] Las nuevas especies de bacterias grampositivas clínicamente relevantes también incluyen Catabacter hongkongensis , que es un patógeno emergente perteneciente a Bacillota . [19]
La transformación es uno de los tres procesos de transferencia horizontal de genes , en los que el material genético exógeno pasa de una bacteria donante a una bacteria receptora, siendo los otros dos procesos la conjugación (transferencia de material genético entre dos células bacterianas en contacto directo) y la transducción (inyección de ADN bacteriano donante por un virus bacteriófago en una bacteria huésped receptora). [20] [21] En la transformación, el material genético pasa a través del medio intermedio y la absorción depende completamente de la bacteria receptora. [20]
En 2014, se sabía que alrededor de 80 especies de bacterias eran capaces de transformación, divididas aproximadamente en partes iguales entre bacterias grampositivas y gramnegativas ; la cifra podría ser una sobreestimación, ya que varios de los informes están respaldados por artículos únicos. [20] La transformación entre bacterias grampositivas se ha estudiado en especies de importancia médica como Streptococcus pneumoniae , Streptococcus mutans , Staphylococcus aureus y Streptococcus sanguinis y en bacterias grampositivas del suelo Bacillus subtilis , Bacillus cereus . [22]
Los adjetivos grampositivos y gramnegativos derivan del apellido de Hans Christian Gram ; como adjetivos epónimos , su letra inicial puede ser G mayúscula o g minúscula , dependiendo de qué guía de estilo (por ejemplo, la del CDC ), si la hay, rige el documento que se está escribiendo. [23] Esto se explica con más detalle en Tinción de Gram § Nota ortográfica .