
La edición del genoma , o ingeniería genómica , o edición de genes , es un tipo de ingeniería genética en la que se inserta, elimina, modifica o reemplaza ADN en el genoma de un organismo vivo. A diferencia de las primeras técnicas de ingeniería genética que insertan aleatoriamente material genético en el genoma de un huésped, la edición del genoma dirige las inserciones a ubicaciones específicas del sitio. El mecanismo básico implicado en las manipulaciones genéticas mediante nucleasas programables es el reconocimiento de los loci genómicos diana y la unión del dominio de unión al ADN efector (DBD), las roturas de doble hebra (DSB) en el ADN diana mediante las endonucleasas de restricción ( FokI y Cas ), y la reparación de DSB mediante recombinación dirigida por homología (HDR) o unión de extremos no homólogos (NHEJ). [1] [2]
La edición del genoma fue pionera en la década de 1990, [3] antes de la llegada de las actuales plataformas de edición de genes basadas en nucleasas, pero su uso estaba limitado por la baja eficiencia de la edición. La edición del genoma con nucleasas diseñadas, es decir, las tres clases principales de estas enzimas (nucleasas con dedos de zinc (ZFN), nucleasas efectoras similares a activadores de la transcripción (TALEN) y meganucleasas diseñadas, fueron seleccionadas por Nature Methods como el Método del año 2011. [4] El sistema CRISPR-Cas fue seleccionado por Science como Avance del Año 2015. [5]
A partir de 2015, [actualizar]se utilizaron cuatro familias de nucleasas diseñadas: meganucleasas , nucleasas con dedos de zinc (ZFN), nucleasas basadas en efectores similares a activadores de la transcripción (TALEN) y el sistema de repeticiones palindrómicas cortas agrupadas y regularmente espaciadas ( CRISPR / Cas9 ). [6] [7] [8] [9] Nueve editores de genoma estaban disponibles en 2017 [actualizar]. [10]
En 2018, los métodos comunes para dicha edición utilizaban nucleasas diseñadas o "tijeras moleculares". Estas nucleasas crean roturas de doble hebra (DSB) específicas de un sitio en ubicaciones deseadas del genoma. Las roturas de doble hebra inducidas se reparan mediante unión de extremos no homólogos (NHEJ) o recombinación homóloga (HR), lo que da como resultado mutaciones dirigidas ("ediciones").
En mayo de 2019, abogados en China informaron, a la luz de la supuesta creación por parte del científico chino He Jiankui de los primeros humanos editados genéticamente (ver Controversia de Lulu y Nana ), la redacción de regulaciones que prohibirían a cualquiera que manipulara el genoma humano mediante técnicas de edición genética. , al igual que CRISPR , sería responsable de cualquier consecuencia adversa relacionada. [11] Recientemente se ha debatido una perspectiva cautelosa sobre los posibles puntos ciegos y riesgos de CRISPR y las biotecnologías relacionadas, [12] centrándose en la naturaleza estocástica de los procesos de control celular.
El Instituto Roslin de la Universidad de Edimburgo diseñó cerdos resistentes a un virus que causa el síndrome respiratorio y reproductivo porcino , lo que cuesta a los criadores de cerdos estadounidenses y europeos 2.600 millones de dólares al año. [13]
En febrero de 2020, un ensayo estadounidense demostró de forma segura la edición del gen CRISPR en 3 pacientes con cáncer. [14] En 2020, se aprobó la venta en Japón de Sicilian Rouge High GABA, un tomate que produce más de un aminoácido que se dice que promueve la relajación. [13]
En 2021, Inglaterra (no el resto del Reino Unido) planeó eliminar las restricciones a las plantas y animales editados genéticamente, pasando de una regulación compatible con la Unión Europea a normas más cercanas a las de Estados Unidos y algunos otros países. Un informe de la Comisión Europea de abril de 2021 encontró "fuertes indicios" de que el régimen regulatorio actual no era apropiado para la edición de genes. [13] Más adelante en 2021, los investigadores anunciaron una alternativa CRISPR, proteínas de actividad guiada por elementos móviles obligados (OMEGA), incluidas IscB, IsrB y TnpB, como endonucleasas que se encuentran en transposones y guiadas por pequeños ARN ω. [15] [16]
La ingeniería genética como método para introducir nuevos elementos genéticos en los organismos existe desde los años 1970. Un inconveniente de esta tecnología ha sido la naturaleza aleatoria con la que se inserta el ADN en el genoma del huésped , lo que puede perjudicar o alterar otros genes dentro del organismo. Sin embargo, se han descubierto varios métodos que dirigen los genes insertados a sitios específicos dentro del genoma de un organismo. [3] También ha permitido la edición de secuencias específicas dentro de un genoma, así como la reducción de efectos fuera del objetivo. Esto podría usarse con fines de investigación, apuntando a mutaciones en genes específicos y en terapia génica . Insertando un gen funcional en un organismo y orientándolo para reemplazar el defectuoso, podría ser posible curar ciertas enfermedades genéticas .
Los primeros métodos para dirigir genes a ciertos sitios dentro del genoma de un organismo (llamados orientación genética ) se basaban en la recombinación homóloga (HR). [17] Al crear construcciones de ADN que contienen una plantilla que coincide con la secuencia del genoma objetivo, es posible que los procesos de recursos humanos dentro de la célula inserten la construcción en la ubicación deseada. El uso de este método con células madre embrionarias condujo al desarrollo de ratones transgénicos con genes específicos desactivados . También ha sido posible activar genes o alterar patrones de expresión genética . [18] En reconocimiento a su descubrimiento de cómo se puede utilizar la recombinación homóloga para introducir modificaciones genéticas en ratones a través de células madre embrionarias, Mario Capecchi , Martin Evans y Oliver Smithies recibieron el Premio Nobel de Fisiología o Medicina en 2007 . [19]
Si se elimina un gen vital, puede resultar letal para el organismo. Para estudiar la función de estos genes se utilizaron recombinasas específicas de sitio (SSR). Los dos tipos más comunes son los sistemas Cre-LoxP y Flp-FRT . La recombinasa Cre es una enzima que elimina el ADN mediante recombinación homóloga entre secuencias de unión conocidas como sitios Lox-P. El sistema Flip-FRT funciona de manera similar: la recombinasa Flip reconoce secuencias FRT. Al cruzar un organismo que contiene los sitios de recombinasa que flanquean el gen de interés con un organismo que expresa el SSR bajo el control de promotores específicos de tejido , es posible desactivar o activar genes sólo en ciertas células. Estas técnicas también se utilizaron para eliminar genes marcadores de animales transgénicos. Modificaciones adicionales de estos sistemas permitieron a los investigadores inducir la recombinación solo bajo ciertas condiciones, permitiendo que los genes fueran eliminados o expresados en momentos o etapas de desarrollo deseados . [18]

Una forma común de edición del genoma se basa en el concepto de mecánica de reparación de roturas de doble cadena (DSB) del ADN. Hay dos vías principales que reparan DSB; unión de extremos no homólogos (NHEJ) y reparación dirigida por homología (HDR). NHEJ utiliza una variedad de enzimas para unir directamente los extremos del ADN, mientras que el HDR, más preciso, utiliza una secuencia homóloga como plantilla para la regeneración de secuencias de ADN faltantes en el punto de ruptura. Esto se puede aprovechar creando un vector con los elementos genéticos deseados dentro de una secuencia que sea homóloga a las secuencias flanqueantes de un DSB. Esto dará como resultado que se inserte el cambio deseado en el sitio del DSB. Si bien la edición de genes basada en HDR es similar a la selección de genes basada en recombinación homóloga, la tasa de recombinación aumenta en al menos tres órdenes de magnitud. [20]

La clave para la edición del genoma es crear un DSB en un punto específico del genoma. Las enzimas de restricción comúnmente utilizadas son efectivas para cortar el ADN, pero generalmente reconocen y cortan en múltiples sitios. Para superar este desafío y crear DSB de sitio específico, hasta la fecha se han descubierto y diseñado mediante bioingeniería tres clases distintas de nucleasas. Estas son las nucleasas con dedos de zinc ( ZFN ), las nucleasas efectoras similares a activadoras de la transcripción ( TALEN ), las meganucleasas y el sistema de repeticiones palindrómicas cortas agrupadas y regularmente espaciadas ( CRISPR /Cas9).
Las meganucleasas , descubiertas a finales de los años 1980, son enzimas de la familia de las endonucleasas que se caracterizan por su capacidad de reconocer y cortar grandes secuencias de ADN (de 14 a 40 pares de bases). [21] Las meganucleasas más extendidas y conocidas son las proteínas de la familia LAGLIDADG, que deben su nombre a una secuencia de aminoácidos conservada .
Las meganucleasas, que se encuentran comúnmente en especies microbianas, tienen la propiedad única de tener secuencias de reconocimiento muy largas (>14 pb), lo que las hace naturalmente muy específicas. [22] [23] Sin embargo, prácticamente no existe ninguna posibilidad de encontrar la meganucleasa exacta necesaria para actuar sobre una secuencia de ADN específica elegida. Para superar este desafío, se han utilizado mutagénesis y métodos de detección de alto rendimiento para crear variantes de meganucleasa que reconocen secuencias únicas. [23] [24] Otros han podido fusionar varias meganucleasas y crear enzimas híbridas que reconocen una nueva secuencia. [25] [26] Sin embargo, otros han intentado alterar los aminoácidos de la meganucleasa que interactúan con el ADN para diseñar meganucelasas de secuencia específica en un método llamado meganucleasa diseñada racionalmente. [27] Otro enfoque implica el uso de modelos informáticos para intentar predecir con la mayor precisión posible la actividad de las meganucleasas modificadas y la especificidad de la secuencia nucleica reconocida. [28]
Se ha creado un gran banco que contiene varias decenas de miles de unidades proteicas. Estas unidades se pueden combinar para obtener meganucleasas quiméricas que reconocen el sitio objetivo, proporcionando así herramientas de investigación y desarrollo que satisfacen una amplia gama de necesidades (investigación fundamental, salud, agricultura, industria, energía, etc.). Estas incluyen la producción a escala industrial. de dos meganucleasas capaces de escindir el gen XPC humano; Las mutaciones en este gen dan como resultado Xeroderma pigmentosum , un trastorno monogénico grave que predispone a los pacientes a sufrir cáncer de piel y quemaduras cuando su piel se expone a los rayos ultravioleta. [29]
Las meganucleasas tienen la ventaja de causar menos toxicidad en las células que métodos como la nucleasa con dedos de zinc (ZFN), probablemente debido a un reconocimiento de secuencia de ADN más estricto; [23] sin embargo, la construcción de enzimas específicas de secuencia para todas las secuencias posibles es costosa y requiere mucho tiempo, ya que no se benefician de las posibilidades combinatorias que utilizan métodos como ZFN y fusiones basadas en TALEN.
A diferencia de las meganucleasas, el concepto detrás de la tecnología ZFN y TALEN se basa en un dominio catalítico de corte de ADN no específico, que luego puede vincularse a una secuencia de ADN específica que reconoce péptidos como los dedos de zinc y los efectores similares a activadores de la transcripción (TALE). [30] El primer paso para esto fue encontrar una endonucleasa cuyo sitio de reconocimiento de ADN y sitio de escisión estuvieran separados entre sí, una situación que no es la más común entre las enzimas de restricción. [30] Una vez que se encontró esta enzima, su porción de escisión podría separarse, lo que sería muy inespecífico ya que no tendría capacidad de reconocimiento. Esta porción podría luego vincularse a péptidos que reconozcan secuencias, lo que podría conducir a una especificidad muy alta.
Los motivos de dedos de zinc aparecen en varios factores de transcripción . El ion zinc, que se encuentra en el 8% de todas las proteínas humanas, desempeña un papel importante en la organización de su estructura tridimensional. En los factores de transcripción, se localiza con mayor frecuencia en los sitios de interacción proteína-ADN, donde estabiliza el motivo. La parte C-terminal de cada dedo es responsable del reconocimiento específico de la secuencia de ADN.
Las secuencias reconocidas son cortas, compuestas por alrededor de 3 pares de bases, pero combinando de 6 a 8 dedos de zinc cuyos sitios de reconocimiento han sido caracterizados, es posible obtener proteínas específicas para secuencias de alrededor de 20 pares de bases. Por tanto, es posible controlar la expresión de un gen específico. Se ha demostrado que esta estrategia puede utilizarse para promover un proceso de angiogénesis en animales. [31] También es posible fusionar una proteína construida de esta manera con el dominio catalítico de una endonucleasa para inducir una rotura específica del ADN y, por lo tanto, utilizar estas proteínas como herramientas de ingeniería genómica. [32]
El método generalmente adoptado para ello consiste en asociar dos proteínas de unión al ADN, cada una de las cuales contiene de 3 a 6 dedos de zinc específicamente elegidos, con el dominio catalítico de la endonucleasa FokI , que necesita dimerizarse para escindir el ADN de doble hebra. Las dos proteínas reconocen dos secuencias de ADN que están separadas por unos pocos nucleótidos. La vinculación de las dos proteínas con dedos de zinc a sus respectivas secuencias acerca los dos dominios FokI. FokI requiere dimerización para tener actividad nucleasa y esto significa que la especificidad aumenta dramáticamente ya que cada socio de nucleasa reconocería una secuencia de ADN única. Para mejorar este efecto, se han diseñado nucleasas FokI que solo pueden funcionar como heterodímeros. [33]
Se utilizan varios enfoques para diseñar nucleasas con dedos de zinc específicas para las secuencias elegidas. El más extendido consiste en combinar unidades de dedos de zinc con especificidades conocidas (montaje modular). Se han desarrollado diversas técnicas de selección, utilizando bacterias, levaduras o células de mamíferos, para identificar las combinaciones que ofrecen la mejor especificidad y la mejor tolerancia celular. Aunque no se ha informado sobre la caracterización directa de todo el genoma de la actividad de la nucleasa con dedos de zinc, un ensayo que mide el número total de roturas del ADN de doble hebra en las células encontró que solo se producen una o dos de esas roturas por encima del fondo en células tratadas con nucleasas con dedos de zinc. con un sitio de reconocimiento compuesto de 24 pb y dominios de nucleasa FokI de heterodímero obligado . [33]
Las nucleasas que funcionan con heterodímeros evitarían la posibilidad de actividad homodímera no deseada y, por tanto, aumentarían la especificidad de la DSB. Aunque las porciones de nucleasa de las construcciones ZFN y TALEN tienen propiedades similares, la diferencia entre estas nucleasas diseñadas está en su péptido de reconocimiento de ADN. Los ZFN se basan en dedos de zinc Cys2-His2 y construcciones TALEN en TALE. Ambos dominios peptídicos que reconocen ADN tienen la característica de que se encuentran naturalmente en combinaciones en sus proteínas. Los dedos de zinc Cys2-His2 generalmente ocurren en repeticiones que están separadas por 3 pb y se encuentran en diversas combinaciones en una variedad de proteínas que interactúan con ácidos nucleicos, como los factores de transcripción . Cada dedo del dominio de dedos de zinc es completamente independiente y la capacidad de unión de un dedo se ve afectada por su vecino. Los TALE, por otro lado, se encuentran en repeticiones con una relación de reconocimiento de uno a uno entre los aminoácidos y los pares de nucleótidos reconocidos. Debido a que tanto los dedos de zinc como los TALE ocurren en patrones repetidos, se pueden probar diferentes combinaciones para crear una amplia variedad de especificidades de secuencia. [22] Los dedos de zinc se han establecido más en estos términos y enfoques como el ensamblaje modular (donde los dedos de zinc correlacionados con una secuencia triplete se unen en una fila para cubrir la secuencia requerida), OPEN (selección de dominios peptídicos de baja rigurosidad vs. nucleótidos tripletes seguidos de selecciones altamente rigurosas de combinación de péptidos versus el objetivo final en sistemas bacterianos), y la detección bacteriana de un híbrido de bibliotecas de dedos de zinc, entre otros métodos, se han utilizado para crear nucleasas específicas de sitio.
Las nucleasas con dedos de zinc son herramientas de investigación y desarrollo que ya se han utilizado para modificar una variedad de genomas, en particular por los laboratorios del Zinc Finger Consortium. La empresa estadounidense Sangamo BioSciences utiliza nucleasas de dedos de zinc para investigar la ingeniería genética de células madre y la modificación de células inmunitarias con fines terapéuticos. [34] [35] Los linfocitos T modificados se encuentran actualmente en ensayos clínicos de fase I para tratar un tipo de tumor cerebral ( glioblastoma ) y en la lucha contra el SIDA. [33]

Las nucleasas efectoras similares a activadores de la transcripción (TALEN) son proteínas de unión al ADN específicas que presentan una serie de repeticiones de 33 o 34 aminoácidos. Las TALEN son enzimas de restricción artificiales diseñadas fusionando el dominio de corte de ADN de una nucleasa con dominios TALE, que pueden adaptarse para reconocer específicamente una secuencia de ADN única. Estas proteínas de fusión sirven como "tijeras de ADN" fácilmente seleccionables para aplicaciones de edición de genes que permiten realizar modificaciones específicas del genoma, como inserción, eliminación, reparación y reemplazo de secuencias en células vivas. [36] Los dominios de unión al ADN, que pueden diseñarse para unirse a cualquier secuencia de ADN deseada, provienen de efectores TAL , proteínas de unión al ADN excretadas por la aplicación Xantomanos patógena de plantas. Los efectores TAL constan de dominios repetidos, cada uno de los cuales contiene una secuencia altamente conservada de 34 aminoácidos y reconocen un único nucleótido de ADN dentro del sitio objetivo. La nucleasa puede crear roturas de doble cadena en el sitio objetivo que pueden repararse mediante uniones de extremos no homólogos (NHEJ) propensas a errores, lo que resulta en alteraciones genéticas mediante la introducción de pequeñas inserciones o eliminaciones. Cada repetición se conserva, con la excepción de los llamados residuos variables de repetición (RVD) en las posiciones de los aminoácidos 12 y 13. Los RVD determinan la secuencia de ADN a la que se unirá TALE. Esta simple correspondencia uno a uno entre las repeticiones de TALE y la secuencia de ADN correspondiente hace que el proceso de ensamblar matrices de repeticiones para reconocer nuevas secuencias de ADN sea sencillo. Estos TALE se pueden fusionar con el dominio catalítico de una ADN nucleasa, FokI, para generar una nucleasa efectora similar a un activador de la transcripción (TALEN). Las construcciones TALEN resultantes combinan especificidad y actividad, generando de manera efectiva nucleasas específicas de secuencia diseñadas que se unen y escinden secuencias de ADN solo en sitios preseleccionados. El sistema de reconocimiento de objetivos TALEN se basa en un código fácil de predecir. Las nucleasas TAL son específicas de su objetivo debido en parte a la longitud de su sitio de unión de más de 30 pares de bases. TALEN se puede realizar dentro de un rango de 6 pares de bases de cualquier nucleótido en todo el genoma. [37]
Las construcciones TALEN se utilizan de manera similar a las nucleasas de dedos de zinc diseñadas y tienen tres ventajas en la mutagénesis dirigida: (1) la especificidad de unión al ADN es mayor, (2) los efectos fuera del objetivo son menores y (3) la construcción de estructuras de unión al ADN dominios es más fácil.
Las CRISPR (repeticiones palindrómicas cortas agrupadas y regularmente interespaciadas) son elementos genéticos que las bacterias utilizan como una especie de inmunidad adquirida para protegerse contra los virus. Consisten en secuencias cortas que se originan a partir de genomas virales y se han incorporado al genoma bacteriano. Cas (proteínas asociadas a CRISPR) procesa estas secuencias y corta las secuencias de ADN viral correspondientes. Al introducir plásmidos que contienen genes Cas y CRISPR construidos específicamente en células eucariotas, el genoma eucariota se puede cortar en cualquier posición deseada. [38]
Uno de los primeros métodos para editar ácidos nucleicos de manera eficiente, que emplea enzimas modificadoras de nucleobases dirigidas por secuencias guía de ácidos nucleicos, se describió por primera vez en la década de 1990 y ha resurgido más recientemente. [3] [39] [40] [41] Este método tiene la ventaja de que no requiere romper las cadenas de ADN genómico y, por lo tanto, evita la inserción aleatoria y las eliminaciones asociadas con la rotura de las cadenas de ADN. Solo es apropiado para la edición precisa que requiere cambios de un solo nucleótido y ha demostrado ser muy eficiente para este tipo de edición. [41] [42]
ARCUT significa cortador de ADN de restricción artificial, es una técnica desarrollada por Komiyama. Este método utiliza ácido nucleico peptídico pseudocomplementario (pcPNA) para identificar el sitio de escisión dentro del cromosoma. Una vez que pcPNA especifica el sitio, la escisión se lleva a cabo con cerio (CE) y EDTA (mezcla química), que realiza la función de empalme. [43]
El método de edición de genes con meganucleasas es el menos eficiente de los métodos mencionados anteriormente. Debido a la naturaleza de su elemento de unión al ADN y del elemento de escisión, se limita a reconocer un objetivo potencial cada 1.000 nucleótidos. [9] ZFN fue desarrollado para superar las limitaciones de la meganucleasa. El número de posibles objetivos que ZFN puede reconocer se incrementó a uno de cada 140 nucleótidos. [9] Sin embargo, ambos métodos son impredecibles debido a que sus elementos de unión al ADN se afectan entre sí. Como resultado, se requieren altos grados de experiencia y procesos de validación largos y costosos.
Las nucleasas TALE, al ser el método más preciso y específico, producen una mayor eficiencia que los dos métodos anteriores. Logra tal eficiencia porque el elemento de unión al ADN consta de una serie de subunidades TALE, cada una de las cuales tiene la capacidad de reconocer una cadena de nucleótidos de ADN específica independientemente de las demás, lo que da como resultado un mayor número de sitios objetivo con alta precisión. Se necesitan aproximadamente una semana y unos cientos de dólares para crear nuevas nucleasas TALE, con experiencia específica en biología molecular e ingeniería de proteínas. [9]
Las nucleasas CRISPR tienen una precisión ligeramente menor en comparación con las nucleasas TALE. Esto se debe a la necesidad de tener un nucleótido específico en un extremo para producir el ARN guía que CRISPR utiliza para reparar la rotura de doble cadena que induce. Se ha demostrado que es el método más rápido y económico, ya que solo cuesta menos de doscientos dólares y unos pocos días de tiempo. [9] CRISPR también requiere la menor cantidad de experiencia en biología molecular, ya que el diseño reside en el ARN guía en lugar de las proteínas. Una ventaja importante que tiene CRISPR sobre los métodos ZFN y TALEN es que puede dirigirse para apuntar a diferentes secuencias de ADN utilizando sus sgRNA CRISPR de ~80 nt, mientras que tanto los métodos ZFN como TALEN requirieron la construcción y prueba de las proteínas creadas para apuntar a cada secuencia de ADN. [44]
Debido a que la actividad fuera del objetivo de una nucleasa activa tendría consecuencias potencialmente peligrosas a nivel genético y del organismo, la precisión de las fusiones basadas en meganucleasas, ZFN, CRISPR y TALEN ha sido un área activa de investigación. Si bien se han informado cifras variables, las ZFN tienden a tener más citotoxicidad que los métodos TALEN o las nucleasas guiadas por ARN, mientras que los enfoques TALEN y guiados por ARN tienden a tener la mayor eficiencia y menos efectos fuera del objetivo. [45] Basado en la distancia teórica máxima entre la unión al ADN y la actividad nucleasa, los enfoques TALEN dan como resultado la mayor precisión. [9]
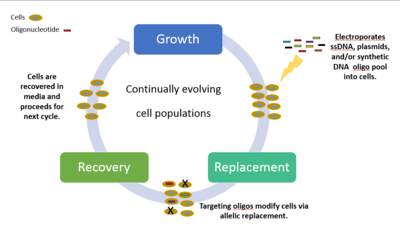
Los métodos para los científicos e investigadores que querían estudiar la diversidad genómica y todos los posibles fenotipos asociados eran muy lentos, costosos e ineficientes. Antes de esta nueva revolución, los investigadores tendrían que realizar manipulaciones de un solo gen y modificar el genoma una pequeña sección a la vez, observar el fenotipo y comenzar el proceso de nuevo con una manipulación de un solo gen diferente. [46] Por lo tanto, investigadores del Instituto Wyss de la Universidad de Harvard diseñaron MAGE, una poderosa tecnología que mejora el proceso de edición del genoma in vivo. Permite manipulaciones rápidas y eficientes de un genoma, todo ello en una máquina lo suficientemente pequeña como para colocarla encima de una pequeña mesa de cocina. Esas mutaciones se combinan con la variación que ocurre naturalmente durante la mitosis celular creando miles de millones de mutaciones celulares.
Se introducen ADN monocatenario (ssDNA) sintético combinado químicamente y un conjunto de oligonucleótidos en áreas específicas de la célula, creando así modificaciones genéticas. El proceso cíclico implica la transformación del ADNss (mediante electroporación ) seguida de un crecimiento, durante el cual las proteínas de recombinación homólogas de bacteriófagos median la hibridación de los ADNss con sus objetivos genómicos. Los experimentos dirigidos a marcadores fenotípicos selectivos se analizan e identifican colocando las células en medios diferenciales. En última instancia, cada ciclo tarda 2,5 horas en procesarse, y se requiere tiempo adicional para desarrollar cultivos isogénicos y caracterizar mutaciones. Al introducir de forma iterativa bibliotecas de ADNss mutagénicos dirigidos a múltiples sitios, MAGE puede generar diversidad genética combinatoria en una población celular. Puede haber hasta 50 ediciones del genoma, desde pares de bases de nucleótidos individuales hasta el genoma completo o redes de genes simultáneamente, con resultados en cuestión de días. [46]
Los experimentos MAGE se pueden dividir en tres clases, caracterizadas por diversos grados de escala y complejidad: (i) muchos sitios objetivo, mutaciones genéticas únicas; (ii) sitio objetivo único, muchas mutaciones genéticas; y (iii) muchos sitios objetivo, muchas mutaciones genéticas. [46] Un ejemplo de la clase tres se reflejó en 2009, donde Church y sus colegas pudieron programar Escherichia coli para producir cinco veces la cantidad normal de licopeno, un antioxidante que normalmente se encuentra en las semillas de tomate y está relacionado con propiedades anticancerígenas. Aplicaron MAGE para optimizar la vía metabólica de la 1-desoxi- D -xilulosa 5-fosfato (DXP) en Escherichia coli para producir en exceso licopeno isoprenoide. Les tomó alrededor de 3 días y poco más de $1,000 en materiales. La facilidad, velocidad y rentabilidad con la que MAGE puede alterar los genomas puede transformar la forma en que las industrias abordan la fabricación y producción de compuestos importantes en las industrias de bioingeniería, bioenergía, ingeniería biomédica, biología sintética, farmacéutica, agrícola y química.

En 2012, se había desarrollado una edición genómica eficiente para una amplia gama de sistemas experimentales, desde plantas hasta animales, a menudo más allá del interés clínico, y se estaba convirtiendo en una estrategia experimental estándar en los laboratorios de investigación. [47] La reciente generación de mutantes mediados por ZFN de rata, pez cebra , maíz y tabaco y las mejoras en los enfoques basados en TALEN dan testimonio de la importancia de los métodos, y la lista se está expandiendo rápidamente. La edición del genoma con nucleasas diseñadas probablemente contribuirá a muchos campos de las ciencias biológicas, desde el estudio de las funciones genéticas en plantas y animales hasta la terapia génica en humanos. Por ejemplo, el campo de la biología sintética , cuyo objetivo es diseñar células y organismos para que realicen funciones novedosas, probablemente se beneficiará de la capacidad de la nucleasa diseñada para agregar o eliminar elementos genómicos y, por lo tanto, crear sistemas complejos. [47] Además, las funciones genéticas se pueden estudiar utilizando células madre con nucleasas diseñadas.
A continuación se enumeran algunas tareas específicas que este método puede realizar:
La combinación de descubrimientos recientes en ingeniería genética, en particular la edición de genes, y las últimas mejoras en las tecnologías de reproducción bovina (por ejemplo, cultivo de embriones in vitro ) permite la edición del genoma directamente en ovocitos fertilizados utilizando endonucleasas sintéticas altamente específicas. Las endonucleasas guiadas por ARN: repeticiones palindrómicas cortas agrupadas y regularmente espaciadas asociadas a Cas9 (CRISPR/Cas9) son una nueva herramienta que aumenta aún más la gama de métodos disponibles . En particular, las endonucleasas diseñadas con CRISPR/Cas9 permiten el uso de múltiples ARN guía para Knockouts (KO) simultáneos en un solo paso mediante inyección directa citoplasmática (CDI) en cigotos de mamíferos. [48]
Además, la edición genética se puede aplicar a ciertos tipos de peces en acuicultura, como el salmón del Atlántico. La edición genética en peces es actualmente experimental, pero las posibilidades incluyen crecimiento, resistencia a enfermedades, esterilidad, reproducción controlada y color. La selección de estos rasgos puede permitir un entorno más sostenible y un mejor bienestar para los peces. [49]
El salmón AquAdvantage es un salmón del Atlántico genéticamente modificado desarrollado por AquaBounty Technologies. El gen regulador de la hormona del crecimiento en el salmón del Atlántico se reemplaza con el gen regulador de la hormona del crecimiento del salmón Chinook del Pacífico y una secuencia promotora de la faneca oceánica [50]
Gracias al desarrollo paralelo de la transcriptómica unicelular, la edición del genoma y nuevos modelos de células madre, ahora estamos entrando en un período científicamente apasionante en el que la genética funcional ya no se limita a modelos animales, sino que se puede realizar directamente en muestras humanas. El análisis de la expresión génica unicelular ha resuelto una hoja de ruta transcripcional del desarrollo humano a partir de la cual se están identificando genes candidatos clave para estudios funcionales. Utilizando datos de transcriptómica global para guiar la experimentación, la herramienta de edición del genoma basada en CRISPR ha hecho posible alterar o eliminar genes clave para dilucidar su función en un entorno humano. [51]

La edición del genoma utilizando meganucleasa , [52] ZFN y TALEN proporciona una nueva estrategia para la manipulación genética en plantas y es probable que ayude en la ingeniería de los rasgos deseados de las plantas mediante la modificación de genes endógenos. Por ejemplo, la adición de genes específicos de un sitio en las principales especies de cultivos se puede utilizar para la "acumulación de rasgos", mediante la cual varios rasgos deseados se vinculan físicamente para garantizar su cosegregación durante los procesos de mejoramiento. [33] Recientemente se han informado avances en tales casos en Arabidopsis thaliana [53] [54] [55] y Zea mays . En Arabidopsis thaliana , utilizando la orientación genética asistida por ZFN, se introdujeron dos genes resistentes a herbicidas (acetolactato sintasa de tabaco SuRA y SuRB) en loci SuR con hasta un 2% de células transformadas con mutaciones. [53] En Zea mays, la alteración del locus objetivo se logró mediante DSB inducidas por ZFN y el NHEJ resultante. En este caso , ZFN también se utilizó para impulsar el casete de expresión génica (PAT) de tolerancia a herbicidas en el locus endógeno IPK1 objetivo. [56] Se ha demostrado que dicha modificación del genoma observada en las plantas regeneradas es heredable y se transmitió a la siguiente generación. [56] Un ejemplo potencialmente exitoso de la aplicación de técnicas de edición del genoma en la mejora de cultivos se puede encontrar en el banano, donde los científicos utilizaron la edición CRISPR/Cas9 para inactivar el virus endógeno del rayado del banano en el genoma B del banano ( Musa spp. ) para superarlo. un desafío importante en el mejoramiento del banano. [57]
Además, la ingeniería genómica basada en TALEN ha sido ampliamente probada y optimizada para su uso en plantas. [58] Las fusiones TALEN también han sido utilizadas por una empresa estadounidense de ingredientes alimentarios, Calyxt, [59] para mejorar la calidad de los productos de aceite de soja [60] y aumentar el potencial de almacenamiento de las patatas [61].
Es necesario realizar varias optimizaciones para mejorar la edición de genomas de plantas utilizando la orientación mediada por ZFN. [62] Existe la necesidad de un diseño confiable y pruebas posteriores de las nucleasas, la ausencia de toxicidad de las nucleasas, la elección apropiada del tejido vegetal para el objetivo, las rutas de inducción de la actividad enzimática, la falta de mutagénesis fuera del objetivo. y una detección fiable de casos mutados. [62]
Un método de administración común de CRISPR/Cas9 en plantas es la transformación basada en Agrobacterium . [63] El ADN-T se introduce directamente en el genoma de la planta mediante un mecanismo T4SS. Los casetes de expresión basados en Cas9 y ARNg se convierten en plásmidos Ti , que se transforman en Agrobacterium para su aplicación en plantas. [63] Para mejorar la entrega de Cas9 en plantas vivas, se están utilizando virus para una entrega transgénica más efectiva. [63]
La práctica ideal de terapia génica es aquella que reemplaza el gen defectuoso con un alelo normal en su ubicación natural. Esto es ventajoso con respecto a un gen administrado por vía viral, ya que no es necesario incluir las secuencias codificantes completas y las secuencias reguladoras cuando solo es necesario alterar una pequeña proporción del gen, como suele ser el caso. [64] [65] La expresión de los genes parcialmente reemplazados también es más consistente con la biología celular normal que los genes completos que son transportados por vectores virales.
El primer uso clínico de la edición del genoma basada en TALEN fue en el tratamiento de la leucemia linfoblástica aguda CD19+ en un niño de 11 meses en 2015. Se diseñaron células T de donantes modificadas para atacar las células leucémicas, hacerlas resistentes al alemtuzumab y evadirlas. detección por el sistema inmunológico del huésped después de la introducción. [66] [67]
Se han realizado extensas investigaciones en células y animales utilizando CRISPR-Cas9 para intentar corregir las mutaciones genéticas que causan enfermedades genéticas como el síndrome de Down, la espina bífida, la anencefalia y los síndromes de Turner y Klinefelter. [68]
En febrero de 2019, los científicos médicos que trabajan con Sangamo Therapeutics , con sede en Richmond, California , anunciaron la primera terapia de edición de genes humanos "en el cuerpo" para alterar permanentemente el ADN , en un paciente con síndrome de Hunter . [69] Los ensayos clínicos realizados por Sangamo que involucran la edición de genes utilizando Zinc Finger Nuclease (ZFN) están en curso. [70]
Los investigadores han utilizado impulsores genéticos CRISPR-Cas9 para modificar genes asociados con la esterilidad en A. gambiae , el vector de la malaria. [71] Esta técnica tiene implicaciones adicionales en la erradicación de otras enfermedades transmitidas por vectores como la fiebre amarilla, el dengue y el Zika. [72]
El sistema CRISPR-Cas9 se puede programar para modular la población de cualquier especie bacteriana dirigiéndose a genotipos clínicos o aislados epidemiológicos. Puede habilitar selectivamente las especies bacterianas beneficiosas sobre las dañinas al eliminar patógenos, lo que le da una ventaja sobre los antibióticos de amplio espectro. [46]
Se están investigando aplicaciones antivirales para terapias dirigidas a virus humanos como el VIH, el herpes y el virus de la hepatitis B. CRISPR se puede utilizar para atacar al virus o al huésped y alterar los genes que codifican las proteínas receptoras de la superficie celular del virus. [44] En noviembre de 2018, He Jiankui anunció que había editado dos embriones humanos para intentar desactivar el gen CCR5 , que codifica un receptor que el VIH utiliza para ingresar a las células. Dijo que las gemelas, Lulu y Nana , habían nacido unas semanas antes. Dijo que las niñas todavía portaban copias funcionales de CCR5 junto con CCR5 discapacitado ( mosaicismo ) y aún eran vulnerables al VIH. El trabajo fue ampliamente condenado por ser poco ético, peligroso y prematuro. [73]
En enero de 2019, científicos en China informaron de la creación de cinco monos clonados idénticos editados genéticamente, utilizando la misma técnica de clonación que se utilizó con Zhong Zhong y Hua Hua , los primeros monos clonados, y la oveja Dolly , y el mismo gen. Edición de la técnica Crispr - Cas9 supuestamente utilizada por He Jiankui para crear los primeros bebés humanos modificados genéticamente, Lulu y Nana . Los clones de monos se hicieron para estudiar varias enfermedades médicas. [74] [75]
En el futuro, un objetivo importante de la investigación sobre la edición del genoma con nucleasas modificadas debe ser la mejora de la seguridad y la especificidad de la acción de las nucleasas. [76] Por ejemplo, mejorar la capacidad de detectar eventos fuera del objetivo puede mejorar nuestra capacidad de aprender sobre formas de prevenirlos. Además, los dedos de zinc utilizados en las ZFN rara vez son completamente específicos y algunos pueden provocar una reacción tóxica. Sin embargo, se ha informado que la toxicidad se reduce mediante modificaciones realizadas en el dominio de escisión de ZFN. [sesenta y cinco]
Además, la investigación de Dana Carroll sobre la modificación del genoma con nucleasas diseñadas ha demostrado la necesidad de comprender mejor la maquinaria básica de recombinación y reparación del ADN. En el futuro, un posible método para identificar objetivos secundarios sería capturar los extremos rotos de las células que expresan las ZFN y secuenciar el ADN flanqueante mediante secuenciación de alto rendimiento. [sesenta y cinco]
Debido a la facilidad de uso y rentabilidad de CRISPR, actualmente se están realizando extensas investigaciones al respecto. Actualmente hay más publicaciones sobre CRISPR que ZFN y TALEN a pesar de lo reciente que es el descubrimiento de CRISPR. [44] Tanto CRISPR como TALEN son las opciones preferidas para implementarse en producciones a gran escala debido a su precisión y eficiencia.
La edición del genoma también se produce como un proceso natural sin ingeniería genética artificial. Los agentes competentes para editar códigos genéticos son virus o agentes de ARN subvirales.
Aunque GEEN tiene mayor eficiencia que muchos otros métodos de genética inversa, todavía no es muy eficiente; en muchos casos menos de la mitad de las poblaciones tratadas obtienen los cambios deseados. [53] Por ejemplo, cuando uno planea usar el NHEJ de la célula para crear una mutación, los sistemas HDR de la célula también estarán trabajando corrigiendo el DSB con tasas de mutación más bajas.
Tradicionalmente, los ratones han sido la elección más común para los investigadores como anfitriones de un modelo de enfermedad. CRISPR puede ayudar a cerrar la brecha entre este modelo y los ensayos clínicos en humanos mediante la creación de modelos de enfermedades transgénicas en animales más grandes, como cerdos, perros y primates no humanos. [77] [78] Usando el sistema CRISPR-Cas9, la proteína Cas9 programada y el sgRNA se pueden introducir directamente en cigotos fertilizados para lograr las modificaciones genéticas deseadas al crear modelos transgénicos en roedores. Esto permite evitar la etapa habitual de selección de células en la generación de líneas transgénicas y, como resultado, reduce el tiempo de generación en un 90%. [78]
Un potencial que aporta CRISPR con su eficacia es la aplicación de xenotrasplantes. En ensayos de investigación anteriores, CRISPR demostró la capacidad de atacar y eliminar retrovirus endógenos, lo que reduce el riesgo de transmitir enfermedades y reduce las barreras inmunitarias. [44] La eliminación de estos problemas mejora la función del órgano donado, lo que acerca esta aplicación a la realidad.
En las plantas, la edición del genoma se considera una solución viable para la conservación de la biodiversidad. Los impulsores genéticos son una herramienta potencial para alterar la tasa de reproducción de especies invasoras , aunque existen importantes riesgos asociados. [79]
Muchos transhumanistas ven la edición del genoma como una herramienta potencial para la mejora humana . [80] [81] [82] El biólogo australiano y profesor de genética David Andrew Sinclair señala que "las nuevas tecnologías de edición del genoma permitirán su uso en individuos (...) para tener (...) niños más sanos" - bebes diseñadores . [83] Según un informe de septiembre de 2016 del Consejo de Bioética de Nuffield, en el futuro será posible mejorar a las personas con genes de otros organismos o genes totalmente sintéticos para, por ejemplo, mejorar la visión nocturna y el sentido del olfato . [84] [85] George Church ha compilado una lista de posibles modificaciones genéticas para rasgos posiblemente ventajosos, como una menor necesidad de dormir , cambios relacionados con la cognición que protegen contra la enfermedad de Alzheimer, resistencias a enfermedades y capacidades de aprendizaje mejoradas , junto con algunos de los estudios asociados. y posibles efectos negativos. [86] [87]
La Academia Nacional de Ciencias y la Academia Nacional de Medicina de Estados Unidos publicaron un informe en febrero de 2017 dando apoyo calificado a la edición del genoma humano. [88] Recomendaron que algún día se podrían permitir ensayos clínicos para la edición del genoma una vez que se hayan encontrado respuestas a los problemas de seguridad y eficiencia "pero sólo para condiciones graves bajo una supervisión estricta". [89]
En la declaración de Evaluación de Amenazas Mundiales de 2016 de la Comunidad de Inteligencia de EE. UU., el Director de Inteligencia Nacional de Estados Unidos, James R. Clapper , nombró la edición del genoma como un arma potencial de destrucción masiva , afirmando que la edición del genoma realizada por países con estándares regulatorios o éticos "diferentes de "Los países occidentales" probablemente aumentan el riesgo de creación de agentes o productos biológicos nocivos. Según la declaración, la amplia distribución, el bajo costo y el ritmo acelerado de desarrollo de esta tecnología, su uso indebido deliberado o no intencional podría tener implicaciones económicas y de seguridad nacional de gran alcance. [90] [91] [92] Por ejemplo, tecnologías como CRISPR podrían usarse para crear "mosquitos asesinos" que causen plagas que acaben con los cultivos básicos. [92]
Según un informe de septiembre de 2016 del Consejo de Bioética de Nuffield , la simplicidad y el bajo costo de las herramientas para editar el código genético permitirán a los aficionados -o " biohackers "- realizar sus propios experimentos, lo que plantea un riesgo potencial por la liberación de genes modificados genéticamente. insectos. La revisión también encontró que los riesgos y beneficios de modificar el genoma de una persona (y que esos cambios se transmitan a las generaciones futuras) son tan complejos que exigen un escrutinio ético urgente. Tales modificaciones podrían tener consecuencias no deseadas que podrían dañar no sólo al niño, sino también a sus futuros hijos, ya que el gen alterado estaría en su esperma o en sus óvulos. [84] [85] En 2001, los investigadores australianos Ronald Jackson e Ian Ramshaw fueron criticados por publicar un artículo en el Journal of Virology que exploraba el control potencial de ratones, una plaga importante en Australia, infectándolos con un virus de la viruela del ratón alterado que causar infertilidad, ya que la información confidencial proporcionada podría conducir a la fabricación de armas biológicas por parte de bioterroristas potenciales que podrían usar el conocimiento para crear cepas resistentes a las vacunas de otros virus de la viruela, como la viruela , que podrían afectar a los humanos. [85] [93] Además, existen preocupaciones adicionales sobre los riesgos ecológicos de la liberación de impulsores genéticos en poblaciones silvestres. [85] [94] [95]
En 2007, el Premio Nobel de Fisiología o Medicina fue otorgado a Mario Capecchi, Martin Evans y Oliver Smithies "por sus descubrimientos de principios para introducir modificaciones genéticas específicas en ratones mediante el uso de células madre embrionarias". [19]
En 2020, el Premio Nobel de Química fue concedido a Emmanuelle Charpentier y Jennifer Doudna por "el desarrollo de un método de edición del genoma". [96]
{{cite book}}: |journal=ignorado ( ayuda )"La OMS lanza un registro mundial sobre la edición del genoma humano". PharmaBiz, 31 de agosto de 2019. Gale General OneFile, consultado el 27 de abril de 2020.