
Una unión Holliday es una estructura de ácido nucleico ramificada que contiene cuatro brazos bicatenarios unidos. Estos brazos pueden adoptar una de varias conformaciones dependiendo de las concentraciones de sal tampón y la secuencia de nucleobases más cercanas a la unión. La estructura lleva el nombre de Robin Holliday , el biólogo molecular que propuso su existencia en 1964.
En biología, las uniones de Holliday son un intermediario clave en muchos tipos de recombinación genética , así como en la reparación de roturas de doble hebra . Estas uniones suelen tener una secuencia simétrica y, por lo tanto, son móviles, lo que significa que los cuatro brazos individuales pueden deslizarse a través de la unión en un patrón específico que preserva en gran medida el emparejamiento de bases . Además, en algunas moléculas de ARN funcionales aparecen uniones de cuatro brazos similares a las uniones de Holliday .
Las uniones Holliday inmóviles, con secuencias asimétricas que bloquean las hebras en una posición específica, fueron creadas artificialmente por científicos para estudiar su estructura como modelo para las uniones Holliday naturales. Estas uniones también encontraron uso posteriormente como componentes estructurales básicos en la nanotecnología del ADN , donde múltiples uniones Holliday pueden combinarse en geometrías diseñadas específicas que proporcionan a las moléculas un alto grado de rigidez estructural .
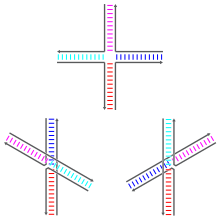
Pueden existir uniones Holliday en una variedad de isómeros conformacionales con diferentes patrones de apilamiento coaxial entre los cuatro brazos de doble hélice. El apilamiento coaxial es la tendencia de los extremos romos de los ácidos nucleicos a unirse entre sí mediante interacciones entre las bases expuestas. Hay tres conformadores posibles: una forma no apilada (o de X abierta) y dos formas apiladas. La forma no apilada domina en ausencia de cationes divalentes como Mg 2+ , debido a la repulsión electrostática entre las cadenas principales cargadas negativamente de las hebras. En presencia de al menos aproximadamente 0,1 m M Mg 2+ , la repulsión electrostática se contrarresta y predominan las estructuras apiladas. En 2000, no se sabía con certeza si el blindaje electrostático era el resultado de la unión de cationes a la unión en un sitio específico o de la presencia de una colección difusa de los iones en solución. [1]
La forma no apilada es una conformación extendida, plana, casi cuadrada. Por otro lado, los confórmeros apilados tienen dos dominios continuos de doble hélice separados por un ángulo de aproximadamente 60° en dirección derecha . Dos de las cuatro hebras permanecen aproximadamente helicoidales y permanecen dentro de cada uno de los dos dominios de doble hélice, mientras que las otras dos se cruzan entre los dos dominios de forma antiparalela . [1]
Las dos posibles formas apiladas se diferencian en qué pares de brazos están apilados entre sí; cuál de los dos domina depende en gran medida de las secuencias de bases más cercanas a la unión. Algunas secuencias dan como resultado un equilibrio entre los dos confórmeros, mientras que otras prefieren un solo confórmero. En particular, las uniones que contienen la secuencia A-CC que une el punto de unión parecen preferir fuertemente el confórmero que permite que se forme un enlace de hidrógeno entre la segunda citosina y uno de los fosfatos en el punto de unión. Si bien la mayoría de los estudios se han centrado en las identidades de las cuatro bases más cercanas a la unión de cada brazo, es evidente que las bases más alejadas también pueden afectar las conformaciones de apilamiento observadas. [1]
En uniones con secuencias simétricas, el punto de bifurcación es móvil y puede migrar en un proceso de caminata aleatoria . La tasa de migración de ramas varía drásticamente con la concentración de iones, con tiempos de un solo paso que aumentan de 0,3 a 0,4 ms sin iones a 270-300 ms con Mg 2+ 10 mM . El cambio en la tasa se correlaciona con la formación de estructuras apiladas versus no apiladas. [1]
Las uniones Holliday con una muesca , o rotura en uno de los hilos, en el punto de unión adoptan una orientación perpendicular y siempre prefieren el conformador de apilamiento que coloca la muesca en un hilo cruzado en lugar de un hilo helicoidal. [1]
Las uniones de ARN Holliday asumen una conformación apilada antiparalela en altas concentraciones de magnesio, una conformación apilada perpendicular en concentraciones moderadas y giran hacia una conformación apilada paralela en concentraciones bajas, mientras que incluso pequeñas concentraciones de iones de calcio favorecen el confórmero antiparalelo. [1]

La unión Holliday es un intermediario clave en la recombinación homóloga , un proceso biológico que aumenta la diversidad genética al cambiar genes entre dos cromosomas , así como eventos de recombinación específicos de sitio que involucran integrasas . También intervienen en la reparación de roturas de doble cadena . [1] Además, pueden surgir estructuras cruciformes que involucran uniones Holliday para aliviar la tensión helicoidal en secuencias simétricas en superenrollamientos de ADN . [2] Si bien las uniones de cuatro brazos también aparecen en moléculas de ARN funcionales , como el ARN espliceosómico U1 y la ribozima en horquilla del virus de la mancha anular del tabaco , éstas generalmente contienen nucleótidos desapareados entre los dominios de doble hélice emparejados y, por lo tanto, no adoptan estrictamente la estructura de vacaciones. [1]
Las uniones Holliday en recombinación homóloga se encuentran entre secuencias idénticas o casi idénticas, lo que lleva a una disposición simétrica de secuencias alrededor de la unión central. Esto permite que ocurra un proceso de migración de ramas donde las hebras se mueven a través del punto de unión. [1] La escisión o resolución de la unión de Holliday puede ocurrir de dos maneras. La escisión del conjunto original de hebras conduce a dos moléculas que pueden mostrar conversión genética pero no cruce cromosómico , mientras que la escisión del otro conjunto de dos hebras hace que las moléculas recombinantes resultantes muestren cruce. Todos los productos, independientemente de la escisión, son heterodúplex en la región de migración de la unión Holliday. [3]
Muchas proteínas pueden reconocer o distorsionar la estructura de unión de Holliday. Una de esas clases contiene enzimas que resuelven uniones y que escinden las uniones, a veces de una manera específica de secuencia. Estas proteínas distorsionan la estructura de la unión de diversas maneras, a menudo tirando de la unión hacia una conformación desapilada, rompiendo los pares de bases centrales y/o cambiando los ángulos entre los cuatro brazos. Otras clases son las proteínas de migración de ramas que aumentan el tipo de cambio en órdenes de magnitud y las recombinasas específicas de sitio . [1] En procariotas, las resolvasas de unión de Holliday se dividen en dos familias, integrasas y nucleasas, que son estructuralmente similares aunque sus secuencias no están conservadas. [3]
En eucariotas, dos modelos principales de cómo la recombinación homóloga repara las roturas de doble hebra en el ADN son la vía de reparación de roturas de doble hebra (DSBR) (a veces llamada modelo de doble unión Holliday ) y la vía de recocido de hebras dependiente de la síntesis (SDSA). [4] En el caso de rotura de doble cadena, el extremo 3' se degrada y el extremo 5' más largo invade la cromátida hermana contigua, formando una burbuja de replicación. A medida que esta burbuja se acerca al ADN roto, la cadena antisentido 5' más larga invade nuevamente la cadena sentido de esta porción de ADN, transcribiendo una segunda copia. Cuando finaliza la replicación, ambas colas se vuelven a conectar para formar dos uniones Holliday, que luego son escindidas en una variedad de patrones por proteínas. [5] Puede verse una animación de este proceso aquí. [6]
Las roturas del ADN de doble hebra en las bacterias se reparan mediante la vía RecBCD de recombinación homóloga. Se cree que las roturas que se producen en una sola de las dos cadenas de ADN, conocidas como espacios de una sola cadena, se reparan mediante la vía RecF . Tanto la vía RecBCD como la RecF incluyen una serie de reacciones conocidas como migración de ramas, en la que se intercambian hebras individuales de ADN entre dos moléculas de ADN dúplex entrecruzadas, y resolución , en la que esas dos moléculas de ADN entrecruzadas se cortan y se restauran a su estado normal. estado de doble hebra. [7] La recombinación homóloga ocurre en varios grupos de virus. En los virus de ADN como el herpesvirus , la recombinación se produce mediante un mecanismo de ruptura y reinserción como en las bacterias y los eucariotas. [8] En las bacterias, la migración de ramas es facilitada por el complejo RuvABC o proteína RecG, motores moleculares que utilizan la energía de la hidrólisis del ATP para mover la unión. Luego, la unión debe resolverse en dos dúplex separados, restaurando la configuración parental o una configuración cruzada. La resolución puede ocurrir de forma horizontal o vertical durante la recombinación homóloga, dando productos de parche (si están en la misma orientación durante la reparación de rotura de doble hebra) o productos de empalme (si están en diferentes orientaciones durante la reparación de rotura de doble hebra). [9] [10] RuvA y RuvB son proteínas de migración de ramas, mientras que RuvC es una enzima que resuelve uniones. [1]
Existe evidencia de recombinación en algunos virus de ARN , específicamente en virus de ARN monocatenario de sentido positivo como los retrovirus , los picornavirus y los coronavirus . Existe controversia sobre si se produce recombinación homóloga en virus ssRNA de sentido negativo como la gripe . [11]
En la levadura en ciernes Saccharomyces cerevisiae , las uniones de Holliday se pueden resolver mediante cuatro vías diferentes que representan esencialmente toda la resolución de las uniones de Holliday in vivo . [12] La vía que produce la mayoría de los cruces en la levadura en ciernes de S. cerevisiae , y posiblemente en los mamíferos, involucra las proteínas EXO1 , el heterodímero MLH1 - MLH3 (llamado MutL gamma) y SGS1 (ortólogo de la helicasa del síndrome de Bloom ). [12] El heterodímero MLH1-MLH3 se une preferentemente a las uniones Holliday. [13] Es una endonucleasa que produce roturas monocatenarias en ADN bicatenario superenrollado. [13] [14] El heterodímero MLH1-MLH3 promueve la formación de recombinantes cruzados . [15] Mientras que las otras tres vías, que involucran las proteínas MUS81 -MMS4, SLX1 y YEN1, respectivamente, pueden promover la resolución de la unión Holliday in vivo, la ausencia de las tres nucleasas tiene solo un impacto modesto en la formación de productos cruzados.
Los mutantes dobles eliminados tanto para MLH3 (vía principal) como para MMS4 (vía menor) mostraron un cruce dramáticamente reducido en comparación con el tipo salvaje (de 6 a 17 veces); sin embargo, la viabilidad de las esporas fue razonablemente alta (62%) y la disyunción cromosómica parecía mayoritariamente funcional. [15]
Aunque MUS81 es un componente de una vía cruzada menor en la meiosis de levaduras, plantas y vertebrados en ciernes, [16] en el protozoo Tetrahymena thermophila , MUS81 parece ser parte de una vía cruzada esencial, si no predominante. [16] La vía MUS81 también parece ser la vía de cruce predominante en la levadura de fisión Schizosaccharomyces pombe . [dieciséis]
Las proteínas MSH4 y MSH5 forman una estructura heterooligomérica (heterodímero) en levaduras y humanos. [17] [18] [19] En la levadura Saccharomyces cerevisiae, MSH4 y MSH5 actúan específicamente para facilitar los cruces entre cromosomas homólogos durante la meiosis. [17] El complejo MSH4/MSH5 se une y estabiliza las uniones dobles de Holliday y promueve su resolución en productos cruzados. Un mutante hipomórfico (parcialmente funcional) MSH4 de S. cerevisiae mostró una reducción del 30% en todo el genoma en el número de cruces y una gran cantidad de meiosis con cromosomas sin intercambio. [20] Sin embargo, este mutante dio lugar a patrones de viabilidad de las esporas que sugieren que la segregación de cromosomas sin intercambio se produjo de manera eficiente. Así, en S. cerevisiae la segregación adecuada aparentemente no depende enteramente de cruces entre pares homólogos.

La nanotecnología del ADN es el diseño y fabricación de estructuras artificiales de ácidos nucleicos como materiales de ingeniería para la nanotecnología en lugar de portadores de información genética en células vivas. El campo utiliza estructuras de ADN ramificadas como componentes fundamentales para crear estructuras más complejas y diseñadas racionalmente. Por tanto, las uniones navideñas son componentes de muchas de estas estructuras de ADN. Como los complejos de uniones Holliday aislados son demasiado flexibles para ensamblarse en grandes conjuntos ordenados, se utilizan motivos estructurales con múltiples uniones Holliday para crear " mosaicos " rígidos que luego pueden ensamblarse en "conjuntos" más grandes. [22] [23]

El motivo más común es el complejo de doble cruce (DX), que contiene dos uniones Holliday muy próximas entre sí, lo que da como resultado una estructura rígida que puede autoensamblarse en matrices más grandes. La estructura de la molécula DX obliga a las uniones Holliday a adoptar una conformación con los dominios de doble hélice directamente uno al lado del otro, en contraste con su ángulo preferido de aproximadamente 60°. El complejo se puede diseñar para forzar las uniones a una orientación paralela o antiparalela, pero en la práctica la variedad antiparalela se comporta mejor y la versión paralela rara vez se utiliza. [22] [23]
El motivo estructural DX es el componente fundamental del método del origami de ADN , que se utiliza para crear estructuras bidimensionales y tridimensionales más grandes de forma arbitraria. En lugar de utilizar mosaicos DX individuales, una única hebra larga de andamio se dobla en la forma deseada mediante una serie de hebras cortas de grapas. Cuando se ensambla, la hebra de andamio es continua a través de los dominios de doble hélice, mientras que las hebras de grapa participan en las uniones Holliday como hebras cruzadas. [25]
Se han demostrado algunos tipos de losas que conservan el ángulo nativo de 60° de la unión Holliday. Una de esas matrices utiliza mosaicos que contienen cuatro uniones Holliday en una disposición de paralelogramo. Esta estructura tenía la ventaja de permitir visualizar directamente el ángulo de unión mediante microscopía de fuerza atómica . Se han utilizado mosaicos de tres uniones Holliday de forma triangular para crear matrices tridimensionales periódicas para su uso en cristalografía de biomoléculas por rayos X. Estas estructuras reciben su nombre por su similitud con las unidades estructurales basadas en el principio de tensegridad , que utiliza miembros tanto en tensión como en compresión. [22] [23]
Robin Holliday propuso la estructura de unión que ahora lleva su nombre como parte de su modelo de recombinación homóloga en 1964, basándose en sus investigaciones sobre los organismos Ustilago maydis y Saccharomyces cerevisiae . El modelo proporcionó un mecanismo molecular que explica tanto la conversión genética como el cruce cromosómico . Holliday se dio cuenta de que la vía propuesta crearía segmentos de ADN heterodúplex con desajustes de bases entre diferentes versiones de un solo gen. Predijo que la célula tendría un mecanismo para reparar los desajustes, que se descubrió más tarde. [3] Antes del modelo de Holliday, el modelo aceptado implicaba un mecanismo de elección de copia [26] donde la nueva cadena se sintetiza directamente a partir de partes de las diferentes cadenas originales. [27]
En el modelo original de Holliday para la recombinación homóloga, las roturas de una sola hebra ocurren en el mismo punto en una hebra de cada ADN parental. Los extremos libres de cada hebra rota migran hacia la otra hélice de ADN. Allí, las hebras invasoras se unen a los extremos libres que encuentran, lo que da como resultado la unión Holliday. A medida que cada hebra cruzada se vuelve a hibridar con su hebra asociada original, desplaza la hebra complementaria original que tenía delante. Esto hace que la unión Holliday migre, creando los segmentos heterodúplex. Dependiendo de qué cadena se utilizó como plantilla para reparar la otra, las cuatro células resultantes de la meiosis podrían terminar con tres copias de un alelo y sólo una del otro, en lugar de las dos habituales de cada una, una propiedad conocida como conversión genética. . [3]
El modelo original de Holliday suponía que el ADN heterodúplex estaría presente en ambos cromosomas, pero los datos experimentales con levaduras lo refutaron. Un modelo actualizado de Matt Meselson y Charley Radding en 1975 introdujo la idea de migración de sucursales. [26] Otras observaciones en la década de 1980 llevaron a la propuesta de mecanismos alternativos para la recombinación, como el modelo de rotura de doble cadena (por Jack Szostak , Frank Stahl y otros) y el modelo de recocido de una sola cadena. Un tercero, el modelo de recocido de cadenas dependiente de la síntesis, no implicaba uniones Holliday. [3]
La primera evidencia experimental de la estructura de la unión de Holliday provino de estudios de microscopía electrónica a fines de la década de 1970, donde la estructura de cuatro brazos era claramente visible en imágenes de ADN de plásmidos y bacteriófagos . Más tarde, en la década de 1980, se identificaron las enzimas responsables de iniciar la formación y la unión a las uniones Holliday, aunque a partir de 2004 la identificación de las resolvasas de las uniones Holliday en mamíferos siguió siendo difícil de alcanzar (sin embargo, consulte la sección "Resolución de las uniones Holliday", más arriba para obtener más información). información reciente). En 1983, Nadrian Seeman construyó por primera vez moléculas artificiales de unión Holliday a partir de oligonucleótidos sintéticos , lo que permitió un estudio más directo de sus propiedades físicas. Gran parte de los primeros análisis de la estructura de la unión de Holliday se infirieron a partir de electroforesis en gel , FRET y estudios de huella de radicales hidroxilo y nucleasas . En la década de 1990, estuvieron disponibles los métodos de cristalografía y RMN de ácidos nucleicos , así como herramientas de modelado molecular computacional . [1] [3] [28]
Inicialmente, los genetistas asumieron que la unión adoptaría una conformación paralela en lugar de antiparalela , porque eso colocaría a los dúplex homólogos en una alineación más estrecha entre sí. [1] Los análisis químicos realizados en la década de 1980 mostraron que la unión en realidad prefería la conformación antiparalela, un hallazgo que se consideró controvertido, y el propio Robin Holliday inicialmente dudó de los hallazgos. [1] [3] La estructura antiparalela más tarde fue ampliamente aceptada debido a los datos de cristalografía de rayos X sobre moléculas in vitro , aunque a partir de 2004 las implicaciones para la estructura in vivo seguían sin estar claras, especialmente la estructura de las uniones a menudo es alterada por proteínas. obligado a ello. [3]
La base conceptual de la nanotecnología del ADN fue establecida por primera vez por Nadrian Seeman a principios de los años 1980. [29] En ese momento se conocían varias estructuras naturales de ADN ramificado, incluida la bifurcación de replicación del ADN y la unión móvil de Holliday, pero la idea de Seeman fue que se podían crear uniones de ácido nucleico inmóviles diseñando adecuadamente las secuencias de las cadenas para eliminar la simetría en la cadena. molécula ensamblada, y que estas uniones inmóviles podrían, en principio, combinarse en redes cristalinas rígidas. El primer artículo teórico que proponía este esquema se publicó en 1982, y al año siguiente se publicó la primera demostración experimental de una unión de ADN inmóvil. [23] [30] Seeman desarrolló el motivo de doble cruce (DX) más rígido , adecuado para formar redes bidimensionales, demostrado en 1998 por él y Erik Winfree . [22] En 2006, Paul Rothemund demostró por primera vez la técnica del origami de ADN para crear de forma fácil y robusta estructuras de ADN plegadas de forma arbitraria. Este método permitió la creación de estructuras mucho más grandes de lo que antes eran posibles y cuyo diseño y síntesis son menos exigentes técnicamente. [31] Seeman finalmente publicó la síntesis de una red tridimensional en 2009, casi treinta años después de que se propusiera lograrla. [32]