BRAF es un gen humanoque codifica una proteína llamada B-Raf. El gen también se conoce como protooncogén B-Raf y homólogo del oncogén viral del sarcoma murino v-Raf B , mientras que la proteína se conoce más formalmente como serina/treonina-proteína quinasa B-Raf . [5] [6]
La proteína B-Raf participa en el envío de señales dentro de las células que están implicadas en la dirección del crecimiento celular . En 2002, se demostró que presentaba mutaciones en algunos cánceres humanos . [7]
Ciertas otras mutaciones hereditarias del gen BRAF causan defectos de nacimiento.
Se han desarrollado medicamentos para tratar los cánceres provocados por mutaciones BRAF . Dos de estos medicamentos, vemurafenib [8] y dabrafenib , están aprobados por la FDA para el tratamiento del melanoma en etapa avanzada. Vemurafenib fue el primer medicamento aprobado que surgió del descubrimiento de fármacos basado en fragmentos . [9]

B-Raf es un miembro de la familia de las quinasas Raf de las proteínas quinasas de transducción de señales de crecimiento . Esta proteína desempeña un papel en la regulación de la vía de señalización de las quinasas MAP / ERK , que afecta la división , diferenciación y secreción celular. [10]
B-Raf es una proteína quinasa específica de serina/treonina , de 766 aminoácidos , con transducción de señales regulada . En términos generales, se compone de tres dominios conservados característicos de la familia de quinasas Raf : región conservada 1 (CR1), un dominio autorregulador de unión a Ras - GTP [11] , región conservada 2 (CR2), una región bisagra rica en serina , y región conservada 3 (CR3), un dominio de proteína quinasa catalítica que fosforila una secuencia de consenso en sustratos proteicos. [12] En su conformación activa, B-Raf forma dímeros a través de enlaces de hidrógeno e interacciones electrostáticas de sus dominios quinasos. [13]
La región conservada 1 (CR1) autoinhibe el dominio quinasa de B-Raf (CR3) de modo que la señalización de B-Raf está regulada en lugar de ser constitutiva. [12] Los residuos 155-227 [14] forman el dominio de unión a Ras (RBD), que se une al dominio efector de Ras-GTP para liberar CR1 y detener la inhibición de la quinasa. Los residuos 234-280 comprenden un motivo de dedo de zinc de unión a DAG /éster de forbol que participa en el acoplamiento de la membrana de B-Raf después de la unión a Ras. [14] [15]
La región conservada 2 (CR2) proporciona un conector flexible que conecta CR1 y CR3 y actúa como una bisagra. [16]
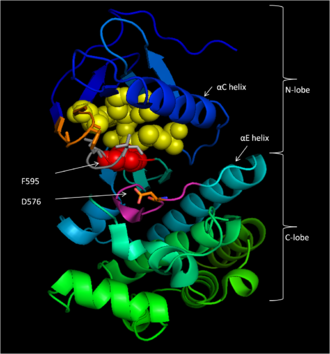
La región conservada 3 (CR3), residuos 457-717, [14] constituye el dominio de la quinasa enzimática de B-Raf. Esta estructura, en gran parte conservada [17] , es bilobal y está conectada por una región bisagra corta. [18] El lóbulo N más pequeño (residuos 457-530) es el principal responsable de la unión del ATP , mientras que el lóbulo C más grande (residuos 535-717) se une a las proteínas del sustrato . [17] El sitio activo es la hendidura entre los dos lóbulos, y el residuo catalítico Asp 576 se encuentra en el lóbulo C, mirando hacia el interior de esta hendidura. [14] [17]
El bucle P de B-Raf (residuos 464-471) estabiliza los grupos fosfato no transferibles del ATP durante la unión del ATP a la enzima. Específicamente, las amidas de la cadena principal S 467, F 468 y G 469 se unen por enlaces de hidrógeno al β-fosfato del ATP para anclar la molécula. Los motivos funcionales de B-Raf se han determinado analizando la homología de la PKA analizada por Hanks y Hunter con el dominio de la quinasa de B-Raf. [17]
V 471, C 532, W 531, T 529, L 514 y A 481 forman un bolsillo hidrofóbico dentro del cual la adenina del ATP se ancla a través de atracciones de Van der Waals tras la unión del ATP. [17] [19]
Los residuos 574-581 componen una sección del dominio quinasa responsable de apoyar la transferencia del fosfato γ de ATP al sustrato proteico de B-Raf. En particular, D 576 actúa como un aceptor de protones para activar el oxígeno hidroxilo nucleofílico en los residuos de serina o treonina del sustrato, lo que permite que se produzca la reacción de transferencia de fosfato mediada por catálisis básica . [17]
D 594, F 595 y G 596 componen un motivo central para la función de B-Raf tanto en su estado inactivo como en su estado activo. En el estado inactivo, F595 ocupa el bolsillo de unión de nucleótidos, lo que impide la entrada de ATP y disminuye la probabilidad de catálisis enzimática. [13] [19] [20] En el estado activo, D594 forma quelato del catión magnesio divalente que estabiliza los grupos β- y γ-fosfato del ATP, orientando el γ-fosfato para la transferencia. [17]
Los residuos 596-600 forman fuertes interacciones hidrofóbicas con el bucle P en la conformación inactiva de la quinasa, bloqueando la quinasa en su estado inactivo hasta que el bucle de activación se fosforila, desestabilizando estas interacciones con la presencia de carga negativa. Esto desencadena el cambio al estado activo de la quinasa. Específicamente, L597 y V600 del bucle de activación interactúan con G466, F468 y V471 del bucle P para mantener el dominio de la quinasa inactivo hasta que se fosforila. [18]
B-Raf es una proteína quinasa específica de serina/treonina . Como tal, cataliza la fosforilación de residuos de serina y treonina en una secuencia de consenso en proteínas objetivo por ATP , produciendo ADP y una proteína fosforilada como productos. [17] Dado que es una quinasa de transducción de señales altamente regulada , B-Raf primero debe unirse a Ras - GTP antes de volverse activa como enzima. [15] Una vez que B-Raf se activa, un núcleo catalítico de proteína quinasa conservado fosforila sustratos proteicos al promover el ataque nucleofílico del átomo de oxígeno hidroxilo de serina o treonina del sustrato activado en el grupo γ-fosfato de ATP a través de sustitución nucleofílica bimolecular . [17] [21] [22] [23]
El dominio quinasa (CR3) de las quinasas Raf humanas es inhibido por dos mecanismos: autoinhibición por su propio dominio regulador CR1 de unión a Ras - GTP y una falta de fosforilación postraduccional de residuos clave de serina y tirosina (S338 e Y341 para c-Raf) en la región bisagra CR2. Durante la activación de B-Raf, el dominio autoinhibitorio CR1 de la proteína primero une el dominio efector de Ras-GTP al dominio de unión a Ras (RBD) de CR1 para liberar el dominio quinasa CR3 como otros miembros de la familia de quinasas Raf humanas . La interacción CR1-Ras se fortalece posteriormente a través de la unión del subdominio rico en cisteína (CRD) de CR1 a Ras y fosfolípidos de membrana . [12] A diferencia de A-Raf y C-Raf , que deben fosforilarse en residuos CR2 que contienen hidroxilo antes de liberar completamente CR1 para volverse activo, B-Raf se fosforila constitutivamente en CR2 S445. [24] Esto permite que la fosfoserina cargada negativamente repela inmediatamente a CR1 a través de interacciones estéricas y electrostáticas una vez que el dominio regulador se libera, liberando el dominio de la quinasa CR3 para interactuar con las proteínas del sustrato.
Después de que se libera el dominio regulador autoinhibitorio CR1, el dominio quinasa CR3 de B-Raf debe cambiar a su confórmero activo de unión a ATP antes de que pueda catalizar la fosforilación de proteínas . En la conformación inactiva, F595 del motivo DFG bloquea el bolsillo de unión de adenina hidrofóbico mientras que los residuos del bucle de activación forman interacciones hidrofóbicas con el bucle P, impidiendo que el ATP acceda a su sitio de unión. Cuando el bucle de activación se fosforila, la carga negativa del fosfato es inestable en el entorno hidrofóbico del bucle P. Como resultado, el bucle de activación cambia de conformación , extendiéndose a través del lóbulo C del dominio quinasa . En este proceso, forma interacciones estabilizadoras de lámina β con la cadena β6. Mientras tanto, el residuo fosforilado se acerca a K507, formando un puente salino estabilizador para bloquear el bucle de activación en su lugar. El motivo DFG cambia de conformación con el bucle de activación, lo que hace que F595 se mueva fuera del sitio de unión del nucleótido de adenina y hacia un bolsillo hidrofóbico bordeado por las hélices αC y αE . Juntos, el DFG y el movimiento del bucle de activación tras la fosforilación abren el sitio de unión de ATP . Dado que todos los demás dominios catalíticos y de unión al sustrato ya están en su lugar, la fosforilación del bucle de activación por sí sola activa el dominio quinasa de B-Raf a través de una reacción en cadena que esencialmente elimina una tapa de un sitio activo preparado de otro modo. [18]

Para catalizar eficazmente la fosforilación de proteínas a través de la sustitución bimolecular de residuos de serina y treonina con ADP como grupo saliente , B-Raf primero debe unirse al ATP y luego estabilizar el estado de transición a medida que se transfiere el fosfato γ del ATP. [17]
B-Raf se une al ATP anclando el nucleótido de adenina en un bolsillo no polar (amarillo, Figura 1) y orientando la molécula a través de enlaces de hidrógeno e interacciones electrostáticas con grupos fosfato. Además de la unión de fosfato mediante bucle P y motivo DFG descrita anteriormente, K483 y E501 desempeñan papeles clave en la estabilización de los grupos fosfato no transferibles. La carga positiva en la amina primaria de K483 le permite estabilizar la carga negativa en los grupos fosfato α y β del ATP cuando se une el ATP. Cuando no hay ATP presente, la carga negativa del grupo carboxilo E501 equilibra esta carga. [17] [18]
Una vez que el ATP se une al dominio de la quinasa B-Raf, el D576 del bucle catalítico activa un grupo hidroxilo del sustrato, lo que aumenta su nucleofilia para impulsar cinéticamente la reacción de fosforilación, mientras que otros residuos del bucle catalítico estabilizan el estado de transición (Figura 2). El N581 forma un quelato del catión magnesio divalente asociado con el ATP para ayudar a orientar la molécula para una sustitución óptima. El K578 neutraliza la carga negativa del grupo γ-fosfato del ATP, de modo que el residuo de sustrato ser/thr activado no experimente tanta repulsión electrón-electrón al atacar al fosfato. Después de que se transfiere el grupo fosfato, se liberan el ADP y la nueva fosfoproteína. [17]
Dado que los mutantes B-Raf constitutivamente activos comúnmente causan cáncer (ver Importancia clínica) al enviar señales excesivas a las células para que crezcan, se han desarrollado inhibidores de B-Raf tanto para las conformaciones inactivas como activas del dominio quinasa como candidatos terapéuticos contra el cáncer. [18] [19] [20]

BAY43-9006 ( Sorafenib , Nexavar) es un inhibidor de B-Raf y C-Raf mutante V600E aprobado por la FDA para el tratamiento del cáncer primario de hígado y riñón . Bay43-9006 desactiva el dominio de la cinasa B-Raf bloqueando la enzima en su forma inactiva. El inhibidor logra esto bloqueando el bolsillo de unión de ATP a través de una alta afinidad por el dominio de la cinasa. Luego se une al bucle de activación clave y a los residuos del motivo DFG para detener el movimiento del bucle de activación y del motivo DFG a la conformación activa. Finalmente, una fracción de fenilo trifluorometilo bloquea estéricamente el motivo DFG y el sitio de conformación activa del bucle de activación, lo que hace imposible que el dominio de la cinasa cambie de conformación para volverse activo. [18]
El anillo piridilo distal de BAY43-9006 se ancla en el bolsillo de unión de nucleótidos hidrofóbico del lóbulo N de la quinasa, interactuando con W531, F583 y F595. Las interacciones hidrofóbicas con el bucle catalítico F583 y el motivo DFG F595 estabilizan la conformación inactiva de estas estructuras, disminuyendo la probabilidad de activación de la enzima. La interacción hidrofóbica adicional de K483, L514 y T529 con el anillo fenilo central aumenta la afinidad del dominio de la quinasa por el inhibidor. La interacción hidrofóbica de F595 con el anillo central también disminuye aún más la favorabilidad energética de un cambio de conformación DFG. Finalmente, las interacciones polares de BAY43-9006 con el dominio de la quinasa continúan esta tendencia de aumentar la afinidad de la enzima por el inhibidor y estabilizar los residuos DFG en la conformación inactiva. E501 y C532 unen por enlace de hidrógeno los grupos urea y piridilo del inhibidor respectivamente, mientras que el carbonilo de la urea acepta un enlace de hidrógeno del nitrógeno amida de la cadena principal de D594 para fijar el motivo DFG en su lugar. [18]
La fracción trifluorometil fenilo consolida la favorabilidad termodinámica de la conformación inactiva cuando el dominio quinasa está unido a BAY43-9006 al bloquear estéricamente el bolsillo hidrofóbico entre las hélices αC y αE que el motivo DFG y el bucle de activación habitarían al cambiar a sus ubicaciones en la conformación activa de la proteína. [18]

PLX4032 ( Vemurafenib ) es un inhibidor de B-Raf mutante V600 aprobado por la FDA para el tratamiento del melanoma en etapa avanzada . [13] A diferencia de BAY43-9006 , que inhibe la forma inactiva del dominio quinasa, Vemurafenib inhibe la forma activa "DFG-in" de la quinasa, [19] [20] anclándose firmemente en el sitio de unión de ATP. Al inhibir solo la forma activa de la quinasa, Vemurafenib inhibe selectivamente la proliferación de células con B-Raf no regulado, normalmente aquellas que causan cáncer .
Dado que Vemurafenib solo se diferencia de su precursor, PLX4720, en un anillo de fenilo añadido por razones farmacocinéticas , [20] el modo de acción de PLX4720 es equivalente al de Vemurafenib. PLX4720 tiene buena afinidad por el sitio de unión de ATP en parte porque su región de anclaje, un 7-aza indol bicíclico , solo se diferencia de la adenina natural que ocupa el sitio en dos lugares donde los átomos de nitrógeno han sido reemplazados por carbono. Esto permite que se conserven fuertes interacciones intermoleculares como el enlace de hidrógeno N7 a C532 y el enlace de hidrógeno N1 a Q530. El excelente ajuste dentro del bolsillo hidrofóbico de unión de ATP (C532, W531, T529, L514, A481) también aumenta la afinidad de unión. La unión de hidrógeno del enlazador de cetona con el agua y el difluorofenilo encajan en un segundo bolsillo hidrófobo (A481, V482, K483, V471, I527, T529, L514 y F583) lo que contribuye a la afinidad de unión excepcionalmente alta en general. La unión selectiva al Raf activo se logra mediante el grupo propilo terminal que se une a un bolsillo selectivo de Raf creado por un desplazamiento de la hélice αC. La selectividad para la conformación activa de la quinasa aumenta aún más mediante un grupo sulfonamida desprotonado sensible al pH que se estabiliza mediante la unión de hidrógeno con el péptido de la cadena principal NH de D594 en el estado activo. En el estado inactivo, el grupo sulfonamida del inhibidor interactúa con el carbonilo de la cadena principal de ese residuo, lo que crea repulsión. Por lo tanto, Vemurafenib se une preferentemente al estado activo del dominio de la quinasa de B-Raf. [19] [20]
Las mutaciones del gen BRAF pueden causar enfermedades de dos maneras. En primer lugar, las mutaciones pueden heredarse y causar defectos de nacimiento. En segundo lugar, las mutaciones pueden aparecer más adelante en la vida y causar cáncer, como un oncogén .
Las mutaciones hereditarias en este gen causan el síndrome cardiofaciocutáneo , una enfermedad caracterizada por defectos cardíacos, retraso mental y una apariencia facial distintiva. [25]
Se han encontrado mutaciones en este gen en cánceres, incluido el linfoma no Hodgkin , el cáncer colorrectal , el melanoma maligno , el carcinoma papilar de tiroides , el carcinoma de pulmón de células no pequeñas , el adenocarcinoma de pulmón , tumores cerebrales que incluyen glioblastoma y xantoastrocitoma pleomórfico , así como enfermedades inflamatorias como la enfermedad de Erdheim-Chester . [10]
La mutación V600E del gen BRAF se ha asociado con la leucemia de células pilosas en numerosos estudios y se ha sugerido su uso en la detección del síndrome de Lynch para reducir la cantidad de pacientes que se someten a una secuenciación innecesaria de MLH1 . [26] [27]
Se han identificado más de 30 mutaciones del gen BRAF asociadas con cánceres humanos. La frecuencia de las mutaciones BRAF varía ampliamente en los cánceres humanos, desde más del 80% en melanomas y nevos , hasta tan solo el 0-18% en otros tumores , como el 1-3% en cánceres de pulmón y el 5% en cáncer colorrectal . [28] En el 90% de los casos, la timina se sustituye por adenina en el nucleótido 1799. Esto lleva a que la valina (V) sea sustituida por glutamato (E) en el codón 600 (ahora denominado V600E ) en el segmento de activación que se ha encontrado en cánceres humanos. [29] Esta mutación se ha observado ampliamente en carcinoma papilar de tiroides , cáncer colorrectal, melanoma y cáncer de pulmón de células no pequeñas . [30] [31] [32] [33] [34] [35] [36] La mutación BRAF-V600E está presente en el 57% de los pacientes con histiocitosis de células de Langerhans. [37] La mutación V600E es una probable mutación impulsora en el 100% de los casos de leucemia de células pilosas . [38] Se ha detectado una alta frecuencia de mutaciones BRAF V600E en el ameloblastoma , una neoplasia odontogénica benigna pero localmente infiltrante. [39] La mutación V600E también puede estar vinculada, como una mutación impulsora única (una "pistola humeante" genética), a ciertos casos de desarrollo de craneofaringioma papilar . [40]
Otras mutaciones que se han encontrado son R461I, I462S, G463E, G463V, G465A, G465E, G465V, G468A, G468E, G469R, N580S, E585K, D593V, F594L, G595R, L596V, T598I, V599D, V599E, V599K, V599R, V600K, A727V, etc. y la mayoría de estas mutaciones se agrupan en dos regiones: el bucle P rico en glicina del lóbulo N y el segmento de activación y las regiones flanqueantes. [18] Estas mutaciones cambian el segmento de activación del estado inactivo al estado activo, por ejemplo, en el artículo citado anteriormente se informó que la cadena lateral alifática de Val599 interactúa con el anillo de fenilo de Phe467 en el bucle P. Se esperaría que reemplazar la cadena lateral hidrofóbica de tamaño mediano de Val con un residuo más grande y cargado como el que se encuentra en el cáncer humano (Glu, Asp, Lys o Arg) desestabilice las interacciones que mantienen el motivo DFG en una conformación inactiva, cambiando así el segmento de activación a la posición activa. Dependiendo del tipo de mutación, la actividad de la quinasa hacia MEK también puede variar. La mayoría de los mutantes estimulan una actividad mejorada de la quinasa B-Raf hacia MEK. Sin embargo, algunos mutantes actúan a través de un mecanismo diferente porque, aunque su actividad hacia MEK se reduce, adoptan una conformación que activa C-RAF de tipo salvaje, que luego envía señales a ERK .
Como se mencionó anteriormente, algunas empresas farmacéuticas están desarrollando inhibidores específicos de la proteína B-raf mutada para uso anticancerígeno porque BRAF es un objetivo bien entendido y de alto rendimiento. [19] [42] Vemurafenib (RG7204 o PLX4032) fue autorizado por la Administración de Alimentos y Medicamentos de los EE. UU . como Zelboraf para el tratamiento del melanoma metastásico en agosto de 2011 con base en datos clínicos de Fase III. Se observó una mejor supervivencia, así como una tasa de respuesta al tratamiento del 53%, en comparación con el 7-12% con el mejor tratamiento quimioterapéutico anterior, dacarbazina . [43] En ensayos clínicos, B-Raf aumentó la probabilidad de supervivencia del paciente con melanoma metastásico. A pesar de la alta eficacia del fármaco, el 20% de los tumores aún desarrollan resistencia al tratamiento. En ratones, el 20% de los tumores se vuelven resistentes después de 56 días. [44] Aunque los mecanismos de esta resistencia aún son objeto de controversia, algunas hipótesis incluyen la sobreexpresión de B-Raf para compensar las altas concentraciones de Vemurafenib [44] y la regulación positiva de la señalización de crecimiento. [45]
Los inhibidores de B-Raf más generales incluyen GDC-0879, PLX-4720, sorafenib , dabrafenib y encorafenib .
El belvarafenib se clasifica como un inhibidor de panRAF. Un inhibidor de panRAF bloquea la función catalítica de ambas proteínas en el dímero. [46]
Se ha demostrado que el gen BRAF interactúa con:
![]() Este artículo incorpora material de dominio público del Diccionario de términos sobre el cáncer. Instituto Nacional del Cáncer de EE. UU .Este artículo incorpora texto de la Biblioteca Nacional de Medicina de los Estados Unidos , que se encuentra en el dominio público .
Este artículo incorpora material de dominio público del Diccionario de términos sobre el cáncer. Instituto Nacional del Cáncer de EE. UU .Este artículo incorpora texto de la Biblioteca Nacional de Medicina de los Estados Unidos , que se encuentra en el dominio público .