Papillomaviridae es una familia de virus de ADN sin envoltura cuyos miembros se conocen como papilomavirus. [1] Se han identificadovarios cientos de especies de virus del papiloma, tradicionalmente denominados "tipos", [2] que infectan a todos los mamíferos cuidadosamente examinados, [2] pero también a otros vertebrados como aves, serpientes, tortugas y peces. [3] [4] [5] La infección por la mayoría de los tipos de virus del papiloma, según el tipo, es asintomática (p. ej., la mayoría de los Beta-PV) o causa pequeños tumores benignos, conocidos como papilomas o verrugas (p. ej., virus del papiloma humano 1, VPH6 o VPH11). ). Sin embargo, los papilomas causados por algunos tipos, como los virus del papiloma humano 16 y 18, conllevan el riesgo de volverse cancerosos . [6]
Los virus del papiloma generalmente se consideran altamente trópicos para el huésped y los tejidos , y se cree que rara vez se transmiten entre especies. [7] Los virus del papiloma se replican exclusivamente en la capa basal de los tejidos de la superficie corporal . Todos los tipos de virus del papiloma conocidos infectan una superficie corporal particular, [2] típicamente la piel o el epitelio de la mucosa de los genitales, el ano, la boca o las vías respiratorias. [8] Por ejemplo, el virus del papiloma humano (VPH) tipo 1 tiende a infectar las plantas de los pies, y el VPH tipo 2, las palmas de las manos, donde pueden causar verrugas . Además, existen descripciones de la presencia de ADN del virus del papiloma en la sangre y en las células mononucleares de la sangre periférica .
Los virus del papiloma se identificaron por primera vez a principios del siglo XX, cuando se demostró que las verrugas cutáneas , o papilomas , podían transmitirse entre individuos mediante un agente infeccioso filtrable. En 1935, Francis Peyton Rous , que había demostrado previamente la existencia de un virus del sarcoma cancerígeno en pollos, pasó a demostrar que un virus del papiloma podía provocar cáncer de piel en conejos infectados. Esta fue la primera demostración de que un virus podría causar cáncer en mamíferos.

Hay más de 100 especies de virus del papiloma reconocidas, [9] aunque el ICTV reconoce oficialmente un número menor, categorizado en 53 géneros, a partir de 2019. [10] [11] [12] Todos los virus del papiloma (PV) tienen organizaciones genómicas similares y cualquier par de PV contiene al menos cinco genes homólogos , aunque la secuencia de nucleótidos puede divergir en más del 50%. Los algoritmos filogenéticos que permiten la comparación de homologías condujeron a árboles filogenéticos que tienen una topología similar, independientemente del gen analizado. [13]
Los estudios filogenéticos sugieren firmemente que las PV normalmente evolucionan junto con sus especies de mamíferos y aves hospedantes, pero las radiaciones adaptativas , los eventos zoonóticos ocasionales y las recombinaciones también pueden afectar su diversificación. [13] Su organización genómica básica parece mantenida durante un período superior a 100 millones de años, y estas comparaciones de secuencias han sentado las bases para una taxonomía PV, que ahora está reconocida oficialmente por el Comité Internacional sobre Taxonomía de Virus . Todos los PV forman la familia Papillomaviridae , que es distinta de Polyomaviridae, eliminando así el término Papovaviridae . Las ramas principales del árbol filogenético de las PV se consideran géneros , que se identifican con letras griegas. Las ramas menores se consideran especies y unen tipos de PV que son genómicamente distintos sin exhibir diferencias biológicas conocidas. Este nuevo sistema taxonómico no afecta la identificación y caracterización tradicional de los "tipos" de PV y sus aislamientos independientes con diferencias genómicas menores, denominados "subtipos" y "variantes", todos los cuales son taxones por debajo del nivel de "especie". [14] Además, se han propuesto agrupaciones filogenéticas en un nivel taxonómico superior. [15]
Esta clasificación puede necesitar revisión a la luz de la existencia de recombinantes del virus del papiloma-polioma. [16] También se han descrito especies adicionales. Se ha aislado el virus del papiloma Sparus aurata 1 de peces. [17]
Se han secuenciado completamente más de 170 tipos de virus del papiloma humano. [18] Se han dividido en 5 géneros: Alphapapillomavirus, Betapapillomavirus, Gammapapillomavirus, Mupapillomavirus y Nupapillomavirus. Se han identificado al menos 200 virus adicionales que esperan secuenciación y clasificación. [ cita necesaria ]

Los tipos individuales de papilomavirus tienden a estar altamente adaptados a la replicación en una sola especie animal. En un estudio, los investigadores tomaron muestras de la piel de la frente de una variedad de animales del zoológico y utilizaron PCR para amplificar cualquier ADN del virus del papiloma que pudiera estar presente. [19] Aunque en el estudio se identificó una amplia variedad de secuencias de virus del papiloma, los autores encontraron poca evidencia de transmisión entre especies. Se descubrió que un cuidador del zoológico dio positivo transitoriamente a una secuencia del virus del papiloma específico de chimpancé. Sin embargo, los autores señalan que la secuencia del virus del papiloma específico del chimpancé podría haber sido el resultado de la contaminación superficial de la piel del cuidador del zoológico, en lugar de una infección productiva. [ cita necesaria ]
El virus del papiloma del conejo cola de algodón (CRPV) puede causar verrugas protuberantes en su huésped nativo, el género de conejos norteamericano Sylvilagus . Estas verrugas con forma de cuerno pueden ser la base original de las leyendas urbanas del conejo astado americano Jackalope y el Wolpertinger europeo . [20] Los conejos domésticos europeos (género Oryctolagus ) pueden infectarse transitoriamente con CRPV en un laboratorio. Sin embargo, dado que los conejos domésticos europeos no producen una progenie de virus infeccioso, se los considera un huésped incidental o "callejón sin salida" para el CRPV. [21]
También se ha documentado la transmisión entre especies del virus del papiloma bovino (BPV) tipo 1. [22] En su huésped natural (bovino), el BPV-1 induce grandes verrugas cutáneas fibrosas. La infección por BPV-1 de los caballos, que son huéspedes incidentales del virus, puede provocar el desarrollo de tumores benignos conocidos como sarcoides . La importancia agrícola del BPV-1 impulsó un esfuerzo exitoso para desarrollar una vacuna contra el virus. [ cita necesaria ]
Algunos informes han identificado virus del papiloma en roedores más pequeños, como los hámsteres sirios , la rata multimamífera africana y el ratón recolector euroasiático . [23] Sin embargo, no se sabe que ningún virus del papiloma sea capaz de infectar a ratones de laboratorio . La falta de un modelo de ratón tratable para la infección por papilomavirus ha sido una limitación importante para la investigación de laboratorio de los papilomavirus. [ cita necesaria ]
Se sabe que cuatro virus del papiloma infectan a las aves: el virus del papiloma 1 de Fringilla coelebs, el virus del papiloma 1 de Francolinus leucoscepus, el virus del papiloma 1 de Psittacus erithacus y el virus 1 del papiloma de Pygoscelis adeliae. [24] Todas estas especies tienen un gen (E9) de función desconocida, lo que sugiere un origen común.
Se cree que la evolución de los virus del papiloma es lenta en comparación con muchos otros tipos de virus, pero actualmente no hay mediciones experimentales disponibles. Probablemente esto se deba a que el genoma del virus del papiloma está compuesto de ADN bicatenario genéticamente estable que se replica con alta fidelidad mediante la maquinaria de replicación del ADN de la célula huésped. [ cita necesaria ]
Se cree que los virus del papiloma generalmente coevolucionan con una especie particular de animal huésped durante muchos años, aunque existen pruebas sólidas en contra de la hipótesis de la coevolución. [13] [25] En un ejemplo particularmente rápido, el VPH-16 ha evolucionado ligeramente a medida que las poblaciones humanas se han expandido por todo el mundo y ahora varía en diferentes regiones geográficas de una manera que probablemente refleja la historia de la migración humana. [26] [27] Los tipos cutaneotrópicos de VPH se intercambian ocasionalmente entre miembros de la familia durante toda la vida, pero también se deben considerar otros donantes en la transmisión viral. [28]
Otros tipos de VPH, como el VPH-13, varían relativamente poco en diferentes poblaciones humanas. De hecho, la secuencia del VPH-13 se parece mucho al virus del papiloma de los bonobos (también conocidos como chimpancés pigmeos). [29] No está claro si esta similitud se debe a la transmisión reciente entre especies o porque el VPH-13 simplemente ha cambiado muy poco en los aproximadamente seis millones de años transcurridos desde que los humanos y los bonobos divergieron. [27]
Se estima que el ancestro común más reciente de este grupo de virus existió hace 424 millones de años . [30]
Hay cinco géneros principales que infectan a los humanos (Alfa, Beta, Gamma, Mu y Nu). El ancestro común más reciente de estos géneros evolucionó hace 49,7 millones de años ( hace 58,5 millones de años) . [31] Se estima que el ancestro más reciente del género gamma evolucionó hace entre 45,3 millones de años y 67,5 millones de años . [ cita necesaria ]
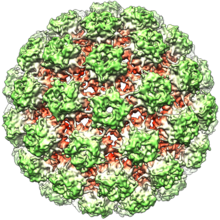
Los virus del papiloma no tienen envoltura, lo que significa que la capa exterior o cápside del virus no está cubierta por una membrana lipídica . Una única proteína viral, conocida como L1, es necesaria y suficiente para la formación de una cápside de 55 a 60 nanómetros compuesta por 72 capsómeros en forma de estrella (ver figura). Como la mayoría de los virus sin envoltura, la cápside es geométricamente regular y presenta simetría icosaédrica . Las partículas autoensambladas similares a virus compuestas de L1 son la base de un grupo exitoso de vacunas profilácticas contra el VPH diseñadas para provocar anticuerpos neutralizantes del virus que protegen contra la infección inicial por VPH. Como tales, los papilomaviridas son estables en el medio ambiente . [ cita necesaria ]
El genoma del virus del papiloma es una molécula de ADN circular bicatenario de aproximadamente 8.000 pares de bases de longitud. Está empaquetado dentro de la capa L1 junto con proteínas histonas celulares , que sirven para envolver y condensar el ADN. [ cita necesaria ]
La cápside del virus del papiloma también contiene una proteína viral conocida como L2, que es menos abundante. Aunque no está claro cómo se organiza L2 dentro del virión, se sabe que realiza varias funciones importantes, incluida la facilitación del empaquetado del genoma viral en viriones nacientes, así como la entrada infecciosa del virus en nuevas células huésped. L2 es de interés como posible objetivo para vacunas contra el VPH con una protección más amplia .
La cápside viral consta de 72 capsómeros de los cuales 12 tienen cinco coordinaciones y 60 son capsómeros de seis coordinadas, dispuestos en una red de superficie icosaédrica T = 7d. [32]
Los virus del papiloma se replican exclusivamente en los queratinocitos . Los queratinocitos forman las capas más externas de la piel, así como algunas superficies mucosas , como el interior de la mejilla o las paredes de la vagina. Estos tejidos superficiales, que se conocen como epitelios escamosos estratificados , están compuestos por capas apiladas de células aplanadas. Las capas celulares se forman mediante un proceso conocido como diferenciación celular , en el que los queratinocitos se especializan gradualmente hasta formar una superficie dura y reticulada que evita la pérdida de humedad y actúa como barrera contra los patógenos. Se cree que las células madre de queratinocitos menos diferenciados, que se reponen en la capa superficial, son el objetivo inicial de las infecciones productivas por virus del papiloma. Los pasos posteriores en el ciclo de vida viral dependen estrictamente del proceso de diferenciación de los queratinocitos. Como resultado, los virus del papiloma sólo pueden replicarse en los tejidos de la superficie corporal. [ cita necesaria ]
Los virus del papiloma acceden a las células madre de los queratinocitos a través de pequeñas heridas, conocidas como microtraumatismos, en la piel o la superficie de las mucosas. Las interacciones entre L1 y los azúcares sulfatados en la superficie celular promueven la adhesión inicial del virus. [33] [34] Luego, el virus puede ingresar desde la superficie celular mediante la interacción con un receptor específico, probablemente a través de la integrina alfa-6 beta-4, [35] [36] y transportarse a vesículas rodeadas de membrana llamadas endosomas . [37] [38] La proteína de la cápside L2 altera la membrana del endosoma a través de un péptido catiónico que penetra en las células , lo que permite que el genoma viral escape y circule, junto con L2, hacia el núcleo celular. [39] [40] [41]
Después de una infección exitosa de un queratinocito, el virus expresa las proteínas E1 y E2, que sirven para replicar y mantener el ADN viral como un episoma circular . Los oncogenes virales E6 y E7 promueven el crecimiento celular al inactivar las proteínas supresoras de tumores p53 y pRb . Las células madre de queratinocitos en la capa basal epitelial pueden mantener los genomas del virus del papiloma durante décadas. [8]
El conocimiento actual es que la replicación del ADN viral probablemente ocurre en la fase G 2 del ciclo celular y depende de la replicación dependiente de la recombinación respaldada por mecanismos de respuesta al daño del ADN (activados por la proteína E7) para producir genomas virales de progenie. [42] Los genomas del virus del papiloma a veces se integran en el genoma del huésped, especialmente en el caso de los VPH oncogénicos, pero no son una parte normal del ciclo de vida del virus y son un callejón sin salida que elimina el potencial de producción de progenie viral. [42]
La expresión de los genes tardíos virales, L1 y L2, se restringe exclusivamente a los queratinocitos diferenciadores en las capas más externas de la piel o la superficie mucosa. El aumento de la expresión de L1 y L2 suele correlacionarse con un aumento espectacular en el número de copias del genoma viral. Dado que las capas externas de los epitelios escamosos estratificados están sujetas a una vigilancia relativamente limitada por parte de las células del sistema inmunitario, se cree que esta restricción de la expresión genética tardía del virus representa una forma de evasión inmunitaria. [ cita necesaria ]
En el núcleo celular se ensamblan nuevos virus descendientes de virus infecciosos . Los virus del papiloma han desarrollado un mecanismo para liberar viriones al medio ambiente. Otros tipos de virus animales sin envoltura utilizan un proceso lítico activo para matar la célula huésped, lo que permite la liberación de partículas de virus descendientes. A menudo, este proceso lítico se asocia con inflamación , lo que podría desencadenar un ataque inmunológico contra el virus. Los virus del papiloma aprovechan la descamación como un mecanismo de liberación sigiloso y no inflamatorio. [ cita necesaria ]
Aunque algunos tipos de virus del papiloma pueden causar cáncer en los tejidos epiteliales que habitan, el cáncer no es un resultado típico de la infección. El desarrollo de cánceres inducidos por el virus del papiloma suele ocurrir a lo largo de muchos años. Los virus del papiloma se han asociado con el desarrollo de cáncer de cuello uterino , cáncer de pene [43] y cánceres orales . [44] También se ha observado una asociación con el cáncer de vulva y el carcinoma urotelial con diferenciación escamosa en pacientes con vejiga neurogénica. [45] [46] Hay un genoma del virus del papiloma que causa cáncer y que codifica dos pequeñas proteínas llamadas E6 y E7 que imitan los oncogenes que causan el cáncer. La forma en que funcionan es que estimulan el crecimiento antinatural de las células y bloquean sus defensas naturales. También actúan sobre muchas proteínas de señalización que controlan la proliferación y la apoptosis. [47]
El hecho de que el ciclo de vida del virus del papiloma requiera estrictamente la diferenciación de queratinocitos ha planteado una barrera sustancial para el estudio de los virus del papiloma en el laboratorio, ya que ha impedido el uso de líneas celulares convencionales para cultivar los virus. Debido a que los viriones infecciosos BPV-1 pueden extraerse de las grandes verrugas que el virus induce en el ganado, ha sido un tipo de virus del papiloma modelo caballo de batalla durante muchos años. El CRPV, el virus del papiloma oral de conejo (ROPV) y el virus del papiloma oral canino (COPV) también se han utilizado ampliamente para estudios de laboratorio. Tan pronto como los investigadores descubrieron que estos virus causan cáncer, trabajaron juntos para encontrar una vacuna. Actualmente, la forma más eficaz de hacerlo es imitar un virus que está compuesto de proteína L1 pero que carece de ADN. Básicamente, nuestro sistema inmunológico construye defensas contra las infecciones, pero si estas infecciones no causan enfermedades, pueden usarse como vacuna. La entrada 6bt3 del PDB muestra cómo las superficies de los anticuerpos atacan la superficie del virus para desactivarlo. [48]
Algunos tipos de VPH de transmisión sexual se han propagado utilizando un sistema de "xenoinjerto" de ratón, en el que se implantan células humanas infectadas con VPH en ratones inmunodeficientes . Más recientemente, algunos grupos han logrado aislar el VPH-16 infeccioso de lesiones cervicales humanas. Sin embargo, el aislamiento de viriones infecciosos mediante esta técnica es arduo y la producción de virus infecciosos es muy baja. [ cita necesaria ]
La diferenciación de queratinocitos se puede imitar in vitro exponiendo queratinocitos cultivados a una interfaz aire/líquido. La adaptación de estos sistemas de "cultivo en balsa" al estudio de los virus del papiloma supuso un avance importante para el estudio in vitro del ciclo de vida viral. [49] Sin embargo, los sistemas de cultivo en balsas son relativamente engorrosos y el rendimiento de VPH infecciosos puede ser bajo. [50]
El desarrollo de un sistema basado en levaduras que permite la replicación episomal estable del VPH proporciona un medio conveniente, rápido y económico para estudiar varios aspectos del ciclo de vida del VPH (Angeletti 2002). Por ejemplo, la transcripción dependiente de E2, la amplificación del genoma y la encapsidación eficiente de los ADN del VPH de longitud completa se pueden recrear fácilmente en levaduras (Angeletti 2005).
Recientemente, se han desarrollado métodos transitorios de alto rendimiento para producir pseudovirus del VPH que portan genes indicadores. Aunque los pseudovirus no son adecuados para estudiar ciertos aspectos del ciclo de vida viral, los estudios iniciales sugieren que su estructura y entrada infecciosa inicial en las células es probablemente similar en muchos aspectos a los auténticos virus del papiloma.
El virus del papiloma humano se une a las moléculas de heparina en la superficie de las células que infecta. Los estudios han demostrado que el cristal de capsómeros L1 aislados tiene cadenas de heparina reconocidas por líneas de lisinas en los surcos de la superficie del virus. También aquellos que tienen los anticuerpos demuestran que pueden bloquear este reconocimiento. [51]
[52]
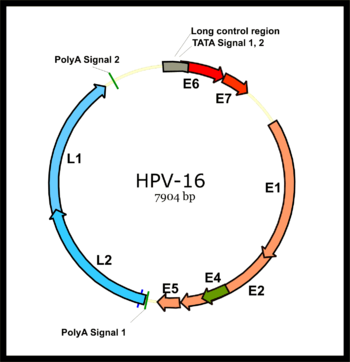
El genoma del virus del papiloma se divide en una región temprana (E), que codifica seis marcos de lectura abiertos (ORF) (E1, E2, E4, E5, E6 y E7) que se expresan inmediatamente después de la infección inicial de una célula huésped, y una región tardía. región (L) que codifica una proteína de la cápsida mayor L1 y una proteína de la cápsida menor L2. Todos los ORF virales están codificados en una cadena de ADN (ver figura). Esto representa una diferencia dramática entre los papilomavirus y los poliomavirus , ya que este último tipo de virus expresa sus genes tempranos y tardíos mediante la transcripción bidireccional de ambas cadenas de ADN. Esta diferencia fue un factor importante en el establecimiento del consenso de que los papilomavirus y los poliomavirus probablemente nunca compartieron un ancestro común, a pesar de las sorprendentes similitudes en las estructuras de sus viriones. [ cita necesaria ]
Una vez infectada la célula huésped, se activa el promotor temprano del VPH16 y se transcribe un ARN primario policistrónico que contiene los seis ORF tempranos. Este ARN policistrónico contiene tres exones y dos intrones y se somete a un empalme activo de ARN para generar múltiples isoformas de ARNm. [52] Uno de los ARN de isoforma empalmados, E6*I, sirve como ARNm de E7 para traducir la oncoproteína E7. [53] Por el contrario, un intrón en el ORF E6 que permanece intacto sin corte y empalme es necesario para la traducción de la oncoproteína E6. [53] Sin embargo, la transcripción temprana viral está sujeta a la regulación viral de E2 y los niveles altos de E2 reprimen la transcripción. Los genomas del VPH se integran en el genoma del huésped mediante la alteración del ORF de E2, evitando la represión de E2 en E6 y E7. Por lo tanto, la integración del genoma viral en el genoma del ADN del huésped aumenta la expresión de E6 y E7 para promover la proliferación celular y la posibilidad de malignidad. [ cita necesaria ]
Un importante promotor tardío viral en la región temprana viral se vuelve activo solo en células diferenciadas y su actividad puede mejorarse en gran medida mediante la replicación del ADN viral. El transcrito tardío es también un ARN policistrónico que contiene dos intrones y tres exones. El empalme de ARN alternativo de esta transcripción tardía es esencial para la expresión de L1 y L2 y puede regularse mediante elementos cis de ARN y factores de empalme del huésped. [52] [54] [55]
Los genes dentro del genoma del virus del papiloma generalmente se identifican por similitud con otros genes previamente identificados. Sin embargo, algunos marcos de lectura abiertos espurios podrían haberse confundido con genes simplemente después de su posición en el genoma, y podrían no ser genes verdaderos. Esto se aplica especialmente a ciertos marcos de lectura abiertos E3, E4, E5 y E8 . [ cita necesaria ]
Codifica una proteína que se une al origen de replicación viral en la región de control larga del genoma viral. E1 utiliza ATP para ejercer una actividad helicasa que separa las cadenas de ADN, preparando así el genoma viral para la replicación mediante factores de replicación del ADN celular .
La proteína E2 sirve como regulador transcripcional maestro para los promotores virales ubicados principalmente en la región de control larga. La proteína tiene un dominio de transactivación unido por una región bisagra relativamente no estructurada a un dominio de unión al ADN bien caracterizado. E2 facilita la unión de E1 al origen de replicación viral. E2 también utiliza una proteína celular conocida como Bromodominio -4 (Brd4) para unir el genoma viral a los cromosomas celulares. [56] Esta unión a la matriz nuclear de la célula garantiza una distribución fiel de los genomas virales a cada célula hija después de la división celular. Se cree que E2 sirve como regulador negativo de la expresión de los oncogenes E6 y E7 en queratinocitos de la capa basal infectados de forma latente por VPH . Los cambios genéticos, como la integración del ADN viral en el cromosoma de la célula huésped, que inactivan la expresión de E2, tienden a aumentar la expresión de los oncogenes E6 y E7, lo que da como resultado una transformación celular y posiblemente una mayor desestabilización genética.
Este pequeño gen putativo existe sólo en unos pocos tipos de virus del papiloma. No se sabe que el gen se exprese como una proteína y no parece cumplir ninguna función.
Aunque las proteínas E4 se expresan en niveles bajos durante la fase temprana de la infección viral, la expresión de E4 aumenta dramáticamente durante la fase tardía de la infección. En otras palabras, su denominación "E" puede ser un nombre poco apropiado. En el caso del VPH-1, la E4 puede representar hasta el 30% de la proteína total en la superficie de una verruga. [57] Se cree que la proteína E4 de muchos tipos de virus del papiloma facilita la liberación del virión al medio ambiente al alterar los filamentos intermedios del citoesqueleto de los queratinocitos . Los mutantes virales incapaces de expresar E4 no apoyan la replicación de alto nivel del ADN viral, pero aún no está claro cómo E4 facilita la replicación del ADN. También se ha demostrado que E4 participa en la detención de células en la fase G2 del ciclo celular .
Las E5 son proteínas pequeñas y muy hidrofóbicas que desestabilizan la función de muchas proteínas de membrana en la célula infectada. [58] La proteína E5 de algunos tipos de virus del papiloma animal (principalmente el virus del papiloma bovino tipo 1) funciona como un oncogén principalmente activando la señalización de los receptores del factor de crecimiento derivados de plaquetas que promueve el crecimiento celular . Sin embargo, las proteínas E5 de los virus del papiloma humano asociados al cáncer parecen activar la cascada de señales iniciada por el factor de crecimiento epidérmico tras la unión del ligando. También se ha demostrado que HPV16 E5 y HPV2 E5 regulan negativamente la expresión superficial de las proteínas de clase I del complejo mayor de histocompatibilidad , lo que puede impedir que las células T asesinas eliminen la célula infectada .

E6 es un péptido de 151 aminoácidos que incorpora un motivo tipo 1 con una secuencia consenso –(T/S)-(X)-(V/I)-COOH. [60] [61] También tiene dos motivos de dedos de zinc . [60]
E6 es de particular interés porque parece tener múltiples funciones en la célula e interactuar con muchas otras proteínas. Sin embargo, su función principal es mediar en la degradación de p53 , una importante proteína supresora de tumores , reduciendo la capacidad de la célula para responder al daño del ADN . [62] [63]
También se ha demostrado que E6 se dirige a otras proteínas celulares, alterando así varias vías metabólicas . Uno de esos objetivos es NFX1-91 , que normalmente reprime la producción de telomerasa , una proteína que permite que las células se dividan un número ilimitado de veces. Cuando E6 degrada NFX1-91, los niveles de telomerasa aumentan, inactivando un mecanismo importante que mantiene bajo control el crecimiento celular. [64] Además, E6 puede actuar como cofactor transcripcional, específicamente, un activador de la transcripción, cuando interactúa con el factor de transcripción celular, E2F1/DP1. [60]
E6 también puede unirse a dominios PDZ , secuencias cortas que a menudo se encuentran en proteínas de señalización. El motivo estructural de E6 permite la interacción con dominios PDZ en genes supresores de tumores DLG (discos grandes) y hDLG (Drosophila grande). [61] [65] La unión en estos lugares provoca la transformación de la proteína DLG y la alteración de su función supresora. Las proteínas E6 también interactúan con las proteínas MAGUK (familia de guanilato quinasas asociadas a membranas). Estas proteínas, incluidas MAGI-1, MAGI-2 y MAGI-3, suelen ser proteínas estructurales y pueden ayudar con la señalización. [61] [65] Más significativamente, se cree que están involucrados con la actividad supresora de DLG. Cuando E6 forma complejos con los dominios PDZ de las proteínas MAGI, distorsiona su forma y, por lo tanto, impide su función. En general, la proteína E6 sirve para impedir la actividad normal de la proteína de tal manera que permite que una célula crezca y se multiplique al ritmo acelerado característico del cáncer.
Dado que la expresión de E6 es estrictamente necesaria para el mantenimiento de un fenotipo maligno en los cánceres inducidos por el VPH, es un objetivo atractivo para las vacunas terapéuticas contra el VPH diseñadas para erradicar los tumores de cáncer de cuello uterino establecidos.
En la mayoría de los tipos de virus del papiloma, la función principal de la proteína E7 es inactivar miembros de la familia pRb de proteínas supresoras de tumores. Junto con E6, E7 sirve para prevenir la muerte celular ( apoptosis ) y promover la progresión del ciclo celular , preparando así a la célula para la replicación del ADN viral. E7 también participa en la inmortalización de células infectadas activando la telomerasa celular . Al igual que el E6, el E7 es objeto de intenso interés en investigación y se cree que ejerce una amplia variedad de otros efectos sobre las células infectadas. Al igual que con E6, la expresión continua de E7 es necesaria para la supervivencia de líneas celulares cancerosas, como HeLa , que se derivan de tumores inducidos por el VPH. [66]
Sólo unos pocos tipos de virus del papiloma codifican una proteína corta del gen E8. En el caso de BPV-4 (género de papilomavirus Xi ), el marco de lectura abierto E8 puede sustituir al marco de lectura abierto E6, que está ausente en este género de papilomavirus. [67] Estos genes E8 son química y funcionalmente similares a los genes E5 de algunos virus del papiloma humano y también se denominan E5/E8.
L1 se autoensambla espontáneamente en capsómeros pentamericanos. Los capsómeros purificados pueden formar cápsides, que se estabilizan mediante enlaces disulfuro entre moléculas L1 vecinas. Las cápsides L1 ensambladas in vitro son la base de las vacunas profilácticas contra varios tipos de VPH. En comparación con otros genes del virus del papiloma, las secuencias de aminoácidos de la mayoría de las porciones de L1 están bien conservadas entre los tipos. Sin embargo, los bucles superficiales de L1 pueden diferir sustancialmente, incluso para diferentes miembros de una especie de papilomavirus particular. Esto probablemente refleja un mecanismo de evasión de respuestas de anticuerpos neutralizantes provocadas por infecciones previas por virus del papiloma. [68]
L2 existe en estado oxidado dentro del virión del virus del papiloma, y los dos residuos de cisteína conservados forman un enlace disulfuro intramolecular . [69] Además de cooperar con L1 para empaquetar el ADN viral en el virión, se ha demostrado que L2 interactúa con varias proteínas celulares durante el proceso de entrada infecciosa. Después de la unión inicial del virión a la célula, la proteasa celular furina debe escindir L2 . [70] El virión se internaliza, probablemente a través de un proceso mediado por clatrina, en un endosoma, donde se cree que las condiciones ácidas conducen a la exposición de porciones de L2 que desestabilizan la membrana. [39] Las proteínas celulares beta- actina [71] y sintaxina -18 [72] también pueden participar en eventos de entrada mediados por L2. Después del escape del endosoma, L2 y el genoma viral se importan al núcleo celular, donde viajan a un dominio subnuclear conocido como cuerpo ND-10 que es rico en factores de transcripción . [40] Pequeñas porciones de L2 están bien conservadas entre diferentes tipos de virus del papiloma, y las vacunas experimentales dirigidas a estos dominios conservados pueden ofrecer protección contra una amplia gama de tipos de VPH. [73]