La biología molecular / m ə ˈ l ɛ k j ʊ l ər / es una rama de la biología que busca comprender las bases moleculares de la actividad biológica en y entre las células , incluida la síntesis, modificación, mecanismos e interacciones biomoleculares . [1] [2] [3]
La biología molecular se describió por primera vez como un enfoque centrado en los fundamentos de los fenómenos biológicos: descubrir las estructuras de las moléculas biológicas, así como sus interacciones, y cómo estas interacciones explican las observaciones de la biología clásica. [4]
El término biología molecular fue utilizado por primera vez en 1945 por el físico William Astbury . En 1953, Francis Crick , James Watson , Rosalind Franklin y sus colegas que trabajaban en la Unidad del Consejo de Investigación Médica del Laboratorio Cavendish crearon el modelo de doble hélice del ADN . Propusieron la estructura del ADN basándose en investigaciones previas realizadas por Franklin y Maurice Wilkins . Esto llevó al descubrimiento de material de ADN en otros microorganismos, plantas y animales. [5]
El campo de la biología molecular incluye técnicas que permiten a los científicos aprender sobre los procesos moleculares. [6] Estas técnicas se utilizan para atacar de manera eficiente nuevos medicamentos , diagnosticar enfermedades y comprender mejor la fisiología celular. [7] Algunas investigaciones clínicas y terapias médicas que surgen de la biología molecular están cubiertas por la terapia génica , mientras que el uso de la biología molecular o la biología celular molecular en la medicina ahora se conoce como medicina molecular .


La biología molecular se encuentra en la intersección de la bioquímica y la genética; A medida que estas disciplinas científicas surgieron y evolucionaron en el siglo XX, quedó claro que ambas buscaban determinar los mecanismos moleculares que subyacen a las funciones celulares vitales. [8] Los avances en biología molecular han estado estrechamente relacionados con el desarrollo de nuevas tecnologías y su optimización. [9] La biología molecular ha sido dilucidada por el trabajo de muchos científicos y, por lo tanto, la historia del campo depende de la comprensión de estos científicos y sus experimentos. [ cita necesaria ]
El campo de la genética surgió como un intento de comprender los mecanismos moleculares de la herencia genética y la estructura de un gen . Gregor Mendel fue pionero en este trabajo en 1866, cuando escribió por primera vez las leyes de la herencia genética basándose en sus estudios de cruces en plantas de guisantes. [10] Una de esas leyes de herencia genética es la ley de segregación , que establece que los individuos diploides con dos alelos para un gen particular transmitirán uno de estos alelos a su descendencia. [11] Debido a su trabajo crítico, el estudio de la herencia genética se conoce comúnmente como genética mendeliana . [12]
Un hito importante en la biología molecular fue el descubrimiento de la estructura del ADN . Este trabajo lo inició en 1869 Friedrich Miescher , un bioquímico suizo que propuso por primera vez una estructura llamada nucleína , que ahora sabemos que es (ácido desoxirribonucleico), o ADN. [13] Descubrió esta sustancia única estudiando los componentes de los vendajes llenos de pus y observando las propiedades únicas de las "sustancias que contienen fósforo". [14] Otro contribuyente notable al modelo de ADN fue Phoebus Levene , quien propuso el "modelo polinucleotídico" de ADN en 1919 como resultado de sus experimentos bioquímicos con levadura. [15] En 1950, Erwin Chargaff amplió el trabajo de Levene y dilucida algunas propiedades críticas de los ácidos nucleicos: primero, la secuencia de los ácidos nucleicos varía según la especie. [16] En segundo lugar, la concentración total de purinas (adenina y guanina) es siempre igual a la concentración total de pirimidinas (cisteína y timina). [13] Esto ahora se conoce como regla de Chargaff. En 1953, James Watson y Francis Crick publicaron la estructura de doble hélice del ADN, [17] utilizando el trabajo de cristalografía de rayos X realizado por Rosalind Franklin y Maurice Wilkins . Watson y Crick describieron la estructura del ADN y conjeturaron sobre las implicaciones de esta estructura única para los posibles mecanismos de replicación del ADN. [17] Watson y Crick recibieron el Premio Nobel de Fisiología o Medicina en 1962, junto con Wilkins, por proponer un modelo de la estructura del ADN. [5]
En 1961, se demostró que cuando un gen codifica una proteína , tres bases secuenciales del ADN de un gen especifican cada aminoácido sucesivo de la proteína. [18] Así, el código genético es un código triplete, donde cada triplete (llamado codón ) especifica un aminoácido particular. Además, se demostró que los codones no se superponen entre sí en la secuencia de ADN que codifica una proteína y que cada secuencia se lee desde un punto de partida fijo. Durante 1962-1964, mediante el uso de mutantes letales condicionales de un virus bacteriano, [19] se lograron avances fundamentales en nuestra comprensión de las funciones e interacciones de las proteínas empleadas en la maquinaria de replicación y reparación del ADN , recombinación del ADN y en el ensamblaje de estructuras moleculares. [20]

En 1928, Frederick Griffith , encontró una propiedad de virulencia en la bacteria neumococo, que estaba matando ratas de laboratorio. Según Mendel, que prevalecía en aquella época, la transferencia de genes sólo podía ocurrir de las células madre a las hijas. Griffith propuso otra teoría, afirmando que la transferencia de genes que ocurre en miembros de la misma generación se conoce como transferencia genética horizontal (THG). Este fenómeno ahora se conoce como transformación genética. [ cita necesaria ]
El experimento de Griffith abordó la bacteria Streptococcus pneumoniae , que tenía dos cepas diferentes, una virulenta y suave y otra avirulenta y rugosa. La cepa suave tenía una apariencia brillante debido a la presencia de un tipo de polisacárido específico: un polímero de glucosa y una cápsula de ácido glucurónico. Debido a esta capa de polisacárido de bacterias, el sistema inmunológico del huésped no puede reconocer las bacterias y mata al huésped. La otra cepa, avirulenta y áspera, carece de esta cápsula de polisacárido y tiene una apariencia opaca y áspera. [ cita necesaria ]
Se sabe que la presencia o ausencia de cápsula en la cepa está determinada genéticamente. Las deformaciones suaves y rugosas se producen en varios tipos diferentes, como SI, S-II, S-III, etc. y RI, R-II, R-III, etc., respectivamente. Todos estos subtipos de bacterias S y R se diferencian entre sí en el tipo de antígeno que producen. [5]

La confirmación de que el ADN es el material genético causante de la infección provino del experimento de Hershey-Chase . Utilizaron E. coli y bacteriófagos para el experimento. Este experimento también se conoce como experimento de la licuadora, ya que se utilizó una licuadora de cocina como aparato principal. Alfred Hershey y Martha Chase demostraron que el ADN inyectado por una partícula de fago en una bacteria contiene toda la información necesaria para sintetizar la progenie de partículas de fago. Utilizaron radioactividad para marcar la cubierta proteica del bacteriófago con azufre radiactivo y el ADN con fósforo radiactivo, en dos tubos de ensayo diferentes, respectivamente. Después de mezclar el bacteriófago y E. coli en el tubo de ensayo, comienza el período de incubación en el que el fago transforma el material genético en las células de E. coli . Luego, la mezcla se mezcla o se agita, lo que separa el fago de las células de E. coli . Se centrifugó toda la mezcla y se comprobó el sedimento que contenía células de E. coli y se descartó el sobrenadante. Las células de E. coli mostraron fósforo radiactivo, lo que indicó que el material transformado era ADN, no la cubierta proteica.
El ADN transformado se une al ADN de E. coli y la radiactividad sólo se ve en el ADN del bacteriófago. Este ADN mutado puede transmitirse a la siguiente generación y surgió la teoría de la transducción. La transducción es un proceso en el que el ADN bacteriano transporta el fragmento de bacteriófagos y lo transmite a la siguiente generación. Este también es un tipo de transferencia horizontal de genes. [5]
A principios de la década de 2020, la biología molecular entró en una época dorada definida por el desarrollo técnico tanto vertical como horizontal. Verticalmente, las nuevas tecnologías están permitiendo el seguimiento en tiempo real de los procesos biológicos a nivel atómico. [21] Los biólogos moleculares hoy en día tienen acceso a datos de secuenciación cada vez más asequibles a profundidades cada vez mayores, lo que facilita el desarrollo de nuevos métodos de manipulación genética en nuevos organismos no modelo. Asimismo, los biólogos moleculares sintéticos impulsarán la producción industrial de moléculas pequeñas y macro mediante la introducción de vías metabólicas exógenas en diversas líneas celulares procarióticas y eucariotas. [22]
Horizontalmente, los datos de secuenciación son cada vez más asequibles y se utilizan en muchos campos científicos diferentes. Esto impulsará el desarrollo de industrias en los países en desarrollo y aumentará la accesibilidad a los investigadores individuales. Del mismo modo, ahora los individuos pueden concebir e implementar experimentos de edición de genes CRISPR-Cas9 por menos de 10.000 dólares en organismos novedosos, lo que impulsará el desarrollo de aplicaciones industriales y médicas. [23]

La siguiente lista describe un punto de vista sobre las relaciones interdisciplinarias entre la biología molecular y otros campos relacionados. [24]
Si bien los investigadores practican técnicas específicas de la biología molecular, es común combinarlas con métodos provenientes de la genética y la bioquímica . Gran parte de la biología molecular es cuantitativa y recientemente se ha realizado una cantidad significativa de trabajo utilizando técnicas informáticas como la bioinformática y la biología computacional . La genética molecular , el estudio de la estructura y función de los genes, ha estado entre los subcampos más destacados de la biología molecular desde principios de la década de 2000. Otras ramas de la biología se basan en la biología molecular, ya sea estudiando directamente las interacciones de las moléculas por derecho propio, como en la biología celular y la biología del desarrollo , o indirectamente, donde se utilizan técnicas moleculares para inferir atributos históricos de poblaciones o especies , como en campos de la biología evolutiva como la genética de poblaciones y la filogenética . También existe una larga tradición de estudiar biomoléculas "desde cero", o molecularmente, en biofísica . [27]

La clonación molecular se utiliza para aislar y luego transferir una secuencia de ADN de interés a un vector plásmido. [28] Esta tecnología de ADN recombinante se desarrolló por primera vez en la década de 1960. [29] En esta técnica, una secuencia de ADN que codifica una proteína de interés se clona mediante la reacción en cadena de la polimerasa (PCR) y/o enzimas de restricción en un plásmido ( vector de expresión ). El vector plásmido suele tener al menos 3 características distintivas: un origen de replicación, un sitio de clonación múltiple (MCS) y un marcador selectivo (normalmente resistencia a antibióticos ). Además, aguas arriba del MCS se encuentran las regiones promotoras y el sitio de inicio de la transcripción , que regulan la expresión del gen clonado.
Este plásmido se puede insertar en células bacterianas o animales. La introducción de ADN en células bacterianas se puede realizar mediante transformación mediante la absorción de ADN desnudo, conjugación mediante contacto célula-célula o mediante transducción mediante un vector viral. La introducción de ADN en células eucariotas , como las células animales, por medios físicos o químicos se denomina transfección . Se encuentran disponibles varias técnicas de transfección diferentes, como la transfección con fosfato cálcico, la electroporación , la microinyección y la transfección de liposomas . El plásmido puede integrarse en el genoma , lo que da como resultado una transfección estable, o puede permanecer independiente del genoma y expresarse temporalmente, lo que se denomina transfección transitoria. [30] [31]
El ADN que codifica una proteína de interés ahora se encuentra dentro de una célula y la proteína ahora puede expresarse. Se encuentran disponibles una variedad de sistemas, como promotores inducibles y factores de señalización celular específicos, para ayudar a expresar la proteína de interés en niveles altos. Luego se pueden extraer grandes cantidades de una proteína de la célula bacteriana o eucariota. Se puede probar la actividad enzimática de la proteína en diversas situaciones, se puede cristalizar la proteína para poder estudiar su estructura terciaria o, en la industria farmacéutica, se puede estudiar la actividad de nuevos fármacos contra la proteína. [32]
La reacción en cadena de la polimerasa (PCR) es una técnica extremadamente versátil para copiar ADN. En resumen, la PCR permite copiar o modificar una secuencia de ADN específica de formas predeterminadas. La reacción es extremadamente potente y, en condiciones perfectas, podría amplificar una molécula de ADN hasta convertirla en 1.070 millones de moléculas en menos de dos horas. La PCR tiene muchas aplicaciones, incluido el estudio de la expresión genética, la detección de microorganismos patógenos, la detección de mutaciones genéticas y la introducción de mutaciones en el ADN. [33] La técnica de PCR se puede utilizar para introducir sitios de enzimas de restricción en los extremos de las moléculas de ADN, o para mutar bases particulares del ADN; este último es un método denominado mutagénesis dirigida al sitio . La PCR también se puede utilizar para determinar si un fragmento de ADN particular se encuentra en una biblioteca de ADNc . La PCR tiene muchas variaciones, como la PCR con transcripción inversa ( RT-PCR ) para la amplificación de ARN y, más recientemente, la PCR cuantitativa que permite la medición cuantitativa de moléculas de ADN o ARN. [34] [35]
.jpg/440px-Two_percent_Agarose_Gel_in_Borate_Buffer_cast_in_a_Gel_Tray_(Front,_angled).jpg)

La electroforesis en gel es una técnica que separa moléculas por su tamaño mediante un gel de agarosa o poliacrilamida. [36] Esta técnica es una de las principales herramientas de la biología molecular. El principio básico es que los fragmentos de ADN se pueden separar aplicando una corriente eléctrica a través del gel; debido a que la columna vertebral del ADN contiene grupos fosfato cargados negativamente, el ADN migrará a través del gel de agarosa hacia el extremo positivo de la corriente. [36] Las proteínas también se pueden separar según su tamaño utilizando un gel SDS-PAGE , o según su tamaño y su carga eléctrica mediante lo que se conoce como electroforesis en gel 2D . [37]
El ensayo de Bradford es una técnica de biología molecular que permite la cuantificación rápida y precisa de moléculas de proteínas utilizando las propiedades únicas de un tinte llamado Coomassie Brilliant Blue G-250. [38] El azul Coomassie experimenta un cambio de color visible de marrón rojizo a azul brillante al unirse a la proteína. [38] En su estado catiónico inestable, el azul de Coomassie tiene una longitud de onda de fondo de 465 nm y emite un color marrón rojizo. [39] Cuando el azul de Coomassie se une a la proteína en una solución ácida, la longitud de onda de fondo cambia a 595 nm y el tinte emite un color azul brillante. [39] Las proteínas en el ensayo se unen al azul de Coomassie en aproximadamente 2 minutos, y el complejo proteína-tinte es estable durante aproximadamente una hora, aunque se recomienda que las lecturas de absorbancia se tomen dentro de los 5 a 20 minutos posteriores al inicio de la reacción. [38] La concentración de proteína en el ensayo de Bradford se puede medir usando un espectrofotómetro de luz visible y, por lo tanto, no requiere equipo extenso. [39]
Este método fue desarrollado en 1975 por Marion M. Bradford y ha permitido una cuantificación de proteínas significativamente más rápida y precisa en comparación con métodos anteriores: el procedimiento de Lowry y el ensayo de biuret. [38] A diferencia de los métodos anteriores, el ensayo de Bradford no es susceptible a la interferencia de varias moléculas no proteicas, incluidos el etanol, el cloruro de sodio y el cloruro de magnesio. [38] Sin embargo, es susceptible a la influencia de agentes tampón alcalinos fuertes, como el dodecilsulfato de sodio (SDS). [38]
Los términos transferencia Northern , Western y Eastern se derivan de lo que inicialmente fue una broma de biología molecular que jugaba con el término transferencia Southern , en honor a la técnica descrita por Edwin Southern para la hibridación de ADN transferido. Patricia Thomas, desarrolladora de la transferencia de ARN que luego se conoció como transferencia Northern , en realidad no utilizó el término. [40]
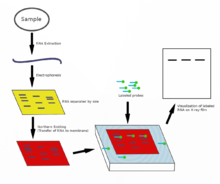
La transferencia Southern , que lleva el nombre de su inventor, el biólogo Edwin Southern , es un método para detectar la presencia de una secuencia de ADN específica dentro de una muestra de ADN. Las muestras de ADN antes o después de la digestión con enzimas de restricción (endonucleasa de restricción) se separan mediante electroforesis en gel y luego se transfieren a una membrana mediante transferencia mediante acción capilar . Luego, la membrana se expone a una sonda de ADN marcada que tiene una secuencia de bases complementaria a la secuencia del ADN de interés. [41] La transferencia Southern se utiliza con menos frecuencia en la ciencia de laboratorio debido a la capacidad de otras técnicas, como la PCR , para detectar secuencias de ADN específicas a partir de muestras de ADN. Sin embargo, estas transferencias todavía se utilizan para algunas aplicaciones, como medir el número de copias de transgenes en ratones transgénicos o en la ingeniería de líneas de células madre embrionarias con genes desactivados . [27]
La transferencia Northern se utiliza para estudiar la presencia de moléculas de ARN específicas como comparación relativa entre un conjunto de diferentes muestras de ARN. Es esencialmente una combinación de electroforesis en gel de ARN desnaturalizante y una transferencia . En este proceso, el ARN se separa según el tamaño y luego se transfiere a una membrana que luego se sonda con un complemento marcado de una secuencia de interés. Los resultados se pueden visualizar de diversas formas según la etiqueta utilizada; sin embargo, la mayoría da como resultado la revelación de bandas que representan los tamaños del ARN detectado en la muestra. La intensidad de estas bandas está relacionada con la cantidad de ARN diana en las muestras analizadas. El procedimiento se usa comúnmente para estudiar cuándo y cuánta expresión génica está ocurriendo midiendo cuánto de ese ARN está presente en diferentes muestras, asumiendo que no ocurre regulación postranscripcional y que los niveles de ARNm reflejan niveles proporcionales de la proteína correspondiente que se está procesando. producido. Es una de las herramientas más básicas para determinar en qué momento y bajo qué condiciones ciertos genes se expresan en los tejidos vivos. [42] [43]
Una transferencia Western es una técnica mediante la cual se pueden detectar proteínas específicas a partir de una mezcla de proteínas. [44] Las transferencias Western se pueden utilizar para determinar el tamaño de las proteínas aisladas, así como para cuantificar su expresión. [45] En la transferencia Western , las proteínas se separan primero por tamaño, en un gel fino intercalado entre dos placas de vidrio en una técnica conocida como SDS-PAGE . Luego, las proteínas del gel se transfieren a una membrana de soporte de fluoruro de polivinilideno (PVDF), nitrocelulosa, nailon u otra membrana. Esta membrana puede luego ser sondada con soluciones de anticuerpos . Los anticuerpos que se unen específicamente a la proteína de interés pueden visualizarse mediante una variedad de técnicas, incluidos productos coloreados, quimioluminiscencia o autorradiografía . A menudo, los anticuerpos están marcados con enzimas. Cuando un sustrato quimioluminiscente se expone a la enzima , permite la detección. El uso de técnicas de transferencia Western permite no solo la detección sino también el análisis cuantitativo. Se pueden utilizar métodos análogos a la transferencia Western para teñir directamente proteínas específicas en células vivas o secciones de tejido . [44] [46]
La técnica de transferencia Eastern se utiliza para detectar modificaciones postraduccionales de proteínas. Las proteínas transferidas a la membrana de PVDF o nitrocelulosa se prueban para detectar modificaciones utilizando sustratos específicos. [47]

Una micromatriz de ADN es una colección de puntos adheridos a un soporte sólido, como un portaobjetos de microscopio , donde cada punto contiene uno o más fragmentos de oligonucleótidos de ADN monocatenario . Las matrices permiten colocar grandes cantidades de puntos muy pequeños (100 micrómetros de diámetro) en un solo portaobjetos. Cada mancha tiene una molécula de fragmento de ADN que es complementaria a una única secuencia de ADN . Una variación de esta técnica permite calificar la expresión genética de un organismo en una etapa particular del desarrollo ( perfil de expresión ). En esta técnica, el ARN de un tejido se aísla y se convierte en ADN complementario marcado (ADNc). Luego, este ADNc se hibrida con los fragmentos de la matriz y se puede realizar la visualización de la hibridación. Dado que se pueden crear múltiples matrices con exactamente la misma posición de fragmentos, son particularmente útiles para comparar la expresión genética de dos tejidos diferentes, como un tejido sano y uno canceroso. Además, se pueden medir qué genes se expresan y cómo esa expresión cambia con el tiempo o con otros factores. Hay muchas formas diferentes de fabricar microarrays; los más comunes son los chips de silicio, portaobjetos de microscopio con manchas de ~100 micrómetros de diámetro, matrices personalizadas y matrices con manchas más grandes en membranas porosas (macromatrices). Puede haber desde 100 puntos hasta más de 10.000 en una matriz determinada. También se pueden crear matrices con moléculas distintas del ADN. [48] [49] [50] [51]
El oligonucleótido alelo específico (ASO) es una técnica que permite la detección de mutaciones de una sola base sin necesidad de PCR o electroforesis en gel. Las sondas marcadas cortas (de 20 a 25 nucleótidos de longitud) están expuestas al ADN objetivo no fragmentado, la hibridación se produce con alta especificidad debido a la corta longitud de las sondas e incluso un solo cambio de base dificultará la hibridación. Luego se lava el ADN diana y se eliminan las sondas marcadas que no se hibridaron. Luego se analiza el ADN objetivo para detectar la presencia de la sonda mediante radioactividad o fluorescencia. En este experimento, como en la mayoría de las técnicas de biología molecular, se debe utilizar un control para garantizar el éxito de la experimentación. [52] [53]
En biología molecular, se desarrollan continuamente procedimientos y tecnologías y se abandonan las tecnologías más antiguas. Por ejemplo, antes de la llegada de la electroforesis en gel de ADN ( agarosa o poliacrilamida ), el tamaño de las moléculas de ADN se determinaba típicamente mediante velocidad de sedimentación en gradientes de sacarosa , una técnica lenta y laboriosa que requería instrumentación costosa; Antes de los gradientes de sacarosa, se utilizó viscometría . Aparte de su interés histórico, a menudo vale la pena conocer la tecnología más antigua, ya que en ocasiones es útil para resolver otro problema nuevo para el cual la técnica más nueva no es apropiada. [54]