En biología y genealogía genética , el ancestro común más reciente ( MRCA ), también conocido como último ancestro común ( LCA ), de un conjunto de organismos es el individuo más reciente del que descienden todos los organismos del conjunto . El término también se utiliza en referencia a la ascendencia de grupos de genes ( haplotipos ) en lugar de organismos.
El MRCA de un conjunto de individuos a veces puede determinarse haciendo referencia a un pedigrí establecido . Sin embargo, en general, es imposible identificar el MRCA exacto de un gran conjunto de individuos, pero a menudo se puede dar una estimación del tiempo en el que vivió el MRCA. Las estimaciones de tiempo hasta el ancestro común más reciente ( TMRCA ) se pueden dar en función de los resultados de las pruebas de ADN y las tasas de mutación establecidas como se practica en la genealogía genética, o mediante referencia a un modelo matemático no genético o una simulación por computadora.
En los organismos que utilizan la reproducción sexual , el MRCA matrilineal y el MRCA patrilineal son los MRCA de una población determinada considerando solo la descendencia matrilineal y patrilineal , respectivamente. El MRCA de una población, por definición, no puede ser más antiguo que su MRCA matrilineal o patrilineal. En el caso del Homo sapiens , el MRCA matrilineal y patrilineal también se conocen como " Eva mitocondrial " (mt-MRCA) y " Adán cromosómico Y " (Y-MRCA) respectivamente.
Se desconoce la edad del MRCA humano. No es mayor que la edad del Y-MRCA o del mt-MRCA, estimada en unos 200.000 años.
A diferencia de los pedigríes de humanos individuales o linajes domesticados donde se conoce el parentesco histórico, en la inferencia de relaciones entre especies o grupos superiores de taxones ( sistemática o filogenética ), los antepasados no son directamente observables ni reconocibles. Son inferencias basadas en patrones de relación entre taxones inferidos en un análisis filogenético de organismos y/o fósiles existentes . [1]
El último ancestro común universal (LUCA) es el ancestro común más reciente de toda la vida actual en la Tierra, y se estima que vivió hace entre 3.500 y 3.800 millones de años (en el Paleoarqueano ). [2] [3] [nota 1]
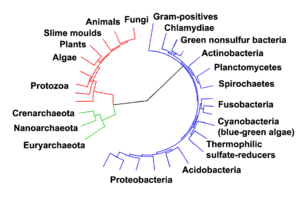
El proyecto de descripción completa de las relaciones filogenéticas entre todas las especies biológicas se denomina " árbol de la vida ". Esto implica la inferencia de edades de divergencia para todos los clados hipotéticos ; por ejemplo, se estima que el MRCA de todos los carnívoros (es decir, el MRCA de " perros y gatos ") divergió hace unos 42 millones de años ( Miacidae ). [6]
El concepto del último ancestro común desde la perspectiva de la evolución humana se describe para una audiencia popular en The Ancestor's Tale de Richard Dawkins (2004). Dawkins enumera los "concesores" del linaje humano en orden creciente de edad, incluidos los homínidos (humano- chimpancé ), los homínidos (humanos- gorila ), los homínidos (humanos- orangutanes ), los hominoideos (humanos- gibones ), etc., en 40 etapas. en total, hasta el último ancestro común universal (humano- bacteria ).
También es posible considerar la ascendencia de genes individuales (o grupos de genes, haplotipos ) en lugar de un organismo en su conjunto. La teoría coalescente describe un modelo estocástico de cómo la ascendencia de tales marcadores genéticos se relaciona con la historia de una población.
A diferencia de los organismos, un gen se transmite de una generación de organismos a la siguiente, ya sea como réplicas perfectas de sí mismo o como genes descendientes ligeramente mutados . Mientras que los organismos tienen gráficos de ascendencia y gráficos de progenie mediante reproducción sexual , un gen tiene una única cadena de ancestros y un árbol de descendientes. Un organismo producido por fertilización cruzada sexual ( alogamia ) tiene al menos dos ancestros (sus padres inmediatos), pero un gen siempre tiene un ancestro por generación.

El ADN mitocondrial (ADNmt) es casi inmune a la mezcla sexual, a diferencia del ADN nuclear cuyos cromosomas se mezclan y recombinan en la herencia mendeliana . Por lo tanto, el ADN mitocondrial se puede utilizar para rastrear la herencia matrilineal y encontrar la Eva mitocondrial (también conocida como Eva africana ), el ancestro común más reciente de todos los humanos a través de la vía del ADN mitocondrial.
Asimismo, el cromosoma Y está presente como un cromosoma sexual único en el individuo masculino y se transmite a los descendientes masculinos sin recombinación. Puede utilizarse para rastrear la herencia patrilineal y encontrar el Adán cromosómico Y , el ancestro común más reciente de todos los humanos a través de la vía del ADN-Y.
Los investigadores han establecido fechas aproximadas para la Eva mitocondrial y el Adán cromosómico Y mediante pruebas de ADN genealógico . Se estima que la Eva mitocondrial vivió hace unos 200.000 años. Un artículo publicado en marzo de 2013 determinó que, con un 95% de confianza y siempre que no haya errores sistemáticos en los datos del estudio, el Adán cromosómico Y vivió hace entre 237.000 y 581.000 años. [7] [8]
Por lo tanto, el MRCA de todos los humanos vivos hoy tendría que haber vivido más recientemente que cualquiera de los dos. [9] [nota 2]
Es más complicado inferir la ascendencia humana a través de cromosomas autosómicos . Aunque un cromosoma autosómico contiene genes que se transmiten de padres a hijos a través de una selección independiente de solo uno de los dos padres, la recombinación genética ( cruce cromosómico ) mezcla genes de cromátidas no hermanas de ambos padres durante la meiosis , cambiando así la composición genética de el cromosoma.
Se estima que diferentes tipos de MRCA vivieron en diferentes momentos del pasado. Estas estimaciones de tiempo hasta MRCA ( TMRCA ) también se calculan de manera diferente según el tipo de MRCA que se considere. Los MRCA patrilineales y matrilineales (Eva mitocondrial y Adán cromosómico Y) se rastrean mediante marcadores de un solo gen, por lo que sus TMRCA se calculan en función de los resultados de las pruebas de ADN y las tasas de mutación establecidas como se practica en la genealogía genética. El tiempo hasta el MRCA genealógico (el ancestro común más reciente por cualquier línea de descendencia) de todos los seres humanos vivos no se puede rastrear genéticamente porque el ADN de la gran mayoría de los ancestros se pierde por completo después de unos cientos de años. Por lo tanto, se calcula basándose en modelos matemáticos no genéticos y simulaciones por computadora.
Dado que la Eva mitocondrial y el Adán cromosómico Y son rastreados por genes únicos a través de una única línea parental ancestral, el tiempo hasta estos MRCA genéticos será necesariamente mayor que el del MRCA genealógico. Esto se debe a que los genes individuales se unirán más lentamente que el rastreo de la genealogía humana convencional a través de ambos padres. Este último considera sólo a los seres humanos individualmente, sin tener en cuenta si algún gen del MRCA calculado realmente sobrevive en cada persona de la población actual. [11]
El ADN mitocondrial se puede utilizar para rastrear la ascendencia de un conjunto de poblaciones. En este caso, las poblaciones se definen por la acumulación de mutaciones en el ADNmt y se crean árboles especiales para las mutaciones y el orden en que ocurrieron en cada población. El árbol se forma mediante la prueba de un gran número de individuos en todo el mundo para detectar la presencia o ausencia de un determinado conjunto de mutaciones. Una vez hecho esto es posible determinar cuántas mutaciones separan a una población de otra. El número de mutaciones, junto con la tasa de mutación estimada del ADNmt en las regiones analizadas, permite a los científicos determinar el tiempo aproximado hasta MRCA ( TMRCA ), que indica el tiempo transcurrido desde que las poblaciones compartieron por última vez el mismo conjunto de mutaciones o pertenecieron al mismo haplogrupo. .
En el caso del ADN del cromosoma Y, se llega a TMRCA de una manera diferente. Los haplogrupos de ADN-Y se definen mediante polimorfismo de un solo nucleótido en varias regiones del ADN-Y. El tiempo hasta MRCA dentro de un haplogrupo se define por la acumulación de mutaciones en secuencias STR del cromosoma Y de ese haplogrupo únicamente. El análisis de la red de ADN-Y de los haplotipos Y-STR que muestran un cúmulo sin estrellas indica la variabilidad de Y-STR debido a múltiples individuos fundadores. Se puede considerar que el análisis que arroja un cúmulo de estrellas representa una población descendiente de un solo ancestro. En este caso, la variabilidad de la secuencia Y-STR , también llamada variación de microsatélites , puede considerarse como una medida del tiempo transcurrido desde que el antepasado fundó esta población en particular. Los descendientes de Genghis Khan o uno de sus antepasados representan un famoso cúmulo de estrellas que se remonta a la época de Genghis Khan. [12]
Los cálculos de TMRCA se consideran evidencia crítica cuando se intenta determinar las fechas de migración de varias poblaciones a medida que se expanden por todo el mundo. Por ejemplo, si se considera que una mutación ocurrió hace 30.000 años, entonces esta mutación debería encontrarse entre todas las poblaciones que divergieron después de esta fecha. Si la evidencia arqueológica indica una expansión cultural y la formación de poblaciones regionalmente aisladas, entonces esto debe reflejarse en el aislamiento de mutaciones genéticas posteriores en esta región. Si la divergencia genética y la divergencia regional coinciden, se puede concluir que la divergencia observada se debe a la migración, como lo demuestra el registro arqueológico. Sin embargo, si la fecha de la divergencia genética ocurre en un momento diferente al del registro arqueológico, entonces los científicos tendrán que buscar evidencia arqueológica alternativa para explicar la divergencia genética. La cuestión se ilustra mejor en el debate en torno a la difusión démica versus la difusión cultural durante el Neolítico europeo . [13]
Se desconoce la edad del MRCA de todos los seres humanos vivos . Es necesariamente más joven que la edad del MRCA matrilineal o patrilineal, los cuales tienen una edad estimada de hace aproximadamente 100.000 y 200.000 años. [14]
Un estudio realizado por los matemáticos Joseph T. Chang, Douglas Rohde y Steve Olson utilizó un modelo teórico para calcular que el MRCA pudo haber vivido muy recientemente, posiblemente hace tan sólo 2.000 años. Concluye que el MRCA de todos los humanos probablemente vivía en el este de Asia, lo que les habría dado acceso clave a poblaciones extremadamente aisladas en Australia y América. Las posibles ubicaciones para el MRCA incluyen lugares como las penínsulas de Chuckchi y Kamchatka que están cerca de Alaska, lugares como Indonesia y Malasia que están cerca de Australia o un lugar como Taiwán o Japón que es más intermedio con Australia y las Américas. Chang consideró que la colonización europea de América y Australia era demasiado reciente para haber tenido un impacto sustancial en la antigüedad de la MRCA. De hecho, si los europeos nunca hubieran descubierto América y Australia, el MRCA sólo estaría aproximadamente un 2,3% más atrás en el pasado de lo que está. [15] [16] [17]
Tenga en cuenta que la edad del MRCA de una población no corresponde a un cuello de botella poblacional , y mucho menos a una "primera pareja". Más bien refleja la presencia de un solo individuo con alto éxito reproductivo en el pasado, cuya contribución genética se ha vuelto omnipresente en toda la población con el tiempo. También es incorrecto suponer que la MRCA transmitió toda la información genética, o incluso parte de ella, a cada persona viva. A través de la reproducción sexual , un antepasado transmite la mitad de sus genes a cada descendiente de la siguiente generación; en ausencia de un colapso del pedigrí , después de sólo 32 generaciones la contribución de un solo ancestro sería del orden de 2 −32 , un número proporcional a menos de un único par de bases dentro del genoma humano . [18]
El MRCA es el ancestro común más reciente compartido por todos los individuos de la población considerada. Esta MRCA bien puede tener contemporáneos que también sean ancestrales de parte, pero no de toda, la población existente. El punto de ancestros idénticos es un punto en el pasado más remoto que el MRCA en el que ya no hay organismos que sean ancestrales de parte, pero no de toda, la población moderna. Debido al colapso del pedigrí , los individuos modernos aún pueden exhibir agrupaciones, debido a contribuciones muy diferentes de cada población ancestral. [19]
{{cite journal}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace ){{cite web}}: Mantenimiento CS1: copia archivada como título ( enlace ){{cite journal}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace ){{cite web}}: Mantenimiento CS1: copia archivada como título ( enlace )La paradoja del Barco de Teseo […] se invoca con frecuencia para ilustrar este punto […].
Incluso niveles moderados de transferencia de genes harán imposible reconstruir los genomas de los primeros ancestros;
…
{{cite journal}}: Mantenimiento CS1: varios nombres: lista de autores ( enlace )