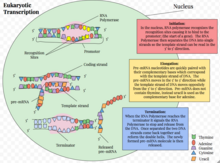
La transcripción eucariota es el proceso elaborado que utilizan las células eucariotas para copiar la información genética almacenada en el ADN en unidades de réplica de ARN complementario transportable. [1] La transcripción genética ocurre tanto en células eucariotas como procariotas . A diferencia de la ARN polimerasa procariota que inicia la transcripción de todos los diferentes tipos de ARN, la ARN polimerasa en eucariotas (incluidos los humanos) viene en tres variaciones, cada una de las cuales traduce un tipo diferente de gen. Una célula eucariota tiene un núcleo que separa los procesos de transcripción y traducción . La transcripción eucariota ocurre dentro del núcleo donde el ADN se empaqueta en nucleosomas y estructuras de cromatina de orden superior . La complejidad del genoma eucariota requiere una gran variedad y complejidad de control de la expresión genética.
La transcripción eucariota se desarrolla en tres etapas secuenciales: iniciación, elongación y terminación. [1]
Los ARN transcritos cumplen diversas funciones. Por ejemplo, los componentes estructurales del ribosoma son transcritos por la ARN polimerasa I. Los genes codificadores de proteínas son transcritos por la ARN polimerasa II en ARN mensajeros (ARNm) que llevan la información desde el ADN hasta el sitio de síntesis de proteínas. [1] Los ARN no codificantes, que se producen en mayor abundancia, representan la gran mayoría de la producción transcripcional de una célula. [2] Estos ARN no codificantes realizan una variedad de funciones celulares importantes. [2]
Los eucariotas tienen tres ARN polimerasas nucleares, cada una con funciones y propiedades distintas. [3] [4]
La ARN polimerasa I (Pol I) cataliza la transcripción de todos los genes de ARNr excepto el 5S. [3] [4] Estos genes de ARNr están organizados en una sola unidad transcripcional y se transcriben en una transcripción continua. Este precursor luego se procesa en tres ARNr: 18S, 5.8S y 28S. La transcripción de los genes de ARNr tiene lugar en una estructura especializada del núcleo llamada nucléolo, [5] donde los ARNr transcritos se combinan con proteínas para formar ribosomas . [6]
La ARN polimerasa II (Pol II) es responsable de la transcripción de todos los ARNm, algunos ARNpn, ARNi y todos los miARN. [3] [4] Muchas transcripciones de Pol II existen transitoriamente como ARN precursores de cadena sencilla (pre-ARN) que se procesan posteriormente para generar ARN maduros. [1] Por ejemplo, los ARNm precursores (pre-ARNm) se procesan ampliamente antes de salir al citoplasma a través del poro nuclear para la traducción de proteínas.
La ARN polimerasa III (Pol III) transcribe ARN pequeños no codificantes, incluidos ARNt, ARNr 5S, ARNsn U6, ARN SRP y otros ARN cortos estables como el ARN ribonucleasa P. [7]

Las ARN polimerasas I, II y III contienen 14, 12 y 17 subunidades, respectivamente. [8] Las tres polimerasas eucariotas tienen cinco subunidades centrales que exhiben homología con las subunidades β, β', α I , α II y ω de la ARN polimerasa de E. coli. Las tres polimerasas eucariotas utilizan una subunidad idéntica similar a ω (RBP6), mientras que las Pol I y III utilizan las mismas subunidades similares a α. Las tres polimerasas eucariotas comparten otras cuatro subunidades comunes entre sí. Las subunidades restantes son exclusivas de cada ARN polimerasa. Las subunidades adicionales que se encuentran en Pol I y Pol III en relación con Pol II son homólogas a los factores de transcripción de Pol II. [8]
Las estructuras cristalinas de las ARN polimerasas I [9] y II [10] brindan una oportunidad para comprender las interacciones entre las subunidades y el mecanismo molecular de la transcripción eucariota en detalle atómico.
El dominio carboxilo terminal (CTD) de RPB1 , la subunidad más grande de la ARN polimerasa II, desempeña un papel importante en la unión de la maquinaria necesaria para la síntesis y el procesamiento de las transcripciones de Pol II. [11] Largo y estructuralmente desordenado, el CTD contiene múltiples repeticiones de la secuencia heptapeptídica YSPTSPS que están sujetas a fosforilación y otras modificaciones postraduccionales durante el ciclo de transcripción. Estas modificaciones y su regulación constituyen el código operativo para que el CTD controle la iniciación, elongación y terminación de la transcripción y acople la transcripción y el procesamiento del ARN. [11]
La iniciación de la transcripción génica en eucariotas ocurre en pasos específicos. [1] Primero, una ARN polimerasa junto con factores de transcripción generales se une a la región promotora del gen para formar un complejo cerrado llamado complejo de preiniciación . La transición posterior del complejo del estado cerrado al estado abierto da como resultado la fusión o separación de las dos cadenas de ADN y el posicionamiento de la cadena molde en el sitio activo de la ARN polimerasa. Sin la necesidad de un cebador, la ARN polimerasa puede iniciar la síntesis de una nueva cadena de ARN utilizando la cadena de ADN molde para guiar la selección de ribonucleótidos y la química de polimerización. [1] Sin embargo, muchas de las síntesis iniciadas se abortan antes de que las transcripciones alcancen una longitud significativa (~10 nucleótidos). Durante estos ciclos abortivos, la polimerasa sigue produciendo y liberando transcripciones cortas hasta que es capaz de producir una transcripción que supera los diez nucleótidos de longitud. Una vez que se alcanza este umbral, la ARN polimerasa pasa el promotor y la transcripción procede a la fase de elongación. [1]
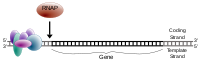
Los genes transcritos por Pol II contienen una región en las inmediaciones del sitio de inicio de la transcripción (TSS) que se une y posiciona el complejo de preiniciación. Esta región se denomina promotor central debido a su papel esencial en la iniciación de la transcripción. [12] [13] En los promotores se encuentran diferentes clases de elementos de secuencia. Por ejemplo, la caja TATA es la secuencia de reconocimiento de ADN altamente conservada para la proteína de unión a la caja TATA, TBP , cuya unión inicia el ensamblaje del complejo de transcripción en muchos genes.
Los genes eucariotas también contienen secuencias reguladoras más allá del promotor central. Estos elementos de control que actúan en cis se unen a activadores o represores transcripcionales para aumentar o disminuir la transcripción a partir del promotor central. Los elementos reguladores bien caracterizados incluyen potenciadores , silenciadores y aisladores . Estas secuencias reguladoras pueden estar distribuidas a lo largo de una gran distancia genómica, a veces ubicadas a cientos de kilobases de los promotores centrales. [1]
Los factores de transcripción generales son un grupo de proteínas implicadas en la iniciación y regulación de la transcripción. [1] Estos factores suelen tener dominios de unión al ADN que unen elementos de secuencia específicos del promotor central y ayudan a reclutar la ARN polimerasa al sitio de inicio de la transcripción. Los factores de transcripción generales para la ARN polimerasa II incluyen TFIID , TFIIA , TFIIB , TFIIF , TFIIE y TFIIH . [1] [14] [15]
La transcripción, un conjunto completo de factores de transcripción generales y la ARN polimerasa deben ensamblarse en el promotor central para formar el complejo de preiniciación de ~2,5 millones de Dalton. [16] Por ejemplo, para los promotores que contienen una caja TATA cerca del TSS, el reconocimiento de la caja TATA por la subunidad TBP de TFIID inicia el ensamblaje de un complejo de transcripción. Las siguientes proteínas en ingresar son TFIIA y TFIIB, que estabilizan el complejo ADN-TFIID y reclutan Pol II en asociación con TFIIF y factores de transcripción adicionales. TFIIB sirve como puente entre el TBP unido a TATA y la ARN polimerasa. También ayuda a colocar el centro activo de la polimerasa en la posición correcta para iniciar la transcripción. Uno de los últimos factores de transcripción en ser reclutados al complejo de preiniciación es TFIIH, que desempeña un papel importante en la fusión y escape del promotor. [17]

Para los genes transcritos por pol II, y a diferencia de la ARN polimerasa bacteriana, la fusión del promotor requiere hidrólisis de ATP y está mediada por TFIIH. [17] TFIIH es una proteína de diez subunidades, que incluye actividades tanto de ATPasa como de proteína quinasa . [18] Mientras que el ADN promotor corriente arriba se mantiene en una posición fija por TFIID, TFIIH tira del ADN bicatenario corriente abajo hacia la hendidura de la polimerasa, impulsando la separación de las cadenas de ADN y la transición del complejo de preiniciación del estado cerrado al abierto. TFIIB ayuda en la formación del complejo abierto uniendo el ADN fundido y estabilizando la burbuja de transcripción .
Una vez que el complejo de iniciación está abierto, el primer ribonucleótido se lleva al sitio activo para iniciar la reacción de polimerización en ausencia de un cebador. [1] Esto genera una cadena de ARN naciente que forma un heterodúplex con la cadena de ADN molde. Sin embargo, antes de entrar en la fase de elongación, la polimerasa puede terminar prematuramente y liberar una transcripción corta y truncada. Este proceso se llama iniciación abortiva. [19] Pueden ocurrir muchos ciclos de iniciación abortiva antes de que la transcripción crezca hasta una longitud suficiente para promover el escape de la polimerasa del promotor. A lo largo de los ciclos de iniciación abortiva, la ARN polimerasa permanece unida al promotor y tira del ADN corriente abajo hacia su hendidura catalítica en un movimiento de tipo aplastamiento. [19]
Cuando un transcrito alcanza la longitud límite de diez nucleótidos, entra en el canal de salida del ARN. [1] La polimerasa rompe sus interacciones con los elementos promotores y cualquier proteína reguladora asociada con el complejo de iniciación que ya no necesita. [20] El escape del promotor en eucariotas requiere hidrólisis de ATP y, en el caso de la fosforilación de Pol II del CTD. Mientras tanto, la burbuja de transcripción colapsa hasta los 12-14 nucleótidos, lo que proporciona la energía cinética necesaria para el escape. [1]
Después de escapar del promotor y desprenderse de la mayoría de los factores de transcripción para la iniciación, la polimerasa adquiere nuevos factores para la siguiente fase de la transcripción: elongación. [21] [22] La elongación de la transcripción es un proceso procesivo . El ADN bicatenario que entra desde el frente de la enzima se descomprime para aprovechar la cadena molde para la síntesis de ARN. Por cada par de bases de ADN separado por la polimerasa que avanza, se forma inmediatamente un par de bases híbrido ARN:ADN. Las cadenas de ADN y la cadena de ARN naciente salen de canales separados; las dos cadenas de ADN se reúnen en el extremo final de la burbuja de transcripción mientras que el ARN monocatenario emerge solo.
Entre las proteínas reclutadas por la polimerasa se encuentran los factores de elongación, llamados así porque estimulan la elongación de la transcripción. [23] Existen diferentes clases de factores de elongación. Algunos factores pueden aumentar la tasa general de transcripción, algunos pueden ayudar a la polimerasa a través de sitios de pausa transitorios y algunos pueden ayudar a la polimerasa a transcribir a través de la cromatina. [24] Uno de los factores de elongación, P-TEFb , es particularmente importante. [25] P-TEFb fosforila el segundo residuo (Ser-2) de las repeticiones CTD (YSPTSPS) de la Pol II unida. P-TEFb también fosforila y activa SPT5 y TAT-SF1. SPT5 es un factor de transcripción universal que ayuda a reclutar la enzima de tapado 5' a Pol II con un CTD fosforilado en Ser-5. TAF-SF1 recluta componentes de la maquinaria de empalme de ARN al CTD fosforilado en Ser-2. P-TEFb también ayuda a suprimir la pausa transitoria de la polimerasa cuando encuentra ciertas secuencias inmediatamente después del inicio. [25]
La fidelidad de la transcripción se logra a través de múltiples mecanismos. Las ARN polimerasas seleccionan el sustrato correcto de trifosfato de nucleósido (NTP) para evitar errores de transcripción. Solo el NTP que se aparea correctamente con la base codificante en el ADN es admitido en el centro activo. [26] [27] La ARN polimerasa realiza dos funciones de lectura de pruebas conocidas para detectar y eliminar nucleótidos mal incorporados: edición pirofosforilítica y edición hidrolítica. [1] La primera elimina el ribonucleótido insertado incorrectamente mediante una simple inversión de la reacción de polimerización, mientras que la segunda implica el retroceso de la polimerasa y la escisión de un segmento del producto de ARN que contiene el error. El factor de elongación TFIIS ( InterPro : IPR006289 ; TCEA1 , TCEA2 , TCEA3) estimula una actividad ribonucleasa inherente en la polimerasa, lo que permite la eliminación de bases mal incorporadas a través de una degradación local limitada del ARN. [28] Nótese que todas las reacciones (síntesis de enlace fosfodiéster, pirofosforólisis, hidrólisis de enlace fosfodiéster) son realizadas por la ARN polimerasa utilizando un solo centro activo. [29]
La elongación de la transcripción no es un proceso suave a lo largo del ADN de doble cadena, ya que la ARN polimerasa sufre una pausa cotranscripcional extensa durante la elongación de la transcripción. [30] [31] En general, la ARN polimerasa II no transcribe a través de un gen a un ritmo constante. Más bien, se detiene periódicamente en ciertas secuencias, a veces durante largos períodos de tiempo antes de reanudar la transcripción. [32] Esta pausa es especialmente pronunciada en los nucleosomas y surge en parte cuando la polimerasa entra en un estado de retroceso transcripcionalmente incompetente. [30] La duración de estas pausas varía de segundos a minutos o más, y la salida de pausas de larga duración puede ser promovida por factores de elongación como TFIIS. [33]
Esta pausa también se utiliza a veces para la corrección de pruebas; aquí la polimerasa retrocede, borra parte del ARN que ya ha producido y vuelve a intentar la transcripción. [1] En casos extremos, por ejemplo, cuando la polimerasa encuentra un nucleótido dañado, se detiene por completo. Más a menudo, una polimerasa en elongación se detiene cerca del promotor. [32] La pausa proximal al promotor durante la elongación temprana es un mecanismo comúnmente utilizado para regular los genes que están listos para expresarse rápidamente o de manera coordinada. La pausa está mediada por un complejo llamado NELF (factor de elongación negativa) en colaboración con DSIF (factor inductor de sensibilidad a DRB que contiene SPT4/SPT5). [34] El bloqueo se libera una vez que la polimerasa recibe una señal de activación, como la fosforilación de Ser-2 de la cola de CTD por P-TEFb. Otros factores de elongación como ELL y TFIIS estimulan la tasa de elongación al limitar la duración de la pausa de la polimerasa. [1]
La elongación de la polimerasa está asociada con un conjunto de factores proteicos necesarios para varios tipos de procesamiento del ARN. [1] El ARNm se tapa tan pronto como emerge del canal de salida del ARN de la polimerasa. Después de la tapa, la desfosforilación de Ser-5 dentro de las repeticiones CTD puede ser responsable de la disociación de la maquinaria de tapado. La fosforilación adicional de Ser-2 provoca el reclutamiento de la maquinaria de empalme del ARN que cataliza la eliminación de intrones no codificantes para generar ARNm maduro. [1] El empalme alternativo expande los complementos proteicos en eucariotas. Al igual que con el tapado y empalme 5', la cola CTD está involucrada en el reclutamiento de enzimas responsables de la 3'-poliadenilación , el evento final de procesamiento del ARN que está acoplado con la terminación de la transcripción. [1]
La última etapa de la transcripción es la terminación, que conduce a la disociación de la transcripción completa y la liberación de la ARN polimerasa del ADN molde. El proceso difiere para cada una de las tres ARN polimerasas. [35] El mecanismo de terminación es el menos comprendido de las tres etapas de transcripción.
La terminación de la transcripción de genes pre-ARNr por la polimerasa Pol I se lleva a cabo mediante un sistema que necesita un factor de terminación de la transcripción específico. [3] El mecanismo utilizado tiene cierta semejanza con la terminación dependiente de rho en procariotas. [36] Las células eucariotas contienen cientos de repeticiones de ADN ribosómico, a veces distribuidas en múltiples cromosomas. La terminación de la transcripción se produce en la región espaciadora intergénica ribosómica que contiene varios sitios de terminación de la transcripción aguas arriba de un sitio de pausa de Pol I. A través de un mecanismo aún desconocido, el extremo 3' de la transcripción se escinde, generando una gran molécula de ARNr primaria que se procesa posteriormente en los ARNr maduros 18S, 5.8S y 28S.
Cuando la Pol II llega al final de un gen, dos complejos proteicos transportados por el CTD, CPSF (factor de especificidad de escisión y poliadenilación) y CSTF (factor de estimulación de escisión), reconocen la señal de poli-A en el ARN transcrito. [35] El CPSF y el CSTF unidos a poli-A reclutan otras proteínas para llevar a cabo la escisión del ARN y luego la poliadenilación. La poli-A polimerasa agrega aproximadamente 200 adeninas al extremo 3' escindido del ARN sin una plantilla. [35] La larga cola de poli-A es exclusiva de las transcripciones realizadas por la Pol II.
En el proceso de terminación de la transcripción por Pol I y Pol II, el complejo de elongación no se disuelve inmediatamente después de que el ARN se escinde. La polimerasa continúa moviéndose a lo largo de la plantilla, generando una segunda molécula de ARN asociada con el complejo de elongación. [1] Se han propuesto dos modelos para explicar cómo se logra finalmente la terminación. [35] El modelo alostérico establece que cuando la transcripción procede a través de la secuencia de terminación, causa el desmontaje de los factores de elongación y/o un ensamblaje de factores de terminación que causan cambios conformacionales del complejo de elongación. [36] [37] El modelo de torpedo sugiere que una exonucleasa 5' a 3' degrada el segundo ARN a medida que emerge del complejo de elongación. La polimerasa se libera a medida que la exonucleasa altamente procesiva la alcanza. Se propone que una visión emergente expresará una fusión de estos dos modelos. [37]
La ARN polimerasa III puede terminar la transcripción de manera eficiente sin la participación de factores adicionales. La señal de terminación de Pol III consiste en un tramo de timinas (en la cadena no molde) ubicada dentro de los 40 pb aguas abajo del extremo 3' de los ARN maduros. [35] La señal de terminación de poli-T detiene la transcripción de Pol III.
La regulación de la expresión génica en eucariotas se logra mediante la interacción de varios niveles de control que actúan tanto localmente para activar o desactivar genes individuales en respuesta a una necesidad celular específica como globalmente para mantener un patrón de expresión génica en toda la cromatina que da forma a la identidad celular. [1] [38] Debido a que el genoma eucariota está envuelto alrededor de histonas para formar nucleosomas y estructuras de cromatina de orden superior, los sustratos para la maquinaria transcripcional están en general parcialmente ocultos. [1] Sin proteínas reguladoras, muchos genes se expresan a un nivel bajo o no se expresan en absoluto. La transcripción requiere el desplazamiento de los nucleosomas posicionados para permitir que la maquinaria transcripcional obtenga acceso al ADN. [39]
Todos los pasos de la transcripción están sujetos a algún grado de regulación. [1] La iniciación de la transcripción en particular es el nivel primario en el que se regula la expresión génica. Dirigirse al paso inicial limitante de la velocidad es el más eficiente en términos de costos de energía para la célula. La iniciación de la transcripción está regulada por elementos que actúan en cis ( potenciadores , silenciadores , aisladores) dentro de las regiones reguladoras del ADN y factores que actúan en trans específicos de la secuencia que actúan como activadores o represores. [1] La transcripción génica también puede regularse después de la iniciación dirigiéndose al movimiento de la polimerasa elongante. [40]
El genoma eucariota está organizado en una estructura de cromatina compacta que permite solo el acceso regulado al ADN. La estructura de la cromatina puede ser globalmente "abierta" y más permisiva desde el punto de vista transcripcional, o globalmente "condensada" y transcripcionalmente inactiva. La primera ( eucromatina ) está ligeramente compacta y es rica en genes bajo transcripción activa. La segunda ( heterocromatina ) incluye regiones pobres en genes, como telómeros y centrómeros, pero también regiones con densidad genética normal pero silenciadas desde el punto de vista transcripcional. La transcripción puede silenciarse mediante modificación de histonas ( desacetilación y metilación ), interferencia de ARN y/o metilación de ADN . [41]
Los patrones de expresión genética que definen la identidad celular se heredan a través de la división celular. [1] Este proceso se denomina regulación epigenética . La metilación del ADN se hereda de manera confiable a través de la acción de las metilasas de mantenimiento que modifican la cadena de ADN naciente generada por la replicación. [1] En las células de mamíferos, la metilación del ADN es el marcador principal de las regiones silenciadas transcripcionalmente. Las proteínas especializadas pueden reconocer el marcador y reclutar histonas desacetilasas y metilasas para restablecer el silenciamiento. Las modificaciones de histonas del nucleosoma también podrían heredarse durante la división celular, sin embargo, no está claro si puede funcionar de forma independiente sin la dirección de la metilación del ADN. [1]
Las dos tareas principales de la iniciación de la transcripción son proporcionar a la ARN polimerasa un acceso al promotor y ensamblar factores de transcripción generales con la polimerasa en un complejo de iniciación de la transcripción. Se han identificado diversos mecanismos de iniciación de la transcripción anulando las señales inhibidoras en el promotor del gen. [1] Los genes eucariotas han adquirido extensas secuencias reguladoras que abarcan una gran cantidad de sitios de unión del regulador y se extienden en kilobases (a veces cientos de kilobases) desde el promotor, tanto aguas arriba como aguas abajo. [1] Los sitios de unión del regulador a menudo se agrupan en unidades llamadas potenciadores. Los potenciadores pueden facilitar la acción altamente cooperativa de varios factores de transcripción (que constituyen los potenciadores ). Los potenciadores remotos permiten la regulación de la transcripción a distancia. Los aisladores situados entre potenciadores y promotores ayudan a definir los genes que un potenciador puede o no influir.
Los activadores transcripcionales eucariotas tienen funciones de unión y activación del ADN independientes. [1] Al unirse a su elemento cis, un activador puede reclutar polimerasa directamente o reclutar otros factores necesarios para la maquinaria transcripcional. Un activador también puede reclutar modificadores de nucleosomas que alteran la cromatina en la proximidad del promotor y, por lo tanto, ayudan a la iniciación. Múltiples activadores pueden trabajar juntos, ya sea reclutando un componente común o dos componentes mutuamente dependientes de la maquinaria transcripcional, o ayudándose mutuamente a unirse a sus sitios de ADN. [1] Estas interacciones pueden sinergizar múltiples entradas de señalización y producir respuestas transcripcionales complejas para abordar las necesidades celulares.
Los represores de la transcripción eucariotas comparten algunos de los mecanismos utilizados por sus homólogos procariotas. Por ejemplo, al unirse a un sitio del ADN que se superpone con el sitio de unión de un activador, un represor puede inhibir la unión del activador. Pero con mayor frecuencia, los represores eucariotas inhiben la función de un activador enmascarando su dominio activador, impidiendo su localización nuclear, promoviendo su degradación o inactivándolo mediante modificaciones químicas. [1] Los represores pueden inhibir directamente la iniciación de la transcripción al unirse a un sitio aguas arriba de un promotor e interactuar con la maquinaria transcripcional. Los represores pueden reprimir indirectamente la transcripción al reclutar modificadores de histonas (desacetilasas y metilasas) o enzimas de remodelación de nucleosomas que afectan la accesibilidad del ADN. [1] La represión de las modificaciones de histonas y ADN también es la base del silenciamiento transcripcional que puede propagarse a lo largo de la cromatina y desactivar múltiples genes. [42]
La fase de elongación comienza una vez que se ha completado el ensamblaje del complejo de elongación y progresa hasta que se encuentra una secuencia de terminación. [1] El movimiento posterior a la iniciación de la ARN polimerasa es el objetivo de otra clase de mecanismos reguladores importantes. Por ejemplo, el activador transcripcional Tat afecta la elongación en lugar de la iniciación durante su regulación de la transcripción del VIH . [43] De hecho, muchos genes eucariotas se regulan mediante la liberación de un bloqueo a la elongación de la transcripción llamado pausa proximal al promotor. [44] La pausa puede influir en la estructura de la cromatina en los promotores para facilitar la actividad genética y conducir a respuestas transcripcionales rápidas o sincrónicas cuando las células se exponen a una señal de activación. [32] La pausa está asociada con la unión de dos factores de elongación negativos, DSIF (SPT4/SPT5) y NELF, al complejo de elongación. Otros factores también pueden influir en la estabilidad y la duración de la polimerasa pausada. [45] La liberación de la pausa se desencadena por el reclutamiento de la quinasa P-TEFb. [40]
La terminación de la transcripción también ha surgido como un área importante de la regulación transcripcional. La terminación está asociada con el reciclaje eficiente de la polimerasa. [46] Los factores asociados con la terminación de la transcripción también pueden mediar la formación de bucles genéticos y, por lo tanto, determinar la eficiencia de la reiniciación.
Cuando la transcripción se detiene por la presencia de una lesión en la cadena transcrita de un gen, las proteínas reparadoras del ADN se reclutan en la ARN polimerasa bloqueada para iniciar un proceso llamado reparación acoplada a la transcripción. [47] En el centro de este proceso se encuentra el factor de transcripción general TFIIH que tiene actividad ATPasa. TFIIH provoca un cambio conformacional en la polimerasa, para exponer la burbuja de transcripción atrapada en el interior, con el fin de que las enzimas reparadoras del ADN accedan a la lesión. [48] Por lo tanto, la ARN polimerasa actúa como proteína detectora de daños en la célula para dirigir las enzimas reparadoras a los genes que se están transcribiendo activamente.
La transcripción eucariota es más compleja que la transcripción procariota . Por ejemplo, en los eucariotas el material genético (ADN), y por lo tanto la transcripción, se localiza principalmente en el núcleo, donde está separado del citoplasma (en el que se produce la traducción) por la membrana nuclear. Esto permite la regulación temporal de la expresión génica a través del secuestro del ARN en el núcleo, y permite el transporte selectivo de ARN maduros al citoplasma. Las bacterias no tienen un núcleo diferenciado que separe el ADN del ribosoma y el ARNm se traduce en proteína tan pronto como se transcribe. El acoplamiento entre los dos procesos proporciona un mecanismo importante para la regulación génica procariota. [1]
En el nivel de iniciación, la ARN polimerasa en procariotas (bacterias en particular) se une fuertemente a la región promotora e inicia una alta tasa basal de transcripción. No se necesita hidrólisis de ATP para la transición de cerrado a abierto, la fusión del promotor es impulsada por reacciones de unión que favorecen la conformación fundida. La cromatina impide en gran medida la transcripción en eucariotas. Se requiere el ensamblaje de un gran complejo de preiniciación de múltiples proteínas para la iniciación específica del promotor. La fusión del promotor en eucariotas requiere hidrólisis de ATP. Como resultado, las ARN polimerasas eucariotas exhiben una baja tasa basal de iniciación de la transcripción. [42]
En los vertebrados, la mayoría de los promotores de genes contienen una isla CpG con numerosos sitios CpG . [49] Cuando muchos de los sitios CpG del promotor de un gen están metilados , el gen se silencia. [50] Los cánceres colorrectales suelen tener de 3 a 6 mutaciones impulsoras y de 33 a 66 mutaciones autoestopistas o pasajeras. [51] Sin embargo, el silenciamiento transcripcional puede ser de mayor importancia que la mutación a la hora de provocar la progresión al cáncer. Por ejemplo, en los cánceres colorrectales, entre 600 y 800 genes se silencian transcripcionalmente mediante la metilación de la isla CpG (véase regulación de la transcripción en el cáncer ). La represión transcripcional en el cáncer también puede producirse por otros mecanismos epigenéticos , como la expresión alterada de microARN . [52] En el cáncer de mama, la represión transcripcional de BRCA1 puede ocurrir con mayor frecuencia por la sobreexpresión del microARN-182 que por la hipermetilación del promotor BRCA1 (ver Baja expresión de BRCA1 en cánceres de mama y de ovario ).