Un alelo [1] , o alelomorfo , es una variante de la secuencia de nucleótidos en una ubicación particular, o locus , en una molécula de ADN . [2]
Los alelos pueden diferir en una sola posición a través de polimorfismos de un solo nucleótido (SNP), [3] pero también pueden tener inserciones y deleciones de hasta varios miles de pares de bases . [4]
La mayoría de los alelos observados producen poco o ningún cambio en la función del producto génico que codifican. Sin embargo, a veces diferentes alelos pueden dar lugar a diferentes rasgos fenotípicos observables , como una pigmentación diferente . Un ejemplo notable de esto es el descubrimiento de Gregor Mendel de que los colores blanco y morado de las flores de las plantas de guisantes eran el resultado de un único gen con dos alelos.
Casi todos los organismos multicelulares tienen dos juegos de cromosomas en algún momento de su ciclo de vida biológico ; es decir, son diploides . Para un locus determinado, si los dos cromosomas contienen el mismo alelo, ellos y el organismo son homocigotos con respecto a ese alelo. Si los alelos son diferentes, ellos y el organismo son heterocigotos con respecto a esos alelos.
Las definiciones populares de "alelo" suelen referirse únicamente a diferentes alelos dentro de los genes. Por ejemplo, la agrupación sanguínea ABO está controlada por el gen ABO , que tiene seis alelos comunes (variantes). En genética de poblaciones , el fenotipo del gen ABO de casi todos los seres humanos vivos es una combinación de solo estos seis alelos. [5] [6]
La palabra "alelo" es una forma corta de "alelomorfo" ("otra forma", una palabra acuñada por los genetistas británicos William Bateson y Edith Rebecca Saunders ) en la década de 1900, [7] [8] que se usó en los primeros días de la genética para describir formas variantes de un gen detectadas como fenotipos diferentes . Deriva del prefijo griego ἀλληλο-, allelo- , que significa "mutuo", "recíproco" o "uno al otro", que a su vez está relacionado con el adjetivo griego ἄλλος, allos (cognado del latín alius ), que significa "otro".
En muchos casos, las interacciones genotípicas entre los dos alelos en un locus pueden describirse como dominantes o recesivas , según a cuál de los dos fenotipos homocigóticos se parezca más el heterocigoto . Cuando el heterocigoto es indistinguible de uno de los homocigotos, el alelo expresado es el que conduce al fenotipo "dominante", [9] [10] y el otro alelo se dice que es "recesivo". El grado y el patrón de dominancia varía entre loci. Este tipo de interacción fue descrito formalmente por primera vez por Gregor Mendel . Sin embargo, muchos rasgos desafían esta categorización simple y los fenotipos están modelados por codominancia y herencia poligénica . [11]
El término alelo " de tipo salvaje " se utiliza a veces para describir un alelo que se cree que contribuye al carácter fenotípico típico que se observa en poblaciones "salvajes" de organismos, como las moscas de la fruta ( Drosophila melanogaster ). Históricamente, se consideraba que un alelo de "tipo salvaje" de este tipo conducía a un fenotipo dominante (abrumador, siempre expresado), común y normal, en contraste con los alelos " mutantes " que conducen a fenotipos recesivos, raros y con frecuencia perjudiciales. Anteriormente se pensaba que la mayoría de los individuos eran homocigotos para el alelo "de tipo salvaje" en la mayoría de los loci genéticos, y que cualquier alelo "mutante" alternativo se encontraba en forma homocigótica en una pequeña minoría de individuos "afectados", a menudo como enfermedades genéticas , y más frecuentemente en forma heterocigótica en " portadores " del alelo mutante. En la actualidad se sabe que la mayoría o todos los loci genéticos son altamente polimórficos, con múltiples alelos, cuyas frecuencias varían de una población a otra, y que una gran parte de la variación genética está oculta en forma de alelos que no producen diferencias fenotípicas obvias. Los alelos de tipo salvaje suelen indicarse con un signo más en superíndice ( es decir , p + para un alelo p ). [12]
Una población o especie de organismos incluye típicamente múltiples alelos en cada locus entre varios individuos. La variación alélica en un locus se puede medir como la cantidad de alelos ( polimorfismo ) presentes o la proporción de heterocigotos en la población. Un alelo nulo es una variante genética que carece de la función normal del gen porque no se expresa o la proteína expresada está inactiva.
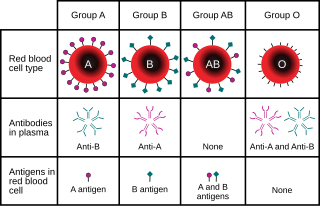
Por ejemplo, en el locus genético de los antígenos de carbohidratos del tipo sanguíneo ABO en humanos, [13] la genética clásica reconoce tres alelos, I A , I B e i, que determinan la compatibilidad de las transfusiones de sangre . Cualquier individuo tiene uno de los seis genotipos posibles (I A I A , I A i, I B I B , I B i , I A I B y ii) que producen uno de los cuatro fenotipos posibles : "Tipo A" (producido por genotipos homocigotos I A I A y heterocigotos I A i), "Tipo B" (producido por genotipos homocigotos I B I B y heterocigotos I B i), "Tipo AB" producido por el genotipo heterocigoto I A I B y "Tipo O" producido por el genotipo homocigoto ii. (Ahora se sabe que cada uno de los alelos A, B y O es en realidad una clase de alelos múltiples con diferentes secuencias de ADN que producen proteínas con propiedades idénticas: se conocen más de 70 alelos en el locus ABO. [14] Por lo tanto, un individuo con sangre "tipo A" puede ser un heterocigoto AO, un homocigoto AA o un heterocigoto AA con dos alelos "A" diferentes).
La frecuencia de los alelos en una población diploide se puede utilizar para predecir las frecuencias de los genotipos correspondientes (véase el principio de Hardy-Weinberg ). Para un modelo simple, con dos alelos;
donde p es la frecuencia de un alelo y q es la frecuencia del alelo alternativo, que necesariamente suman la unidad. Entonces, p 2 es la fracción de la población homocigótica para el primer alelo, 2 pq es la fracción de heterocigotos y q 2 es la fracción homocigótica para el alelo alternativo. Si el primer alelo es dominante con respecto al segundo, entonces la fracción de la población que mostrará el fenotipo dominante es p 2 + 2 pq , y la fracción con el fenotipo recesivo es q 2 .
Con tres alelos:
En el caso de múltiples alelos en un locus diploide, el número de genotipos posibles (G) con un número de alelos (a) viene dado por la expresión:
Una serie de trastornos genéticos se producen cuando un individuo hereda dos alelos recesivos para un rasgo de un solo gen. Los trastornos genéticos recesivos incluyen albinismo , fibrosis quística , galactosemia , fenilcetonuria (PKU) y enfermedad de Tay-Sachs . Otros trastornos también se deben a alelos recesivos, pero debido a que el locus del gen está ubicado en el cromosoma X, de modo que los hombres tienen solo una copia (es decir, son hemicigotos ), son más frecuentes en hombres que en mujeres. Algunos ejemplos incluyen daltonismo rojo-verde y síndrome del cromosoma X frágil .
Otros trastornos, como la enfermedad de Huntington , ocurren cuando un individuo hereda sólo un alelo dominante.
Si bien los rasgos hereditarios se estudian típicamente en términos de alelos genéticos, las marcas epigenéticas como la metilación del ADN se pueden heredar en regiones genómicas específicas en ciertas especies, un proceso denominado herencia epigenética transgeneracional . El término epialelo se utiliza para distinguir estas marcas hereditarias de los alelos tradicionales, que se definen por la secuencia de nucleótidos . [15] Se ha descubierto una clase específica de epialelo, los epialelos metaestables, en ratones y en humanos que se caracteriza por el establecimiento estocástico (probabilístico) del estado epigenético que se puede heredar mitóticamente. [16] [17]
El término "idiomorfo", del griego morphos (forma) e idio (singular, único), se introdujo en 1990 en lugar de "alelo" para denotar secuencias en el mismo locus en diferentes cepas que no tienen similitud de secuencia y probablemente no comparten una relación filogenética común. Se utiliza principalmente en la investigación genética de la micología . [18] [19]