

Una cápside es la cubierta proteica de un virus que encierra su material genético . Consta de varias subunidades estructurales oligoméricas (repetitivas) hechas de proteínas llamadas protómeros . Las subunidades morfológicas tridimensionales observables, que pueden corresponder o no a proteínas individuales, se denominan capsómeros . Las proteínas que forman la cápside se denominan proteínas de la cápside o proteínas de la cubierta viral ( VCP ). La cápside y el genoma interno se denomina nucleocápside .
Las cápsides se clasifican ampliamente según su estructura. La mayoría de los virus tienen cápsides con estructura helicoidal o icosaédrica [2] [3] . Algunos virus, como los bacteriófagos , han desarrollado estructuras más complicadas debido a limitaciones de elasticidad y electrostática. [4] La forma icosaédrica, que tiene 20 caras triangulares equiláteras, se aproxima a una esfera , mientras que la forma helicoidal se asemeja a la forma de un resorte , ocupando el espacio de un cilindro pero sin ser un cilindro en sí. [5] Las caras de la cápside pueden consistir en una o más proteínas. Por ejemplo, la cápside del virus de la fiebre aftosa tiene caras que constan de tres proteínas denominadas VP1–3. [6]
Algunos virus están envueltos , lo que significa que la cápside está recubierta con una membrana lipídica conocida como envoltura viral . La cápside adquiere la envoltura a partir de una membrana intracelular en el huésped del virus; los ejemplos incluyen la membrana nuclear interna, la membrana de Golgi y la membrana externa de la célula . [7]
Una vez que el virus ha infectado una célula y comienza a replicarse, se sintetizan nuevas subunidades de la cápside utilizando el mecanismo de biosíntesis de proteínas de la célula. En algunos virus, incluidos aquellos con cápsides helicoidales y especialmente aquellos con genomas de ARN, las proteínas de la cápside se coensamblan con sus genomas. En otros virus, especialmente en los más complejos con genomas de ADN de doble hebra, las proteínas de la cápside se ensamblan en procápsides precursoras vacías que incluyen una estructura portal especializada en un vértice. A través de este portal, el ADN viral se transloca a la cápside. [8]
Structural analyses of major capsid protein (MCP) architectures have been used to categorise viruses into lineages. For example, the bacteriophage PRD1, the algal virus Paramecium bursaria Chlorella virus-1 (PBCV-1), mimivirus and the mammalian adenovirus have been placed in the same lineage, whereas tailed, double-stranded DNA bacteriophages (Caudovirales) and herpesvirus belong to a second lineage.[9][10][11][12]


The icosahedral structure is extremely common among viruses. The icosahedron consists of 20 triangular faces delimited by 12 fivefold vertexes and consists of 60 asymmetric units. Thus, an icosahedral virus is made of 60N protein subunits. The number and arrangement of capsomeres in an icosahedral capsid can be classified using the "quasi-equivalence principle" proposed by Donald Caspar and Aaron Klug.[13] Like the Goldberg polyhedra, an icosahedral structure can be regarded as being constructed from pentamers and hexamers. The structures can be indexed by two integers h and k, with and ; the structure can be thought of as taking h steps from the edge of a pentamer, turning 60 degrees counterclockwise, then taking k steps to get to the next pentamer. The triangulation number T for the capsid is defined as:
In this scheme, icosahedral capsids contain 12 pentamers plus 10(T − 1) hexamers.[14][15] The T-number is representative of the size and complexity of the capsids.[16] Geometric examples for many values of h, k, and T can be found at List of geodesic polyhedra and Goldberg polyhedra.
Existen muchas excepciones a esta regla: por ejemplo, los poliomavirus y los papilomavirus tienen pentámeros en lugar de hexámeros en posiciones hexavalentes en una red cuasi T = 7. Los miembros del linaje de virus de ARN bicatenario, incluidos reovirus , rotavirus y bacteriófago φ6, tienen cápsides construidas con 120 copias de proteína de la cápside, correspondientes a una cápside T = 2, o posiblemente una cápside T = 1 con un dímero en la unidad asimétrica. De manera similar, muchos virus pequeños tienen una cápside pseudo T = 3 (o P = 3), que está organizada de acuerdo con una red T = 3, pero con polipéptidos distintos que ocupan las tres posiciones cuasi equivalentes [17]
Los números T se pueden representar de diferentes maneras, por ejemplo T = 1 solo se puede representar como un icosaedro o un dodecaedro y, dependiendo del tipo de cuasisimetría, T = 3 se puede presentar como un dodecaedro truncado , un icosidodecaedro , o un icosaedro truncado y sus respectivos duales un icosaedro triakis , un triacontaedro rómbico o un dodecaedro pentakis . [18] [ se necesita aclaración ]
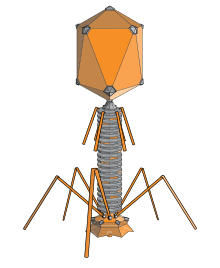
Un icosaedro alargado es una forma común de las cabezas de los bacteriófagos. Esta estructura se compone de un cilindro con una tapa en cada extremo. El cilindro está compuesto por 10 caras triangulares alargadas. El número Q (o T mid ), que puede ser cualquier número entero positivo, [19] especifica el número de triángulos, compuestos por subunidades asimétricas, que forman los 10 triángulos del cilindro. Las tapas se clasifican por el número T (o extremo T ). [20]
La bacteria E. coli es el huésped del bacteriófago T4 que tiene una estructura de cabeza alargada. La proteína gp31 codificada por el bacteriófago parece ser funcionalmente homóloga a la proteína chaperona GroES de E. coli y capaz de sustituirla en el ensamblaje de los viriones del bacteriófago T4 durante la infección. [21] Al igual que GroES, gp31 forma un complejo estable con la chaperonina GroEL que es absolutamente necesaria para el plegamiento y ensamblaje in vivo de la proteína gp23 de la cápsida principal del bacteriófago T4. [21]

Muchos virus de plantas filamentosos y con forma de bastón tienen cápsides con simetría helicoidal . [22] La estructura helicoidal se puede describir como un conjunto de n hélices moleculares 1-D relacionadas por una simetría axial de n veces. [23] Las transformaciones helicoidales se clasifican en dos categorías: sistemas helicoidales unidimensionales y bidimensionales. [23] La creación de una estructura helicoidal completa se basa en un conjunto de matrices traslacionales y rotacionales que están codificadas en el banco de datos de proteínas. [23] La simetría helicoidal viene dada por la fórmula P = μ x ρ , donde μ es el número de unidades estructurales por vuelta de la hélice, ρ es el ascenso axial por unidad y P es el paso de la hélice. Se dice que la estructura es abierta debido a la característica de que se puede encerrar cualquier volumen variando la longitud de la hélice. [24] El virus helicoidal más comprendido es el virus del mosaico del tabaco. [22] El virus es una sola molécula de ARN de cadena (+). Cada proteína de cubierta en el interior de la hélice se une a tres nucleótidos del genoma de ARN. Los virus de la influenza A se diferencian por comprender múltiples ribonucleoproteínas; la proteína viral NP organiza el ARN en una estructura helicoidal. El tamaño también es diferente; el virus del mosaico del tabaco tiene 16,33 subunidades proteicas por vuelta helicoidal, [22] mientras que el virus de la influenza A tiene un bucle en la cola de 28 aminoácidos. [25]
Las funciones de la cápside son:
El virus debe ensamblar una capa proteica protectora estable para proteger el genoma de agentes químicos y físicos letales. Estos incluyen extremos de pH o temperatura y enzimas proteolíticas y nucleolíticas . Para los virus sin envoltura, la propia cápside puede participar en la interacción con los receptores de la célula huésped, lo que lleva a la penetración de la membrana de la célula huésped y la internalización de la cápside. La entrega del genoma se produce mediante el descubrimiento o desmontaje posterior de la cápside y la liberación del genoma en el citoplasma, o mediante la expulsión del genoma a través de una estructura portal especializada directamente en el núcleo de la célula huésped.
Se ha sugerido que muchas proteínas de la cápside viral han evolucionado en múltiples ocasiones a partir de proteínas celulares funcionalmente diversas. [26] El reclutamiento de proteínas celulares parece haber ocurrido en diferentes etapas de la evolución, de modo que algunas proteínas celulares fueron capturadas y refuncionalizadas antes de la divergencia de los organismos celulares en los tres dominios contemporáneos de la vida, mientras que otras fueron secuestradas hace relativamente poco tiempo. Como resultado, algunas proteínas de la cápside están muy extendidas en virus que infectan organismos lejanamente relacionados (p. ej., proteínas de la cápside con pliegue en gelatina ), mientras que otras están restringidas a un grupo particular de virus (p. ej., proteínas de la cápside de alfavirus). [26] [27]
Un modelo computacional (2015) ha demostrado que las cápsides pueden haberse originado antes que los virus y que sirvieron como medio de transferencia horizontal entre comunidades replicadoras ya que estas comunidades no podrían sobrevivir si aumentara el número de genes parásitos, siendo ciertos genes los responsables de la formación. de estas estructuras y aquellas que favorecían la supervivencia de comunidades autorreplicantes. [28] El desplazamiento de estos genes ancestrales entre organismos celulares podría favorecer la aparición de nuevos virus durante la evolución. [27]