

La estructura de las proteínas es la disposición tridimensional de los átomos en una molécula de cadena de aminoácidos . Las proteínas son polímeros , específicamente polipéptidos , formados a partir de secuencias de aminoácidos , que son los monómeros del polímero. Un solo monómero de aminoácido también puede llamarse residuo , lo que indica una unidad repetitiva de un polímero. Las proteínas se forman cuando los aminoácidos experimentan reacciones de condensación , en las que los aminoácidos pierden una molécula de agua por reacción para unirse entre sí con un enlace peptídico . Por convención, una cadena de menos de 30 aminoácidos a menudo se identifica como un péptido , en lugar de una proteína. [1] Para poder realizar su función biológica, las proteínas se pliegan en una o más conformaciones espaciales específicas impulsadas por una serie de interacciones no covalentes , como enlaces de hidrógeno , interacciones iónicas , fuerzas de Van der Waals y empaquetamiento hidrofóbico . Para comprender las funciones de las proteínas a nivel molecular, a menudo es necesario determinar su estructura tridimensional . Este es el tema del campo científico de la biología estructural , que emplea técnicas como la cristalografía de rayos X , la espectroscopia de RMN , la criomicroscopía electrónica (crio-EM) y la interferometría de polarización dual , para determinar la estructura de las proteínas.
Las estructuras proteicas varían en tamaño desde decenas hasta varios miles de aminoácidos. [2] Por tamaño físico, las proteínas se clasifican como nanopartículas , entre 1 y 100 nm. Se pueden formar complejos proteicos muy grandes a partir de subunidades proteicas . Por ejemplo, muchos miles de moléculas de actina se ensamblan en un microfilamento .
Una proteína suele sufrir cambios estructurales reversibles al realizar su función biológica. Las estructuras alternativas de la misma proteína se denominan conformaciones diferentes y las transiciones entre ellas se denominan cambios conformacionales .
Hay cuatro niveles distintos de estructura de proteínas.
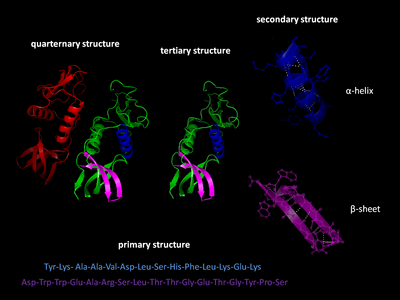
La estructura primaria de una proteína se refiere a la secuencia de aminoácidos en la cadena polipeptídica. La estructura primaria se mantiene unida por enlaces peptídicos que se forman durante el proceso de biosíntesis de proteínas . Los dos extremos de la cadena polipeptídica se denominan extremo carboxilo (C-terminal) y extremo amino (N-terminal) según la naturaleza del grupo libre en cada extremo. El recuento de residuos siempre comienza en el extremo N-terminal ( grupo NH2), que es el extremo donde el grupo amino no está involucrado en un enlace peptídico. La estructura primaria de una proteína está determinada por el gen correspondiente a la proteína. Una secuencia específica de nucleótidos en el ADN se transcribe en ARNm , que es leído por el ribosoma en un proceso llamado traducción . La secuencia de aminoácidos en la insulina fue descubierta por Frederick Sanger , estableciendo que las proteínas tienen secuencias de aminoácidos definitorias. [3] [4] La secuencia de una proteína es única para esa proteína y define la estructura y función de la proteína. La secuencia de una proteína se puede determinar mediante métodos como la degradación de Edman o la espectrometría de masas en tándem . Sin embargo, a menudo se lee directamente de la secuencia del gen utilizando el código genético . Se recomienda estrictamente utilizar las palabras "residuos de aminoácidos" cuando se habla de proteínas porque cuando se forma un enlace peptídico, se pierde una molécula de agua y, por lo tanto, las proteínas están formadas por residuos de aminoácidos. Las modificaciones postraduccionales , como las fosforilaciones y las glicosilaciones , generalmente también se consideran parte de la estructura primaria y no se pueden leer a partir del gen. Por ejemplo, la insulina está compuesta de 51 aminoácidos en 2 cadenas. Una cadena tiene 31 aminoácidos y la otra tiene 20 aminoácidos.

La estructura secundaria se refiere a subestructuras locales altamente regulares en la cadena principal del polipéptido real. Linus Pauling sugirió en 1951 dos tipos principales de estructura secundaria, la hélice α y la cadena β o láminas β . [5] Estas estructuras secundarias se definen por patrones de enlaces de hidrógeno entre los grupos peptídicos de la cadena principal. Tienen una geometría regular, estando restringidas a valores específicos de los ángulos diedros ψ y φ en el diagrama de Ramachandran . Tanto la hélice α como la lámina β representan una forma de saturar todos los donantes y aceptores de enlaces de hidrógeno en la cadena principal del péptido. Algunas partes de la proteína están ordenadas pero no forman ninguna estructura regular. No deben confundirse con la espiral aleatoria , una cadena polipeptídica desplegada que carece de cualquier estructura tridimensional fija. Varias estructuras secundarias secuenciales pueden formar una " unidad supersecundaria ". [6]
La estructura terciaria se refiere a la estructura tridimensional creada por una sola molécula de proteína (una sola cadena polipeptídica ). Puede incluir uno o varios dominios . Las hélices α y las láminas plegadas β se pliegan en una estructura globular compacta . El plegamiento es impulsado por las interacciones hidrofóbicas no específicas , el entierro de residuos hidrofóbicos del agua , pero la estructura es estable solo cuando las partes de un dominio de proteína están bloqueadas en su lugar por interacciones terciarias específicas , como puentes salinos , enlaces de hidrógeno y el empaquetamiento apretado de cadenas laterales y enlaces disulfuro . Los enlaces disulfuro son extremadamente raros en las proteínas citosólicas, ya que el citosol (fluido intracelular) es generalmente un entorno reductor .
La estructura cuaternaria es la estructura tridimensional que consiste en la agregación de dos o más cadenas polipeptídicas individuales (subunidades) que operan como una sola unidad funcional ( multímero ). El multímero resultante se estabiliza mediante las mismas interacciones no covalentes y enlaces disulfuro que en la estructura terciaria. Hay muchas posibles organizaciones de estructura cuaternaria. [7] Los complejos de dos o más polipéptidos (es decir, múltiples subunidades) se denominan multímeros . Específicamente, se llamaría dímero si contiene dos subunidades, trímero si contiene tres subunidades, tetrámero si contiene cuatro subunidades y pentámero si contiene cinco subunidades, y así sucesivamente. Las subunidades se relacionan frecuentemente entre sí mediante operaciones de simetría , como un eje de 2 pliegues en un dímero. Los multímeros formados por subunidades idénticas se denominan con el prefijo "homo-" y los formados por subunidades diferentes se denominan con el prefijo "hetero-", por ejemplo, un heterotetrámero, como las dos cadenas alfa y dos beta de la hemoglobina .
Un ensamblaje de múltiples copias de una cadena polipeptídica particular puede describirse como un homómero , multímero u oligómero . Bertolini et al. en 2021 [8] presentaron evidencia de que la formación de homómeros puede ser impulsada por la interacción entre cadenas polipeptídicas nacientes a medida que son traducidas del ARNm por ribosomas adyacentes cercanos . Se han identificado cientos de proteínas que se ensamblan en homómeros en células humanas. [8] El proceso de ensamblaje a menudo se inicia mediante la interacción de la región N-terminal de las cadenas polipeptídicas. En 1965 se revisó la evidencia de que numerosos productos genéticos forman homómeros (multímeros) en una variedad de organismos con base en evidencia de complementación intragénica . [9]

Las proteínas se describen frecuentemente como compuestas de varias unidades estructurales. Estas unidades incluyen dominios, motivos y pliegues. A pesar de que existen alrededor de 100.000 proteínas diferentes expresadas en sistemas eucariotas , hay muchos menos dominios, motivos estructurales y pliegues diferentes.
Un dominio estructural es un elemento de la estructura general de la proteína que se autoestabiliza y a menudo se pliega independientemente del resto de la cadena proteica. Muchos dominios no son exclusivos de los productos proteicos de un gen o una familia de genes , sino que aparecen en una variedad de proteínas. Los dominios a menudo se nombran y se distinguen porque figuran de manera destacada en la función biológica de la proteína a la que pertenecen; por ejemplo, el " dominio de unión al calcio de la calmodulina ". Debido a que son estables de forma independiente, los dominios pueden "intercambiarse" mediante ingeniería genética entre una proteína y otra para formar proteínas quimeras . Una combinación conservadora de varios dominios que se encuentran en diferentes proteínas, como el dominio de la proteína tirosina fosfatasa y el par de dominios C2 , se denominó "superdominio" que puede evolucionar como una sola unidad. [10]
Los motivos estructurales y de secuencia se refieren a segmentos cortos de la estructura tridimensional de una proteína o secuencia de aminoácidos que se encontraron en una gran cantidad de proteínas diferentes.
Las estructuras terciarias de las proteínas pueden tener múltiples elementos secundarios en la misma cadena polipeptídica. La estructura supersecundaria se refiere a una combinación específica de elementos de estructura secundaria , como unidades β-α-β o un motivo hélice-giro-hélice . Algunos de ellos también pueden denominarse motivos estructurales.
Un pliegue de proteína se refiere a la arquitectura general de la proteína, como un haz de hélice , un barril β , un pliegue de Rossmann o diferentes "pliegues" proporcionados en la base de datos de Clasificación estructural de proteínas . [11] Un concepto relacionado es la topología de proteínas .
Las proteínas no son objetos estáticos, sino que pueblan conjuntos de estados conformacionales . Las transiciones entre estos estados ocurren típicamente en nanoescalas , y han sido vinculadas a fenómenos funcionalmente relevantes tales como señalización alostérica [12] y catálisis enzimática . [13] La dinámica de proteínas y los cambios conformacionales permiten que las proteínas funcionen como máquinas biológicas a nanoescala dentro de las células, a menudo en forma de complejos multiproteicos . [14] Los ejemplos incluyen proteínas motoras , tales como miosina , que es responsable de la contracción muscular , kinesina , que mueve carga dentro de las células lejos del núcleo a lo largo de microtúbulos , y dineína , que mueve carga dentro de las células hacia el núcleo y produce el latido axonemal de cilios móviles y flagelos . "En efecto, el cilio móvil es una nanomáquina compuesta por quizás más de 600 proteínas en complejos moleculares, muchos de los cuales también funcionan independientemente como nanomáquinas... Los enlaces flexibles permiten que los dominios proteicos móviles conectados por ellos recluten a sus socios de unión e induzcan alosterio de largo alcance a través de la dinámica de los dominios proteicos ". [15]

Las proteínas suelen considerarse estructuras terciarias relativamente estables que experimentan cambios conformacionales después de verse afectadas por interacciones con otras proteínas o como parte de la actividad enzimática. Sin embargo, las proteínas pueden tener distintos grados de estabilidad, y algunas de las variantes menos estables son proteínas intrínsecamente desordenadas . Estas proteínas existen y funcionan en un estado relativamente "desordenado" que carece de una estructura terciaria estable . Como resultado, son difíciles de describir mediante una única estructura terciaria fija . Se han ideado conjuntos conformacionales como una forma de proporcionar una representación más precisa y "dinámica" del estado conformacional de las proteínas intrínsecamente desordenadas . [17] [16]
Los archivos de conjuntos de proteínas son una representación de una proteína que se puede considerar que tiene una estructura flexible. La creación de estos archivos requiere determinar cuáles de las diversas conformaciones de proteínas teóricamente posibles existen realmente. Un enfoque es aplicar algoritmos computacionales a los datos de proteínas para intentar determinar el conjunto de conformaciones más probable para un archivo de conjunto . Existen múltiples métodos para preparar datos para la base de datos de conjuntos de proteínas que se dividen en dos metodologías generales: enfoques de agrupación y de dinámica molecular (MD) (diagramados en la figura). El enfoque basado en agrupación utiliza la secuencia de aminoácidos de la proteína para crear una gran cantidad de conformaciones aleatorias. Luego, esta agrupación se somete a un mayor procesamiento computacional que crea un conjunto de parámetros teóricos para cada conformación en función de la estructura. Se seleccionan los subconjuntos conformacionales de esta agrupación cuyos parámetros teóricos promedio coinciden estrechamente con los datos experimentales conocidos para esta proteína. El enfoque alternativo de dinámica molecular toma múltiples conformaciones aleatorias a la vez y las somete a todas a datos experimentales. Aquí, los datos experimentales sirven como limitaciones que se deben colocar en las conformaciones (por ejemplo, distancias conocidas entre átomos). Sólo se aceptan las conformaciones que logran permanecer dentro de los límites establecidos por los datos experimentales. Este enfoque a menudo aplica grandes cantidades de datos experimentales a las conformaciones, lo que es una tarea muy exigente desde el punto de vista computacional. [16]
Los conjuntos conformacionales se generaron para una serie de proteínas altamente dinámicas y parcialmente desplegadas, como Sic1 / Cdc4 , [18] p15 PAF , [19] MKK7 , [20] Beta-sinucleína [21] y P27 [22].
Tal como se traduce, los polipéptidos salen del ribosoma principalmente como una espiral aleatoria y se pliegan a su estado nativo . [23] [24] Generalmente se supone que la estructura final de la cadena de proteínas está determinada por su secuencia de aminoácidos ( dogma de Anfinsen ). [25]
La estabilidad termodinámica de las proteínas representa la diferencia de energía libre entre los estados plegado y desplegado de la proteína. Esta diferencia de energía libre es muy sensible a la temperatura, por lo que un cambio de temperatura puede provocar el desplegado o la desnaturalización. La desnaturalización de la proteína puede provocar la pérdida de la función y la pérdida del estado nativo. La energía libre de estabilización de las proteínas globulares solubles normalmente no supera los 50 kJ/mol. [ cita requerida ] Teniendo en cuenta la gran cantidad de enlaces de hidrógeno que se producen para la estabilización de las estructuras secundarias y la estabilización del núcleo interno a través de interacciones hidrofóbicas, la energía libre de estabilización surge como una pequeña diferencia entre grandes cantidades. [26]


Alrededor del 90% de las estructuras proteínicas disponibles en el Protein Data Bank se han determinado mediante cristalografía de rayos X. [27] Este método permite medir la distribución tridimensional (3-D) de la densidad de electrones en la proteína, en el estado cristalizado , y de ese modo inferir las coordenadas 3-D de todos los átomos que se van a determinar con una cierta resolución. Aproximadamente el 7% de las estructuras proteínicas conocidas se han obtenido mediante técnicas de resonancia magnética nuclear (RMN). [28] Para complejos proteínicos más grandes, la criomicroscopía electrónica puede determinar las estructuras proteínicas. La resolución es típicamente menor que la de la cristalografía de rayos X, o RMN, pero la resolución máxima aumenta constantemente. Esta técnica sigue siendo particularmente valiosa para complejos proteínicos muy grandes, como las proteínas de la cubierta del virus y las fibras amiloides .
La composición general de la estructura secundaria se puede determinar mediante dicroísmo circular . La espectroscopia vibracional también se puede utilizar para caracterizar la conformación de péptidos, polipéptidos y proteínas. [29] La espectroscopia infrarroja bidimensional se ha convertido en un método valioso para investigar las estructuras de péptidos y proteínas flexibles que no se pueden estudiar con otros métodos. [30] [31] A menudo se obtiene una imagen más cualitativa de la estructura de la proteína mediante proteólisis , que también es útil para detectar muestras de proteínas más cristalizables. Las nuevas implementaciones de este enfoque, incluida la proteólisis paralela rápida (FASTpp) , pueden investigar la fracción estructurada y su estabilidad sin la necesidad de purificación. [32] Una vez que se ha determinado experimentalmente la estructura de una proteína, se pueden realizar estudios más detallados computacionalmente, utilizando simulaciones dinámicas moleculares de esa estructura. [33]
Una base de datos de estructura de proteínas es una base de datos que se modela en torno a las diversas estructuras de proteínas determinadas experimentalmente. El objetivo de la mayoría de las bases de datos de estructura de proteínas es organizar y anotar las estructuras de proteínas, proporcionando a la comunidad biológica acceso a los datos experimentales de una manera útil. Los datos incluidos en las bases de datos de estructura de proteínas a menudo incluyen coordenadas 3D, así como información experimental, como dimensiones de celdas unitarias y ángulos para estructuras determinadas por cristalografía de rayos X. Aunque la mayoría de los casos, en este caso proteínas o determinaciones de estructura específicas de una proteína, también contienen información de secuencia y algunas bases de datos incluso proporcionan medios para realizar consultas basadas en secuencias, el atributo principal de una base de datos de estructura es la información estructural, mientras que las bases de datos de secuencias se centran en la información de secuencias y no contienen información estructural para la mayoría de las entradas. Las bases de datos de estructura de proteínas son fundamentales para muchos esfuerzos en biología computacional , como el diseño de fármacos basado en la estructura , tanto en el desarrollo de los métodos computacionales utilizados como en el suministro de un gran conjunto de datos experimentales utilizados por algunos métodos para proporcionar información sobre la función de una proteína. [34]
Las estructuras de proteínas se pueden agrupar en función de su similitud estructural, clase topológica o un origen evolutivo común . La base de datos de Clasificación estructural de proteínas [35] y la base de datos CATH [36] proporcionan dos clasificaciones estructurales diferentes de proteínas. Cuando la similitud estructural es grande, las dos proteínas posiblemente hayan divergido de un ancestro común, [37] y la estructura compartida entre proteínas se considera evidencia de homología . La similitud estructural se puede utilizar entonces para agrupar proteínas en superfamilias de proteínas . [38] Si la estructura compartida es significativa pero la fracción compartida es pequeña, el fragmento compartido puede ser la consecuencia de un evento evolutivo más dramático como la transferencia horizontal de genes , y la unión de proteínas que comparten estos fragmentos en superfamilias de proteínas ya no está justificada. [37] La topología de una proteína también se puede utilizar para clasificar proteínas. La teoría de nudos y la topología de circuitos son dos marcos topológicos desarrollados para la clasificación de pliegues de proteínas basados en el cruce de cadenas y los contactos intracadena respectivamente.
La generación de una secuencia de proteínas es mucho más sencilla que la determinación de su estructura. Sin embargo, la estructura de una proteína proporciona mucha más información sobre su función que su secuencia. Por lo tanto, se han desarrollado varios métodos para la predicción computacional de la estructura de una proteína a partir de su secuencia. [39] Los métodos de predicción ab initio utilizan solo la secuencia de la proteína. Los métodos de modelado por homología y enhebrado pueden construir un modelo 3D para una proteína de estructura desconocida a partir de estructuras experimentales de proteínas relacionadas evolutivamente, llamadas familia de proteínas .
{{cite book}}: |journal=ignorado ( ayuda )