La traducción eucariota es el proceso biológico mediante el cual el ARN mensajero se traduce en proteínas en eucariotas . Consta de cuatro fases: iniciación, elongación, terminación y reencapsulación.

La iniciación de la traducción es el proceso por el cual el ribosoma y sus factores asociados se unen a un ARNm y se ensamblan en el codón de inicio. Este proceso se define como dependiente de la caperuza, en el que el ribosoma se une inicialmente a la caperuza 5' y luego viaja hasta el codón de terminación, o como independiente de la caperuza, en el que el ribosoma no se une inicialmente a la caperuza 5'.
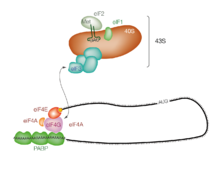
La iniciación de la traducción generalmente implica la interacción de ciertas proteínas clave, los factores de iniciación , con una etiqueta especial unida al extremo 5' de una molécula de ARNm, la tapa 5' , así como con el UTR 5' . Estas proteínas se unen a la subunidad ribosómica pequeña (40S) y mantienen el ARNm en su lugar. [1]
eIF3 está asociado con la subunidad ribosomal 40S y desempeña un papel en evitar que la subunidad ribosomal grande (60S) se una prematuramente. eIF3 también interactúa con el complejo eIF4F , que consta de otros tres factores de iniciación: eIF4A , eIF4E y eIF4G . eIF4G es una proteína de andamiaje que se asocia directamente con eIF3 y los otros dos componentes. eIF4E es la proteína de unión a la caperuza. La unión de la caperuza por eIF4E a menudo se considera el paso limitante de la velocidad de la iniciación dependiente de la caperuza, y la concentración de eIF4E es un nexo regulador del control de la traducción. Ciertos virus escinden una porción de eIF4G que se une a eIF4E, evitando así que la traducción dependiente de la caperuza secuestre la maquinaria del huésped a favor de los mensajes virales (independientes de la caperuza). eIF4A es una helicasa de ARN dependiente de ATP que ayuda al ribosoma al resolver ciertas estructuras secundarias formadas a lo largo de la transcripción del ARNm. [2] La proteína de unión a poli(A) (PABP) también se asocia con el complejo eIF4F a través de eIF4G y se une a la cola de poli(A) de la mayoría de las moléculas de ARNm eucariotas. Se ha implicado a esta proteína en la circularización del ARNm durante la traducción. [3]
Este complejo de preiniciación 43S (43S PIC) acompañado por los factores proteicos se mueve a lo largo de la cadena de ARNm hacia su extremo 3', en un proceso conocido como 'escaneo', para alcanzar el codón de inicio (típicamente AUG). En eucariotas y arqueas , el aminoácido codificado por el codón de inicio es metionina . El ARNt iniciador cargado con Met (Met-ARNt i Met ) es llevado al sitio P de la subunidad ribosómica pequeña por el factor de iniciación eucariota 2 (eIF2) . Hidroliza GTP y señala la disociación de varios factores de la subunidad ribosómica pequeña, lo que finalmente conduce a la asociación de la subunidad grande (o subunidad 60S ). El ribosoma completo ( 80S ) luego comienza la elongación de la traducción.
La regulación de la síntesis de proteínas está parcialmente influenciada por la fosforilación de eIF2 (a través de la subunidad α), que es parte del complejo ternario eIF2-GTP-Met-ARNt i Met (eIF2-TC). Cuando se fosforilan grandes cantidades de eIF2, se inhibe la síntesis de proteínas. Esto ocurre en condiciones de privación de aminoácidos o después de una infección viral. Sin embargo, una pequeña fracción de este factor de iniciación se fosforila de forma natural. Otro regulador es 4EBP , que se une al factor de iniciación eIF4E e inhibe sus interacciones con eIF4G , impidiendo así la iniciación dependiente de cap. Para oponerse a los efectos de 4EBP, los factores de crecimiento fosforilan 4EBP, reduciendo su afinidad por eIF4E y permitiendo la síntesis de proteínas. [ cita requerida ]
Si bien la síntesis de proteínas está regulada globalmente mediante la modulación de la expresión de factores de iniciación clave, así como del número de ribosomas, los ARNm individuales pueden tener diferentes tasas de traducción debido a la presencia de elementos de secuencia reguladores. Se ha demostrado que esto es importante en una variedad de entornos, incluida la meiosis de la levadura y la respuesta al etileno en las plantas. Además, trabajos recientes en levaduras y seres humanos sugieren que la divergencia evolutiva en secuencias reguladoras cis puede afectar la regulación de la traducción. [4] Además, las helicasas de ARN como DHX29 y Ded1/DDX3 pueden participar en el proceso de iniciación de la traducción, especialmente para ARNm con 5'UTR estructurados. [5]
El ejemplo mejor estudiado de iniciación de la traducción independiente de la caperuza en eucariotas utiliza el sitio de entrada interno del ribosoma (IRES). A diferencia de la traducción dependiente de la caperuza, la traducción independiente de la caperuza no requiere una caperuza 5' para iniciar el escaneo desde el extremo 5' del ARNm hasta el codón de inicio. El ribosoma puede localizarse en el sitio de inicio mediante unión directa, factores de iniciación y/o ITAF (factores transactivos de IRES) sin necesidad de escanear todo el UTR 5' . Este método de traducción es importante en condiciones que requieren la traducción de ARNm específicos durante el estrés celular, cuando la traducción general se reduce. Los ejemplos incluyen factores que responden a la apoptosis y respuestas inducidas por estrés. [6]
La elongación depende de factores de elongación eucariotas . Al final del paso de iniciación, el ARNm se posiciona de manera que el siguiente codón pueda traducirse durante la etapa de elongación de la síntesis de proteínas. El ARNt iniciador ocupa el sitio P en el ribosoma , y el sitio A está listo para recibir un aminoacil-ARNt. Durante la elongación de la cadena, cada aminoácido adicional se agrega a la cadena polipeptídica naciente en un microciclo de tres pasos. Los pasos en este microciclo son (1) posicionar el aminoacil-ARNt correcto en el sitio A del ribosoma, que es llevado a ese sitio por eEF1, (2) formar el enlace peptídico y (3) desplazar el ARNm por un codón en relación con el ribosoma con la ayuda de eEF2. A diferencia de las bacterias, en las que la iniciación de la traducción ocurre tan pronto como se sintetiza el extremo 5' de un ARNm, en los eucariotas, este acoplamiento estrecho entre la transcripción y la traducción no es posible porque la transcripción y la traducción se llevan a cabo en compartimentos separados de la célula (el núcleo y el citoplasma ). Los precursores eucariotas del ARNm deben procesarse en el núcleo (p. ej., capping, poliadenilación , empalme) en los ribosomas antes de ser exportados al citoplasma para su traducción. La traducción también puede verse afectada por la pausa ribosómica , que puede desencadenar un ataque endonucleolítico del ARNt, un proceso denominado desintegración del ARNm sin efecto. La pausa ribosómica también ayuda al plegamiento co-traduccional del polipéptido naciente en el ribosoma y retrasa la traducción de la proteína mientras está codificando el ARNt. Esto puede desencadenar el cambio de marco ribosómico. [7]
La terminación de la elongación depende de factores de liberación eucariotas . El proceso es similar al de la terminación bacteriana , pero a diferencia de la terminación bacteriana, existe un factor de liberación universal , eRF1, que reconoce los tres codones de terminación. Tras la terminación, el ribosoma se desmonta y se libera el polipéptido completo. eRF3 es una GTPasa dependiente del ribosoma que ayuda a eRF1 a liberar el polipéptido completo. El genoma humano codifica unos pocos genes cuyo codón de terminación del ARNm es sorprendentemente permeable: en estos genes, la terminación de la traducción es ineficiente debido a bases de ARN especiales en la proximidad del codón de terminación. La terminación permeable en estos genes conduce a una lectura transversa de hasta el 10% de los codones de terminación de estos genes. Algunos de estos genes codifican dominios proteicos funcionales en su extensión de lectura transversa de modo que pueden surgir nuevas isoformas proteicas . Este proceso se ha denominado "lectura transversa funcional". [8]
La traducción es uno de los principales consumidores de energía en las células, por lo que está estrictamente regulada. Se han desarrollado numerosos mecanismos que controlan y regulan la traducción tanto en eucariotas como en procariotas . La regulación de la traducción puede afectar la tasa global de síntesis de proteínas, que está estrechamente relacionada con el estado metabólico y proliferativo de una célula. Para profundizar en este intrincado proceso, los científicos suelen utilizar una técnica conocida como perfil ribosómico. [9] Este método permite a los investigadores tomar una instantánea del translatoma, mostrando qué partes del ARNm están siendo traducidas en proteínas por los ribosomas en un momento dado. El perfil ribosómico proporciona información valiosa sobre la dinámica de la traducción, revelando la compleja interacción entre la secuencia genética, la estructura del ARNm y la regulación de la traducción. Ampliando este concepto, un desarrollo más reciente es el perfil ribosómico de una sola célula, una técnica que nos permite estudiar el proceso de traducción a la resolución de células individuales. [10] El perfil de ribosomas de células individuales tiene el potencial de arrojar luz sobre la naturaleza heterogénea de las células, lo que conduce a una comprensión más matizada de cómo la regulación de la traducción puede afectar el comportamiento celular, el estado metabólico y la capacidad de respuesta a diversos estímulos o condiciones.
En algunas células, ciertos aminoácidos pueden agotarse y, por lo tanto, afectar la eficiencia de la traducción. Por ejemplo, las células T activadas secretan interferón-γ , que desencadena la escasez intracelular de triptófano al regular positivamente la enzima indolamina 2,3-dioxigenasa 1 (IDO1). Sorprendentemente, a pesar del agotamiento del triptófano , la síntesis de proteínas en marco continúa a través de los codones de triptófano . Esto se logra mediante la incorporación de fenilalanina en lugar de triptófano. Los péptidos resultantes se denominan "sustituyentes" W>F. Estos sustituyentes W>F son abundantes en ciertos tipos de cáncer y se han asociado con una mayor expresión de IDO1. Funcionalmente, los sustituyentes W>F pueden perjudicar la actividad de las proteínas . [11]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )