La reacción en cadena de la polimerasa en tiempo real ( PCR en tiempo real o qPCR cuando se utiliza cuantitativamente) es una técnica de laboratorio de biología molecular basada en la reacción en cadena de la polimerasa (PCR). Monitorea la amplificación de una molécula de ADN objetivo durante la PCR (es decir, en tiempo real), no al final de la misma, como en la PCR convencional. La PCR en tiempo real se puede utilizar cuantitativamente y semicuantitativamente (es decir, por encima o por debajo de una cierta cantidad de moléculas de ADN).
Dos métodos comunes para la detección de productos de PCR en PCR en tiempo real son (1) colorantes fluorescentes no específicos que se intercalan con cualquier ADN bicatenario y (2) sondas de ADN específicas de secuencia que consisten en oligonucleótidos marcados con un reportero fluorescente , que permite la detección solo después de la hibridación de la sonda con su secuencia complementaria.
Las directrices de Información mínima para la publicación de experimentos de PCR cuantitativa en tiempo real ( MIQE ) proponen que se utilice la abreviatura qPCR para PCR cuantitativa en tiempo real y que se utilice RT-qPCR para transcripción inversa (qPCR). [1] El acrónimo "RT-PCR" comúnmente denota reacción en cadena de la polimerasa con transcripción inversa y no PCR en tiempo real, pero no todos los autores se adhieren a esta convención. [2]

Las células de todos los organismos regulan la expresión génica mediante el recambio de las transcripciones génicas ( ARN monocatenario ): la cantidad de un gen expresado en una célula se puede medir por el número de copias de una transcripción de ARN de ese gen presente en una muestra. Para detectar y cuantificar de forma robusta la expresión génica a partir de pequeñas cantidades de ARN, es necesaria la amplificación de la transcripción génica. La reacción en cadena de la polimerasa (PCR) es un método común para amplificar el ADN; para la PCR basada en ARN, la muestra de ARN primero se transcribe de forma inversa a ADN complementario (ADNc) con transcriptasa inversa .
Para amplificar pequeñas cantidades de ADN se utiliza la misma metodología que en la PCR convencional utilizando un molde de ADN, al menos un par de cebadores específicos , desoxirribonucleótidos trifosfato, una solución tampón adecuada y una ADN polimerasa termoestable . A esta mezcla se añade una sustancia marcada con un fluoróforo en un termociclador que contiene sensores para medir la fluorescencia del fluoróforo después de haber sido excitado a la longitud de onda requerida permitiendo medir la velocidad de generación de uno o más productos específicos. Esto permite medir la velocidad de generación del producto amplificado en cada ciclo de PCR. Los datos así generados pueden analizarse mediante un software informático para calcular la expresión génica relativa (o número de copias de ARNm ) en varias muestras. La PCR cuantitativa también puede aplicarse a la detección y cuantificación de ADN en muestras para determinar la presencia y abundancia de una secuencia de ADN particular en estas muestras. [3] Esta medición se realiza después de cada ciclo de amplificación, y esta es la razón por la que este método se llama PCR en tiempo real (es decir, PCR inmediata o simultánea).
La PCR cuantitativa y la micromatriz de ADN son metodologías modernas para estudiar la expresión génica . Se utilizaban métodos más antiguos para medir la abundancia de ARNm: visualización diferencial , ensayo de protección de ARNasa y transferencia Northern . La transferencia Northern se utiliza a menudo para estimar el nivel de expresión de un gen visualizando la abundancia de su transcripción de ARNm en una muestra. En este método, el ARN purificado se separa mediante electroforesis en gel de agarosa , se transfiere a una matriz sólida (como una membrana de nailon) y se sondea con una sonda de ADN o ARN específica que es complementaria al gen de interés. Aunque esta técnica todavía se utiliza para evaluar la expresión génica, requiere cantidades relativamente grandes de ARN y proporciona solo información cualitativa o semicuantitativa de los niveles de ARNm. [4] Los errores de estimación que surgen de las variaciones en el método de cuantificación pueden ser el resultado de la integridad del ADN, la eficiencia enzimática y muchos otros factores. Por esta razón, se han desarrollado varios sistemas de estandarización (a menudo llamados métodos de normalización ). Algunos de ellos se han desarrollado para cuantificar la expresión génica total, pero los más comunes tienen como objetivo cuantificar el gen específico que se está estudiando en relación con otro gen llamado gen normalizador, que se selecciona por su nivel de expresión casi constante. Estos genes se seleccionan a menudo entre los genes de mantenimiento , ya que sus funciones relacionadas con la supervivencia celular básica normalmente implican la expresión génica constitutiva . [5] [6] Esto permite a los investigadores informar una relación de la expresión de los genes de interés dividida por la expresión del normalizador seleccionado, lo que permite la comparación del primero sin conocer realmente su nivel absoluto de expresión.
Los genes normalizadores más utilizados son aquellos que codifican las siguientes moléculas: tubulina , gliceraldehído-3-fosfato deshidrogenasa , albúmina , ciclofilina y ARN ribosómico . [4]
La PCR en tiempo real se lleva a cabo en un termociclador con capacidad para iluminar cada muestra con un haz de luz de al menos una longitud de onda especificada y detectar la fluorescencia emitida por el fluoróforo excitado . El termociclador también es capaz de calentar y enfriar rápidamente las muestras, aprovechando así las propiedades fisicoquímicas de los ácidos nucleicos y de la ADN polimerasa .
El proceso de PCR generalmente consiste en una serie de cambios de temperatura que se repiten entre 25 y 50 veces. Estos ciclos normalmente constan de tres etapas: la primera, alrededor de los 95 °C, permite la separación de la doble cadena del ácido nucleico; la segunda, a una temperatura de alrededor de 50-60 °C, permite la unión de los cebadores con el molde de ADN; [7] la tercera, entre 68 y 72 °C, facilita la polimerización llevada a cabo por la ADN polimerasa. Debido al pequeño tamaño de los fragmentos, el último paso suele omitirse en este tipo de PCR ya que la enzima es capaz de replicar el amplicón de ADN durante el cambio entre la etapa de alineamiento y la de desnaturalización. Además, en la PCR de cuatro pasos la fluorescencia se mide durante fases de temperatura cortas que duran solo unos segundos en cada ciclo, con una temperatura de, por ejemplo, 80 °C, con el fin de reducir la señal causada por la presencia de dímeros de cebadores cuando se utiliza un colorante no específico. [8] Las temperaturas y los tiempos utilizados para cada ciclo dependen de una amplia variedad de parámetros, tales como: la enzima utilizada para sintetizar el ADN, la concentración de iones divalentes y desoxirribonucleótidos trifosfatos (dNTP) en la reacción y la temperatura de enlace de los cebadores. [9]
La técnica de PCR en tiempo real se puede clasificar según la química utilizada para detectar el producto de PCR, fluorocromos específicos o no específicos.
Un colorante que se une al ADN se une a todo el ADN bicatenario (ds) en la PCR, lo que aumenta el rendimiento cuántico de fluorescencia del colorante. Por lo tanto, un aumento en el producto de ADN durante la PCR conduce a un aumento en la intensidad de fluorescencia medida en cada ciclo. Sin embargo, los colorantes de dsADN como SYBR Green se unirán a todos los productos de PCR de dsADN, incluidos los productos de PCR no específicos (como el dímero de cebador ). Esto puede interferir potencialmente con, o impedir, el monitoreo preciso de la secuencia objetivo deseada.
En la PCR en tiempo real con colorantes de dsADN, la reacción se prepara de la forma habitual, con la adición de un colorante fluorescente de dsADN. A continuación, la reacción se realiza en un instrumento de PCR en tiempo real y, después de cada ciclo, se mide la intensidad de la fluorescencia con un detector; el colorante solo emite fluorescencia cuando se une al dsADN (es decir, el producto de PCR). Este método tiene la ventaja de que solo se necesita un par de cebadores para realizar la amplificación, lo que reduce los costes; se pueden controlar múltiples secuencias diana en un tubo utilizando diferentes tipos de colorantes.

Las sondas fluorescentes de señalización detectan únicamente el ADN que contiene la secuencia complementaria a la sonda; por lo tanto, el uso de la sonda de señalización aumenta significativamente la especificidad y permite realizar la técnica incluso en presencia de otro ADN de doble cadena. Mediante el uso de etiquetas de diferentes colores, las sondas fluorescentes se pueden utilizar en ensayos multiplex para controlar varias secuencias diana en el mismo tubo. La especificidad de las sondas fluorescentes de señalización también evita la interferencia de las mediciones causada por los dímeros de cebadores , que son subproductos potenciales indeseables en la PCR. Sin embargo, las sondas fluorescentes de señalización no evitan el efecto inhibidor de los dímeros de cebadores, que pueden reducir la acumulación de los productos deseados en la reacción.
El método se basa en una sonda basada en ADN con un indicador fluorescente en un extremo y un inhibidor de fluorescencia en el extremo opuesto de la sonda. La proximidad del indicador al inhibidor impide la detección de su fluorescencia; la ruptura de la sonda por la actividad exonucleasa 5' a 3' de la polimerasa Taq rompe la proximidad entre el indicador y el inhibidor y, por lo tanto, permite la emisión ininterrumpida de fluorescencia, que se puede detectar después de la excitación con un láser. Por lo tanto, un aumento del producto al que se dirige la sonda del indicador en cada ciclo de PCR provoca un aumento proporcional de la fluorescencia debido a la ruptura de la sonda y la liberación del indicador.

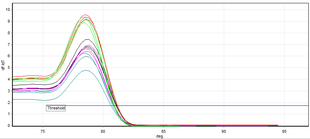
La PCR en tiempo real permite identificar fragmentos específicos de ADN amplificados mediante el análisis de su temperatura de fusión (también llamada valor Tm , de melting temperature ). El método utilizado suele ser la PCR con colorantes de unión al ADN bicatenario como reporteros y el colorante utilizado suele ser SYBR Green. La temperatura de fusión del ADN es específica del fragmento amplificado. Los resultados de esta técnica se obtienen comparando las curvas de disociación de las muestras de ADN analizadas. [11]
A diferencia de la PCR convencional, este método evita el uso previo de técnicas de electroforesis para demostrar los resultados de todas las muestras. Esto se debe a que, a pesar de ser una técnica cinética, la PCR cuantitativa suele evaluarse en un punto final distinto, por lo que la técnica suele proporcionar resultados más rápidos y/o utilizar menos reactivos que la electroforesis. Si se requiere una electroforesis posterior, solo es necesario analizar aquellas muestras que la PCR en tiempo real haya demostrado ser dudosas y/o ratificar los resultados de las muestras que hayan dado positivo para un determinado determinante.
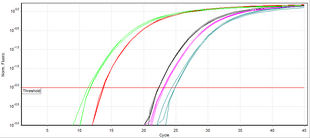
A diferencia de la PCR de punto final (PCR convencional), la PCR en tiempo real permite monitorear el producto deseado en cualquier punto del proceso de amplificación midiendo la fluorescencia (en el marco de tiempo real, se realiza la medición de su nivel sobre un umbral dado). Un método comúnmente empleado de cuantificación de ADN por PCR en tiempo real se basa en representar gráficamente la fluorescencia contra el número de ciclos en una escala logarítmica . Un umbral para la detección de fluorescencia basada en ADN se establece de 3 a 5 veces la desviación estándar del ruido de la señal por encima del fondo. El número de ciclos en los que la fluorescencia excede el umbral se denomina ciclo umbral (C t ) o, según las pautas de MIQE, ciclo de cuantificación (C q ) . [1]
Durante la fase de amplificación exponencial, la cantidad de la plantilla de ADN objetivo (amplicón) se duplica en cada ciclo. Por ejemplo, una muestra de ADN cuyo C q precede al de otra muestra en 3 ciclos contenía 2 3 = 8 veces más plantilla. Sin embargo, la eficiencia de la amplificación suele ser variable entre cebadores y plantillas. Por lo tanto, la eficiencia de una combinación de cebador-plantilla se evalúa en un experimento de titulación con diluciones en serie de plantilla de ADN para crear una curva estándar del cambio en (C q ) con cada dilución. Luego, la pendiente de la regresión lineal se utiliza para determinar la eficiencia de la amplificación, que es del 100% si una dilución de 1:2 da como resultado una diferencia de (C q ) de 1. El método del umbral de ciclo hace varias suposiciones del mecanismo de reacción y se basa en datos de regiones de baja relación señal-ruido del perfil de amplificación que pueden introducir una variación sustancial durante el análisis de datos. [12]
Para cuantificar la expresión génica, la (C q ) de un ARN o ADN del gen de interés se resta de la (C q ) de ARN/ADN de un gen de mantenimiento en la misma muestra para normalizar la variación en la cantidad y calidad del ARN entre diferentes muestras. Este procedimiento de normalización se denomina comúnmente método ΔC t [13] y permite comparar la expresión de un gen de interés entre diferentes muestras. Sin embargo, para dicha comparación, la expresión del gen de referencia normalizador debe ser muy similar en todas las muestras. Por lo tanto, la elección de un gen de referencia que cumpla este criterio es de gran importancia y, a menudo, un desafío, porque solo muy pocos genes muestran niveles iguales de expresión en una variedad de diferentes condiciones o tejidos. [14] [15] Aunque el análisis del umbral de ciclo está integrado con muchos sistemas de software comerciales, existen métodos más precisos y confiables para analizar los datos del perfil de amplificación que deben considerarse en los casos en que la reproducibilidad es una preocupación. [12]
También se han sugerido métodos de cuantificación de PCR cuantitativa basados en mecanismos, que tienen la ventaja de que no requieren una curva estándar para la cuantificación. Se ha demostrado que métodos como MAK2 [16] tienen un rendimiento cuantitativo igual o mejor que los métodos de curva estándar. Estos métodos basados en mecanismos utilizan el conocimiento sobre el proceso de amplificación de la polimerasa para generar estimaciones de la concentración de la muestra original. Una extensión de este enfoque incluye un modelo preciso de todo el perfil de la reacción de PCR, que permite el uso de datos de alta relación señal-ruido y la capacidad de validar la calidad de los datos antes del análisis. [12]
Según la investigación de Ruijter et al. [17], MAK2 asume una eficiencia de amplificación constante durante la reacción de PCR. Sin embargo, el análisis teórico de la reacción en cadena de la polimerasa, de la que se derivó MAK2, ha revelado que la eficiencia de amplificación no es constante durante toda la PCR. Si bien la cuantificación de MAK2 proporciona estimaciones confiables de la concentración de ADN objetivo en una muestra en condiciones normales de qPCR, MAK2 no cuantifica de manera confiable la concentración del objetivo para ensayos de qPCR con competímetros.
La reacción en cadena de la polimerasa cuantitativa tiene numerosas aplicaciones en el laboratorio . Se utiliza habitualmente tanto para diagnóstico como para investigación básica . Los usos de la técnica en la industria incluyen la cuantificación de la carga microbiana en alimentos o materia vegetal, la detección de OGM ( organismos modificados genéticamente ) y la cuantificación y genotipificación de patógenos virales humanos.
La cuantificación de la expresión génica mediante métodos tradicionales de detección de ADN no es fiable. La detección de ARNm en un Northern blot o de productos de PCR en un gel o Southern blot no permite una cuantificación precisa. [18] Por ejemplo, a lo largo de los 20 a 40 ciclos de una PCR típica, la cantidad de producto de ADN alcanza una meseta que no está directamente correlacionada con la cantidad de ADN objetivo en la PCR inicial. [19]
La PCR en tiempo real se puede utilizar para cuantificar ácidos nucleicos mediante dos métodos comunes: cuantificación relativa y cuantificación absoluta. [20] La cuantificación absoluta proporciona el número exacto de moléculas de ADN diana mediante comparación con estándares de ADN utilizando una curva de calibración . Por lo tanto, es esencial que la PCR de la muestra y el estándar tengan la misma eficiencia de amplificación . [21] La cuantificación relativa se basa en genes de referencia internos para determinar las diferencias de expresión del gen diana. La cuantificación se expresa como el cambio en los niveles de expresión del ARNm interpretado como ADN complementario (ADNc, generado por transcripción inversa del ARNm). La cuantificación relativa es más fácil de llevar a cabo, ya que no requiere una curva de calibración, ya que la cantidad del gen estudiado se compara con la cantidad de un gen de referencia de control.
Como las unidades utilizadas para expresar los resultados de la cuantificación relativa no son importantes, los resultados se pueden comparar entre varios RTqPCR diferentes. La razón para utilizar uno o más genes de mantenimiento es corregir la variación no específica, como las diferencias en la cantidad y calidad del ARN utilizado, que pueden afectar la eficiencia de la transcripción inversa y, por lo tanto, la de todo el proceso de PCR. Sin embargo, el aspecto más crucial del proceso es que el gen de referencia debe ser estable. [22]
La selección de estos genes de referencia se ha llevado a cabo tradicionalmente en biología molecular mediante estudios cualitativos o semicuantitativos como el examen visual de geles de ARN, la densitometría northern blot o la PCR semicuantitativa (PCR mimics). Ahora, en la era del genoma , es posible realizar una estimación más detallada para muchos organismos utilizando tecnologías transcriptómicas . [23] Sin embargo, la investigación ha demostrado que la amplificación de la mayoría de los genes de referencia utilizados para cuantificar la expresión del ARNm varía según las condiciones experimentales. [24] [25] [26] Por lo tanto, es necesario realizar un estudio metodológico inicial estadísticamente sólido para seleccionar el gen de referencia más adecuado.
Se han desarrollado varios algoritmos estadísticos que pueden detectar qué gen o genes son los más adecuados para su uso en determinadas condiciones. Algunos como geNORM o BestKeeper pueden comparar pares o medias geométricas de una matriz de diferentes genes y tejidos de referencia . [4] [6]
La PCR cualitativa diagnóstica se aplica para detectar rápidamente ácidos nucleicos que son diagnósticos de, por ejemplo, enfermedades infecciosas , [27] [28] cáncer y anomalías genéticas. La introducción de ensayos de PCR cualitativos en el laboratorio de microbiología clínica ha mejorado significativamente el diagnóstico de enfermedades infecciosas, [29] y se utiliza como una herramienta para detectar enfermedades emergentes, como nuevas cepas de gripe y coronavirus , [30] en pruebas de diagnóstico . [31] [32]
La PCR cuantitativa también es utilizada por microbiólogos que trabajan en los campos de la seguridad alimentaria, el deterioro y la fermentación de los alimentos y para la evaluación del riesgo microbiano de la calidad del agua (potable y recreativa) y en la protección de la salud pública. [33]
La qPCR también se puede utilizar para amplificar marcadores taxonómicos o funcionales de genes en el ADN tomado de muestras ambientales. [34] Los marcadores están representados por fragmentos genéticos de ADN o ADN complementario. [34] Al amplificar un determinado elemento genético, se puede cuantificar la cantidad del elemento en la muestra antes de la amplificación. [34] El uso de marcadores taxonómicos (genes ribosomales) y qPCR puede ayudar a determinar la cantidad de microorganismos en una muestra y puede identificar diferentes familias, géneros o especies según la especificidad del marcador. [34] El uso de marcadores funcionales (genes codificadores de proteínas) puede mostrar la expresión genética dentro de una comunidad, lo que puede revelar información sobre el medio ambiente. [34]
La industria agrícola se esfuerza constantemente por producir propágulos o plántulas de plantas libres de patógenos para evitar pérdidas económicas y salvaguardar la salud. Se han desarrollado sistemas que permiten detectar pequeñas cantidades de ADN de Phytophthora ramorum , un oomiceto que mata robles y otras especies, mezclado con el ADN de la planta huésped. La discriminación entre el ADN del patógeno y el de la planta se basa en la amplificación de secuencias ITS, espaciadores ubicados en la zona codificante del gen del ARN ribosómico , característicos de cada taxón. [35] También se han desarrollado versiones de campo de esta técnica para identificar el mismo patógeno. [36]
La qPCR con transcripción inversa (RT-qPCR) se puede utilizar para detectar OGM dada su sensibilidad y rango dinámico en la detección de ADN. Alternativas como el análisis de ADN o proteínas suelen ser menos sensibles. Se utilizan cebadores específicos que amplifican no el transgén sino el promotor , el terminador o incluso las secuencias intermedias utilizadas durante el proceso de ingeniería del vector. Como el proceso de creación de una planta transgénica normalmente conduce a la inserción de más de una copia del transgén, su cantidad también se evalúa comúnmente. Esto se lleva a cabo a menudo mediante cuantificación relativa utilizando un gen de control de la especie tratada que solo está presente como una única copia. [37] [38]
Los virus pueden estar presentes en humanos por infección directa o coinfecciones lo que dificulta el diagnóstico mediante técnicas clásicas y puede dar lugar a un pronóstico y tratamiento incorrectos. El uso de qPCR permite tanto la cuantificación como el genotipado (caracterización de la cepa, realizada mediante curvas de melting) de un virus como el de la hepatitis B. [39] El grado de infección, cuantificado como las copias del genoma viral por unidad de tejido del paciente, es relevante en muchos casos; por ejemplo, la probabilidad de que el virus del herpes simple tipo 1 se reactive está relacionada con el número de neuronas infectadas en los ganglios . [40] Esta cuantificación se realiza bien con transcripción inversa o bien sin ella, como ocurre si el virus llega a integrarse en el genoma humano en cualquier punto de su ciclo, como sucede en el caso del VPH (virus del papiloma humano), donde algunas de sus variantes se asocian a la aparición de cáncer de cuello uterino . [41] La PCR en tiempo real también ha permitido cuantificar el citomegalovirus humano (CMV), que se observa en pacientes inmunodeprimidos tras un trasplante de órgano sólido o de médula ósea. [42]
{{cite journal}}: CS1 maint: varios nombres: lista de autores ( enlace )