El virus del enanismo arbustivo del tomate ( TBSV ) es un virus de la familia de los tombusvirus . [2] Se informó por primera vez en tomates en 1935 y afecta principalmente a los cultivos de hortalizas, aunque generalmente no se considera un patógeno vegetal económicamente significativo. Dependiendo del huésped, el TBSV causa retraso en el crecimiento, moteado de las hojas y frutos deformados o ausentes. Es probable que el virus se transmita por el suelo en el entorno natural, pero también puede transmitirse mecánicamente, por ejemplo, a través de herramientas de corte contaminadas. El TBSV se ha utilizado como un sistema modelo en la investigación virológica sobre el ciclo de vida de los virus de las plantas , particularmente en infecciones experimentales de la planta huésped modelo Nicotiana benthamiana . [3] [4]

El TBSV tiene un amplio rango de hospedadores en condiciones experimentales y se ha informado que infecta a más de 120 especies de plantas que abarcan 20 familias. Sin embargo, en condiciones naturales su rango es mucho más estrecho y generalmente comprende cultivos de hortalizas y plantas ornamentales. Se identificó por primera vez en plantas de tomate y también se ha documentado que afecta a la manzana , la alcachofa , la cereza , la vid , el lúpulo y la pimienta . Aunque causa una pérdida significativa de rendimiento en las plantas de tomate, no se considera un patógeno económicamente significativo. [4] [5] Sin embargo, es un sistema modelo muy bien establecido para el estudio de virus de plantas, generalmente a través de la infección experimental de Nicotiana benthamiana o Nicotiana clevelandii , parientes de las plantas de tabaco en las que el TBSV puede causar una infección sistémica. Cabe destacar que la planta modelo común Arabidopsis thaliana no es un hospedador. [3] [4] El TBSV también puede replicarse en levadura en condiciones de laboratorio. [6]
Los síntomas del TBSV dependen del hospedador. Las infecciones locales pueden causar lesiones necróticas o cloróticas . Las infecciones sistémicas pueden causar retraso en el crecimiento, frutos deformados o ausentes y hojas dañadas; en entornos agrícolas, el rendimiento puede verse significativamente reducido. El aspecto atrofiado y "arbustivo" de las plantas de tomate en las que se descubrió el virus por primera vez le dio al patógeno su nombre. En algunos hospedadores, sobre todo N. benthamiana , el TBSV puede causar necrosis sistémica letal. [4] [5]
Se cree que el TBSV se transmite pasivamente en la naturaleza, principalmente a través del suelo o el agua. No se conocen organismos vectores ; se ha descartado específicamente la transmisión por pulgones , ácaros y el hongo Olpidium brassicae . [5] Sin embargo, se ha observado que el virus de la necrosis del pepino (CNV), estrechamente relacionado con el tombusvirus, se transmite por zoosporas de Olpidium bornovanus , por lo que la transmisión del TBSV por un vector aún desconocido sigue siendo una posibilidad. [4] El TBSV también se puede transmitir a través de semillas o por inoculación mecánica. [4] [5] En pruebas experimentales, el virus puede sobrevivir al paso por el sistema digestivo humano si se consume en los alimentos y seguirá siendo infeccioso; se ha planteado la hipótesis de que podría propagarse a través de las aguas residuales . [7]
El TBSV se distribuye de forma bastante amplia en Europa central y occidental, el norte de África y América del Norte y del Sur. [4] [5] No se recomiendan medidas de control específicas para el virus, aunque las pautas de manejo de plagas distribuidas por la Universidad de California recomiendan evitar campos con antecedentes de TBSV o utilizar rotaciones de cultivos prolongadas . [8]
El TBSV pertenece al género Tombusvirus de la familia Tombusviridae . [9] Tanto el género como la familia derivan sus nombres de una abreviatura de "virus del enanismo arbustivo del tomate". [10]

El TBSV es un virus icosaédrico sin envoltura con una cápside viral T=3 compuesta por 180 subunidades de una única proteína de la cápside . Su estructura fue estudiada extensamente mediante cristalografía de rayos X desde finales de la década de 1950; su simetría icosaédrica fue identificada por primera vez por el biólogo estructural Donald Caspar , quien también fue pionero en el estudio del virus del mosaico del tabaco . [11] Un mapa de resolución casi atómica fue obtenido en 1978 por un equipo de investigación que incluía a Stephen C. Harrison . [12] [13]

El TBSV es un virus de ARN monocatenario de sentido positivo con un genoma lineal de ~4800 nucleótidos . [14] [15] El genoma contiene cinco genes que codifican una replicasa compuesta por dos proteínas (p33 y p92), una proteína de la cápside (llamada CP o p41) y dos proteínas adicionales, el supresor del silenciamiento del ARN p19 y la proteína de movimiento p22. [4] Estas dos proteínas se expresan a partir de genes superpuestos dispuestos de modo que el marco de lectura abierto de p19 esté completamente dentro del ORF de p22. [16] El genoma contiene un posible gen adicional, llamado pX, de función desconocida. [4]
Juntos, p33 y p92 forman el complejo de replicasa viral. P33 es más pequeño y p92 se produce a través de la lectura ribosómica del codón de terminación p33 , lo que da como resultado una secuencia de aminoácidos N-terminal compartida y un gran exceso de p33 en relación con p92. Las proteínas p33 se unen cooperativamente a los ácidos nucleicos monocatenarios, mientras que la proteína p92 es una ARN polimerasa dependiente de ARN (RdRp). Ambas son esenciales para la proliferación viral. Ambas proteínas están asociadas con las membranas celulares . [4]
La proteína de la cápside viral CP, o p41, es una proteína de doble capa que se ensambla en una cápside icosaédrica que contiene 180 copias de la proteína. La formación de viriones no siempre es necesaria para la propagación localizada del virus en las células vegetales vecinas, porque las partículas de ribonucleoproteína que contienen material genético viral pueden propagarse a las células vecinas inmediatas a través de los plasmodesmos . Sin embargo, la proteína de la cápside es necesaria para la infección sistémica. [4]
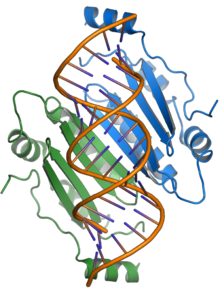
La proteína p19 es un factor de patogenicidad y funciona suprimiendo la vía de silenciamiento del ARN , una forma común de defensa antiviral. La proteína p19 se une a los ARN interferentes cortos y evita su incorporación al complejo de silenciamiento inducido por ARN (RISC), lo que permite la propagación viral en la planta huésped. [3] [17] [18] La presencia de p19 es necesaria para la infección sistémica o para la infección letal en algunos huéspedes; en el huésped experimental N. benthamiana , p19 media en gran medida la necrosis sistémica letal que es el resultado de la infección por TBSV. [4] [17]
La proteína p22 es una proteína de movimiento necesaria para que el virus se propague de una célula a otra. La p22 es una proteína de unión al ARN que está asociada con la pared celular y facilita el movimiento del material genético viral de una célula a su vecina a través de plasmodesmos interconectados . [4]
Un virión de TBSV contiene una copia de su genoma de ARN monocatenario de sentido positivo , que es lineal y carece de una cola de poliadenina 3' o una tapa 5' . Sin embargo, las proteínas p33 y p92 se traducen directamente a partir del ARN genómico. Cuando se replica el genoma, se producen dos moléculas de ARN subgenómico que actúan como ARN mensajero ; una a partir de la cual se expresa el gen p41 (CP), y otra a partir de los genes p19 y p22. Los genes p19 y p22 superpuestos se traducen a través de los efectos del escaneo con fugas . [4] Se han identificado varias interacciones de larga distancia entre áreas linealmente bien separadas del genoma con importancia funcional para garantizar una replicación eficiente. [16]
Las moléculas de ARN interferente defectuoso (ID) son ARN que se producen a partir del genoma viral pero que no son capaces de infectar células por sí solos; en cambio, requieren una coinfección con un virus "auxiliar" intacto. Las infecciones por TBSV a menudo producen cantidades significativas de ID a partir de partes consistentes del genoma en condiciones experimentales, pero este comportamiento no se ha observado en la naturaleza. Es probable que su producción sea específica del hospedador. Las infecciones que dan lugar a ID suelen tener signos más leves. [4] [19]