El supresor del silenciamiento de ARN p19 (también conocido como proteína central P19 de Tombusvirus y modulador de la gravedad de los síntomas de 19 kDa ) es una proteína expresada a partir del gen ORF4 en el genoma de los tombusvirus . Estos virus son virus de ARN monocatenario de sentido positivo que infectan células vegetales , en las que el silenciamiento del ARN forma un sistema de defensa antiviral generalizado y robusto. La proteína p19 sirve como estrategia de contradefensa, uniéndose específicamente a los ARN bicatenarios de 19 a 21 nucleótidos que funcionan como ARN interferente pequeño (siRNA) en el sistema de silenciamiento del ARN. Al secuestrar el siRNA, p19 suprime el silenciamiento del ARN y promueve la proliferación viral. [1] [2] [3] La proteína p19 se considera un factor de virulencia significativo [4] y un componente de una carrera armamentista evolutiva entre las plantas y sus patógenos . [5]
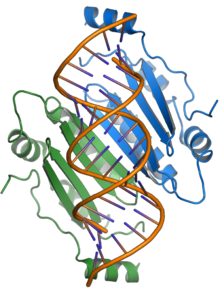
La proteína p19 recibió su nombre por su tamaño, siendo aproximadamente 19 kilodaltons . Forma un homodímero funcional . Las estructuras cristalinas de las proteínas p19 están disponibles en el virus del enanismo arbustivo del tomate [1] y el virus de la mancha anular italiana del clavel ; [2] la proteína consiste en un nuevo pliegue proteico y ejemplifica un mecanismo previamente desconocido para la unión del ARN , utilizando una superficie de unión formada por una lámina beta y flanqueada por hélices alfa para interactuar con ARN bicatenarios de alrededor de 21 nucleótidos de longitud de una manera no específica de secuencia. [1] [2] [7]
La proteína p19 se une a los ARN bicatenarios que funcionan como ARN interferente corto (siRNA) y está especializada para el producto de 21 nucleótidos de la enzima DCL4 (un miembro de una familia de enzimas vegetales con homología con Dicer ). [7] Al unirse al siRNA, p19 secuestra estas especies y evita que interactúen con el complejo de silenciamiento inducido por ARN (RISC), un complejo proteico que media el mecanismo de silenciamiento del ARN antiviral en la célula.
La proteína p19 también es capaz de unirse a moléculas de microARN que son endógenas a la célula huésped, así como a los siRNA que en última instancia se derivan del propio genoma del virus. Cabe destacar que una excepción a este patrón es la ineficiencia de p19 en la interacción con el microARN miR-168, un ARN regulador no codificante que reprime la expresión de argonaute -1 (AGO1). La proteína AGO1 es necesaria para el silenciamiento del ARN, por lo que evitar selectivamente que su represor secuestre el miARN de forma general por parte de p19 tiene el efecto de reducir los niveles celulares de AGO1 y es un mecanismo adicional por el cual p19 inhibe el silenciamiento. [5] [8] Los dos mecanismos son independientes entre sí y pueden ser anulados selectivamente por mutaciones . [9]
El gen que codifica la proteína p19 es un ejemplo de un gen sobreimpreso , una disposición genómica común en los virus en la que múltiples genes están codificados por la misma porción del genoma leído en marcos de lectura alternativos . [10] [11] El marco de lectura abierto ORF4, que codifica p19, está completamente contenido dentro del marco de lectura abierto de otro gen, que se designa ORF3 y codifica la proteína de movimiento p22. Ambos genes, y sus posiciones relativas, se conservan dentro de la familia tombusvirus. [4] [11] Se cree que p19 se originó de novo en este linaje. [11] [12]
El secuestro de dsRNA es una estrategia común de contradefensa viral contra el silenciamiento de ARN, que evolucionó en una forma de carrera armamentista evolutiva entre el virus y el huésped. [5] La proteína p19 no es la única en esta función; en un ejemplo de evolución convergente , esta estrategia parece haber evolucionado al menos tres veces en distintos linajes virales utilizando proteínas con distintas estructuras y medios físicos de unión al ARN. [3] [13] [14]
El virus del enanismo arbustivo del tomate , que es la especie tipo de la familia de los tombusvirus , es un sistema modelo de larga data para el estudio de los virus de plantas . El marco de lectura abierto que codifica p19 se descubrió originalmente a fines de la década de 1980 cuando se secuenció el genoma del virus; posteriormente se demostró que la proteína predicha se expresaba de hecho a partir del gen, aunque inicialmente se subestimó su papel en la promoción de la virulencia y la infectividad. Tras la elucidación de su papel como supresor del silenciamiento del ARN, p19 también se ha utilizado como una herramienta en la investigación de biología molecular sobre el silenciamiento del ARN, la interferencia del ARN y procesos relacionados. [4] [6]