El operón lactosa ( operón lac ) es un operón necesario para el transporte y metabolismo de la lactosa en E. coli y muchas otras bacterias entéricas . Aunque la glucosa es la fuente de carbono preferida por la mayoría de las bacterias entéricas, el operón lac permite la digestión eficaz de la lactosa cuando la glucosa no está disponible a través de la actividad de la β-galactosidasa . [1] La regulación genética del operón lac fue el primer mecanismo de regulación genética que se entendió claramente, por lo que se ha convertido en un ejemplo destacado de regulación genética procariota . A menudo se discute en clases introductorias de biología molecular y celular por esta razón. Este sistema de metabolismo de la lactosa fue utilizado por François Jacob y Jacques Monod para determinar cómo una célula biológica sabe qué enzima sintetizar. Su trabajo sobre el operón lac les valió el Premio Nobel de Fisiología en 1965. [1]
La mayoría de las células bacterianas, incluida la E. coli, carecen de intrones en su genoma. También carecen de membrana nuclear . Por lo tanto, la regulación génica por el operón lac ocurre a nivel transcripcional, al impedir la conversión de ADN en ARNm .
Los operones bacterianos son transcripciones policistrónicas que pueden producir múltiples proteínas a partir de una transcripción de ARNm . En este caso, cuando se requiere lactosa como fuente de azúcar para la bacteria, se pueden expresar los tres genes del operón lac y traducir sus proteínas posteriores: lacZ , lacY y lacA . El producto génico de lacZ es la β-galactosidasa que escinde la lactosa, un disacárido , en glucosa y galactosa . lacY codifica la β-galactosido permeasa , una proteína de membrana que se incrusta en la membrana plasmática para permitir el transporte celular de lactosa al interior de la célula. Finalmente, lacA codifica la β-galactosido transacetilasa .

Tenga en cuenta que la cantidad de pares de bases que se muestra en el diagrama anterior no se utiliza para calcular la escala. De hecho, hay más de 5300 pares de bases en el operón lac . [2]
Sería un desperdicio producir enzimas cuando no hay lactosa disponible o si se dispone de una fuente de energía preferible, como la glucosa. El operón lac utiliza un mecanismo de control de dos partes para garantizar que la célula gaste energía produciendo las enzimas codificadas por el operón lac solo cuando sea necesario. [3]
En ausencia de lactosa, el represor lac , codificado por lacI, detiene la producción de las enzimas y proteínas de transporte codificadas por el operón lac . [4] Lo hace bloqueando la ARN polimerasa dependiente de ADN . Este bloqueo/detención no es perfecto y se produce una cantidad mínima de expresión génica todo el tiempo. La proteína represora siempre se expresa, pero el operón lac (enzimas y proteínas de transporte) se reprime (pero no se detiene por completo).
Cuando hay lactosa disponible pero no glucosa, una parte de lactosa ingresa a la célula mediante la proteína de transporte preexistente codificada por lacY. Esta lactosa se combina con el represor y lo inactiva, lo que permite que se exprese el operón lac . Luego se sintetiza más permeasa β-galactosida , lo que permite que ingrese aún más lactosa y las enzimas codificadas por lacZ y lacA puedan digerirla.
Sin embargo, en presencia de glucosa, independientemente de la presencia de lactosa, el operón se verá reprimido. Esto se debe a que la proteína activadora de catabolitos (CAP), necesaria para la producción de las enzimas, permanece inactiva, y EIIA Glc desactiva la permeasa de lactosa para evitar el transporte de lactosa al interior de la célula. Este mecanismo de control dual provoca la utilización secuencial de glucosa y lactosa en dos fases de crecimiento distintas, conocidas como diauxia .

Sólo lacZ y lacY parecen ser necesarias para la vía catabólica de la lactosa .
En números, lacI tiene 1100 bps, lacZ tiene 3000 bps, lacY tiene 800 bps, lacA tiene 800 bps, con 3 bps correspondientes a 1 aminoácido. [5]
Se utilizan abreviaturas de tres letras para describir fenotipos en bacterias, incluida E. coli .
Algunos ejemplos incluyen:
En el caso de Lac, las células de tipo salvaje son Lac + y pueden utilizar la lactosa como fuente de carbono y energía, mientras que los derivados mutantes Lac− no pueden utilizar lactosa. Normalmente se utilizan las mismas tres letras (en minúsculas y cursiva) para etiquetar los genes implicados en un fenotipo particular, donde cada gen diferente se distingue además por una letra extra. Los genes lac que codifican enzimas son lacZ , lacY y lacA . El cuarto gen lac es lacI , que codifica el represor de la lactosa; la "I" significa inducibilidad .
Se puede distinguir entre genes estructurales que codifican enzimas y genes reguladores que codifican proteínas que afectan la expresión génica. El uso actual amplía la nomenclatura fenotípica para aplicarla a las proteínas: así, LacZ es el producto proteico del gen lacZ , β-galactosidasa. Varias secuencias cortas que no son genes también afectan la expresión génica, incluido el promotor lac , lac p , y el operador lac , lac o . Aunque no es un uso estrictamente estándar, las mutaciones que afectan a lac o se denominan lac o c , por razones históricas.
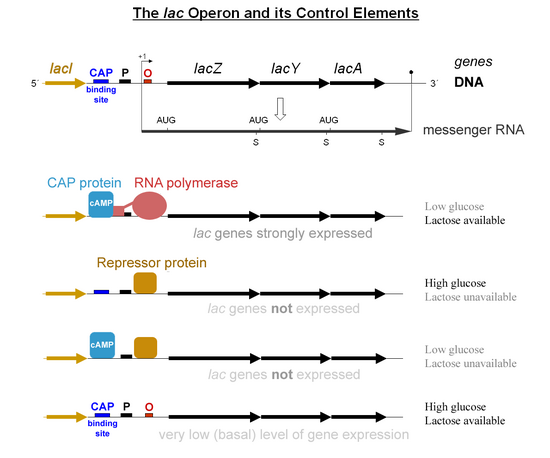
El control específico de los genes lac depende de la disponibilidad del sustrato lactosa para la bacteria. Las proteínas no son producidas por la bacteria cuando la lactosa no está disponible como fuente de carbono. Los genes lac están organizados en un operón ; es decir, están orientados en la misma dirección inmediatamente adyacente en el cromosoma y se co-transcriben en una única molécula de ARNm policistrónico . La transcripción de todos los genes comienza con la unión de la enzima ARN polimerasa (RNAP), una proteína de unión al ADN , que se une a un sitio de unión específico del ADN, el promotor , inmediatamente aguas arriba de los genes. La unión de la ARN polimerasa al promotor es asistida por la proteína activadora del catabolito unida a AMPc (CAP, también conocida como proteína receptora de AMPc). [6] Sin embargo, el gen lacI (gen regulador del operón lac ) produce una proteína que impide que la ARNP se una al operador del operón. Esta proteína solo puede eliminarse cuando la alolactosa se une a ella y la inactiva. La proteína que se forma a partir del gen lacI se conoce como represor lac. El tipo de regulación que sufre el operón lac se denomina inducible negativa, lo que significa que el gen se desactiva por el factor regulador ( represor lac ) a menos que se añada alguna molécula (lactosa). Una vez que se elimina el represor, la ARN polimerasa procede a transcribir los tres genes ( lacZYA ) en ARNm. Cada uno de los tres genes de la cadena de ARNm tiene su propia secuencia Shine-Dalgarno , por lo que los genes se traducen de forma independiente. [7] La secuencia de ADN del operón lac de E. coli , el ARNm lacZYA y los genes lacI están disponibles en GenBank (ver).
El primer mecanismo de control es la respuesta reguladora a la lactosa, que utiliza una proteína reguladora intracelular llamada represor de lactosa para impedir la producción de β-galactosidasa en ausencia de lactosa. El gen lacI que codifica el represor se encuentra cerca del operón lac y siempre se expresa ( constitutivo ). Si falta lactosa en el medio de crecimiento, el represor se une muy fuertemente a una secuencia corta de ADN justo aguas abajo del promotor cerca del comienzo de lacZ llamado operador lac . La unión del represor al operador interfiere con la unión de la ARN polimerasa al promotor y, por lo tanto, el ARNm que codifica LacZ y LacY solo se produce en niveles muy bajos. Sin embargo, cuando las células se cultivan en presencia de lactosa, un metabolito de la lactosa llamado alolactosa, producido a partir de la lactosa por el producto del gen lacZ , se une al represor, lo que provoca un cambio alostérico. De esta manera alterado, el represor no puede unirse al operador, lo que permite que la ARN polimerasa transcriba los genes lac y, de ese modo, genere niveles más altos de las proteínas codificadas.
El segundo mecanismo de control es una respuesta a la glucosa, que utiliza el homodímero de la proteína activadora de catabolitos (CAP) para aumentar en gran medida la producción de β-galactosidasa en ausencia de glucosa. El monofosfato de adenosina cíclico (cAMP) es una molécula señal cuya prevalencia es inversamente proporcional a la de la glucosa. Se une al CAP, lo que a su vez permite que el CAP se una al sitio de unión del CAP (una secuencia de ADN de 16 pb aguas arriba del promotor a la izquierda en el diagrama siguiente, aproximadamente 60 pb aguas arriba del sitio de inicio de la transcripción), [8] lo que ayuda a la ARN polimerasa a unirse al ADN. En ausencia de glucosa, la concentración de cAMP es alta y la unión de CAP-cAMP al ADN aumenta significativamente la producción de β-galactosidasa, lo que permite que la célula hidrolice la lactosa y libere galactosa y glucosa.
Más recientemente, se ha demostrado que la exclusión del inductor bloquea la expresión del operón lac cuando hay glucosa presente. La glucosa es transportada a la célula por el sistema de fosfotransferasa dependiente de PEP . El grupo fosfato del fosfoenolpiruvato se transfiere a través de una cascada de fosforilación que consiste en las proteínas generales PTS (sistema de fosfotransferasa) HPr y EIA y las proteínas PTS específicas de glucosa EIIA Glc y EIIB Glc , el dominio citoplasmático del transportador de glucosa EII. El transporte de glucosa está acompañado por su fosforilación por EIIB Glc , drenando el grupo fosfato de las otras proteínas PTS, incluida EIIA Glc . La forma no fosforilada de EIIA Glc se une a la permeasa lac y evita que lleve lactosa a la célula. Por lo tanto, si están presentes tanto la glucosa como la lactosa, el transporte de glucosa bloquea el transporte del inductor del operón lac . [9]

El represor lac es una proteína de cuatro partes, un tetrámero, con subunidades idénticas. Cada subunidad contiene un motivo hélice-vuelta-hélice (HTH) capaz de unirse al ADN. El sitio del operador donde se une el represor es una secuencia de ADN con simetría de repetición invertida. Los dos semisitios de ADN del operador se unen juntos a dos de las subunidades del represor. Aunque las otras dos subunidades del represor no hacen nada en este modelo, esta propiedad no se entendió durante muchos años.
Finalmente se descubrió que dos operadores adicionales están involucrados en la regulación de lac . [10] Uno (O 3 ) se encuentra aproximadamente −90 pb aguas arriba de O 1 en el extremo del gen lacI , y el otro (O 2 ) está aproximadamente +410 pb aguas abajo de O 1 en la parte temprana de lacZ . Estos dos sitios no se encontraron en el trabajo inicial porque tienen funciones redundantes y las mutaciones individuales no afectan mucho a la represión. Las mutaciones simples en O 2 o O 3 tienen solo efectos de 2 a 3 veces. Sin embargo, su importancia se demuestra por el hecho de que un mutante doble defectuoso tanto en O 2 como en O 3 es desreprimido drásticamente (aproximadamente 70 veces).
En el modelo actual, el represor lac está unido simultáneamente tanto al operador principal O 1 como a O 2 u O 3 . El ADN intermedio se enlaza desde el complejo. La naturaleza redundante de los dos operadores menores sugiere que no es un complejo enlazado específico lo que es importante. Una idea es que el sistema funciona a través de la unión; si el represor unido se libera de O 1 momentáneamente, la unión a un operador menor lo mantiene en la proximidad, de modo que puede volver a unirse rápidamente. Esto aumentaría la afinidad del represor por O 1 .

El represor es una proteína alostérica , es decir, puede asumir una de dos formas ligeramente diferentes, que están en equilibrio entre sí. En una forma, el represor se unirá al ADN operador con alta especificidad, y en la otra forma ha perdido su especificidad. Según el modelo clásico de inducción, la unión del inductor, ya sea alolactosa o IPTG, al represor afecta la distribución del represor entre las dos formas. Por lo tanto, el represor con el inductor unido se estabiliza en la conformación sin unión al ADN. Sin embargo, este modelo simple no puede ser la historia completa, porque el represor está unido de manera bastante estable al ADN, pero se libera rápidamente mediante la adición de inductor. Por lo tanto, parece claro que un inductor también puede unirse al represor cuando el represor ya está unido al ADN. Todavía no se sabe por completo cuál es el mecanismo exacto de unión. [11]
La unión no específica del represor al ADN desempeña un papel crucial en la represión e inducción del operón Lac. El sitio de unión específico para la proteína represora Lac es el operador. La interacción no específica está mediada principalmente por interacciones carga-carga, mientras que la unión al operador se refuerza por interacciones hidrofóbicas. Además, existe una gran cantidad de secuencias de ADN no específicas a las que se puede unir el represor. Esencialmente, cualquier secuencia que no sea el operador se considera no específica. Los estudios han demostrado que sin la presencia de una unión no específica, la inducción (o no represión) del operón Lac no podría ocurrir incluso con niveles saturados de inductor. Se ha demostrado que, sin la unión no específica, el nivel basal de inducción es diez mil veces menor que el observado normalmente. Esto se debe a que el ADN no específico actúa como una especie de "sumidero" para las proteínas represoras, distrayéndolas del operador. Las secuencias no específicas disminuyen la cantidad de represor disponible en la célula. Esto a su vez reduce la cantidad de inductor necesario para desreprimir el sistema. [12]

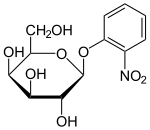


Se han descrito varios derivados o análogos de la lactosa que son útiles para trabajar con el operón lac . Estos compuestos son principalmente galactósidos sustituidos, en los que la fracción de glucosa de la lactosa se reemplaza por otro grupo químico.
El microorganismo experimental utilizado por François Jacob y Jacques Monod fue la bacteria común de laboratorio, E. coli , pero muchos de los conceptos reguladores básicos que fueron descubiertos por Jacob y Monod son fundamentales para la regulación celular en todos los organismos. [17] La idea clave es que las proteínas no se sintetizan cuando no son necesarias: E. coli conserva los recursos celulares y la energía al no producir las tres proteínas Lac cuando no hay necesidad de metabolizar la lactosa, como cuando otros azúcares como la glucosa están disponibles. La siguiente sección analiza cómo E. coli controla ciertos genes en respuesta a las necesidades metabólicas.
Durante la Segunda Guerra Mundial , Monod estaba probando los efectos de las combinaciones de azúcares como fuentes de nutrientes para E. coli y B. subtilis . Monod estaba siguiendo estudios similares que habían sido realizados por otros científicos con bacterias y levaduras. Descubrió que las bacterias cultivadas con dos azúcares diferentes a menudo mostraban dos fases de crecimiento. Por ejemplo, si se proporcionaban glucosa y lactosa, la glucosa se metabolizaba primero (fase de crecimiento I, ver Figura 2) y luego la lactosa (fase de crecimiento II). La lactosa no se metabolizaba durante la primera parte de la curva de crecimiento diaúxico porque la β-galactosidasa no se producía cuando tanto la glucosa como la lactosa estaban presentes en el medio. Monod llamó a este fenómeno diauxie . [18]

Luego, Monod centró su atención en la inducción de la formación de β-galactosidasa que se producía cuando la lactosa era el único azúcar en el medio de cultivo. [19]
Un gran avance conceptual de Jacob y Monod [20] fue reconocer la distinción entre sustancias reguladoras y sitios donde actúan para cambiar la expresión genética. Jacob, un ex soldado, utilizó la analogía de un bombardero que liberaría su carga letal al recibir una transmisión o señal de radio especial. Un sistema que funcione requiere tanto un transmisor terrestre como un receptor en el avión. Ahora, supongamos que el transmisor habitual está roto. Este sistema puede ponerse en funcionamiento mediante la introducción de un segundo transmisor funcional. En cambio, dijo, consideremos un bombardero con un receptor defectuoso. El comportamiento de este bombardero no puede cambiarse mediante la introducción de un segundo avión funcional.
Para analizar mutantes reguladores del operón lac , Jacob desarrolló un sistema mediante el cual una segunda copia de los genes lac ( lacI con su promotor, y lacZYA con promotor y operador) podría introducirse en una sola célula. A continuación, se prueba un cultivo de dichas bacterias, que son diploides para los genes lac pero por lo demás normales, para determinar el fenotipo regulador. En particular, se determina si LacZ y LacY se producen incluso en ausencia de IPTG (debido a que el represor de la lactosa producido por el gen mutante no es funcional). Este experimento, en el que se prueban genes o grupos de genes por pares, se denomina prueba de complementación .

Esta prueba se ilustra en la figura ( lacA se omite para simplificar). Primero, se muestran ciertos estados haploides (es decir, la célula lleva solo una copia de los genes lac ). El panel (a) muestra la represión, (b) muestra la inducción por IPTG, y (c) y (d) muestran el efecto de una mutación en el gen lacI o en el operador, respectivamente. En el panel (e) se muestra la prueba de complementación para el represor. Si una copia de los genes lac lleva una mutación en lacI , pero la segunda copia es de tipo salvaje para lacI , el fenotipo resultante es normal, pero lacZ se expresa cuando se expone al inductor IPTG. Se dice que las mutaciones que afectan al represor son recesivas al tipo salvaje (y ese tipo salvaje es dominante ), y esto se explica por el hecho de que el represor es una proteína pequeña que puede difundirse en la célula. La copia del operón lac adyacente al gen lacI defectuoso es efectivamente apagada por la proteína producida a partir de la segunda copia de lacI .
Si se lleva a cabo el mismo experimento utilizando una mutación operadora, se obtiene un resultado diferente (panel (f)). El fenotipo de una célula que porta un sitio operador mutante y uno de tipo salvaje es que LacZ y LacY se producen incluso en ausencia del inductor IPTG; porque el sitio operador dañado no permite la unión del represor para inhibir la transcripción de los genes estructurales. La mutación operadora es dominante. Cuando el sitio operador donde debe unirse el represor está dañado por la mutación, la presencia de un segundo sitio funcional en la misma célula no hace ninguna diferencia en la expresión de los genes controlados por el sitio mutante.
Una versión más sofisticada de este experimento utiliza operones marcados para distinguir entre las dos copias de los genes lac y mostrar que el o los genes estructurales no regulados son los que están al lado del operador mutante (panel (g). Por ejemplo, supongamos que una copia está marcada por una mutación que inactiva lacZ de modo que solo puede producir la proteína LacY, mientras que la segunda copia lleva una mutación que afecta a lacY y solo puede producir LacZ. En esta versión, solo la copia del operón lac que está adyacente al operador mutante se expresa sin IPTG. Decimos que la mutación del operador es cis-dominante , es dominante al tipo salvaje pero afecta solo a la copia del operón que está inmediatamente adyacente a ella.
Esta explicación es engañosa en un sentido importante, porque parte de una descripción del experimento y luego explica los resultados en términos de un modelo. Pero, de hecho, a menudo es cierto que el modelo viene primero y que se diseña un experimento específicamente para ponerlo a prueba. Jacob y Monod imaginaron primero que debía haber un sitio en el ADN con las propiedades del operador y luego diseñaron sus pruebas de complementación para demostrarlo.
El predominio de mutantes operadores también sugiere un procedimiento para seleccionarlos específicamente. Si se seleccionan mutantes reguladores de un cultivo de tipo salvaje utilizando fenil-Gal, como se describió anteriormente, las mutaciones del operador son raras en comparación con los mutantes represores porque el tamaño del objetivo es muy pequeño. Pero si en cambio comenzamos con una cepa que lleva dos copias de toda la región lac (que es diploide para lac ), las mutaciones del represor (que todavía ocurren) no se recuperan porque la complementación por el segundo gen lacI de tipo salvaje confiere un fenotipo de tipo salvaje. En contraste, la mutación de una copia del operador confiere un fenotipo mutante porque es dominante para la segunda copia de tipo salvaje.
La explicación de la diauxie dependía de la caracterización de mutaciones adicionales que afectaban a los genes lac además de las explicadas por el modelo clásico. Posteriormente se identificaron otros dos genes, cya y crp , que se mapeaban lejos de lac y que, cuando mutaban, resultaban en un nivel disminuido de expresión en presencia de IPTG e incluso en cepas de la bacteria que carecían del represor u operador. El descubrimiento de cAMP en E. coli condujo a la demostración de que los mutantes defectuosos en el gen cya pero no en el gen crp podían ser restaurados a la actividad completa mediante la adición de cAMP al medio.
El gen cya codifica la adenilato ciclasa, que produce AMPc. En un mutante cya , la ausencia de AMPc hace que la expresión de los genes lacZYA sea más de diez veces menor de lo normal. La adición de AMPc corrige la baja expresión de Lac característica de los mutantes cya . El segundo gen, crp , codifica una proteína llamada proteína activadora de catabolitos (CAP) o proteína receptora de AMPc (CRP). [22]
Sin embargo, las enzimas que metabolizan la lactosa se producen en pequeñas cantidades en presencia de glucosa y lactosa (a veces denominada expresión permeable) debido al hecho de que la ARN polimerasa todavía puede unirse a veces e iniciar la transcripción incluso en ausencia de CAP. La expresión permeable es necesaria para permitir el metabolismo de parte de la lactosa después de que se agote la fuente de glucosa, pero antes de que se active por completo la expresión de lac .
En resumen:
El retraso entre las fases de crecimiento refleja el tiempo necesario para producir cantidades suficientes de enzimas metabolizadoras de lactosa. Primero, la proteína reguladora CAP tiene que ensamblarse en el promotor lac , lo que resulta en un aumento en la producción de ARNm lac . Más copias disponibles del ARNm lac resultan en la producción (ver traducción ) de significativamente más copias de LacZ (β-galactosidasa, para el metabolismo de la lactosa) y LacY (permeasa de la lactosa para transportar la lactosa a la célula). Después de un retraso necesario para aumentar el nivel de las enzimas metabolizadoras de la lactosa, las bacterias entran en una nueva fase rápida de crecimiento celular .
Dos enigmas de la represión catabólica se relacionan con la forma en que los niveles de AMPc se acoplan a la presencia de glucosa y, en segundo lugar, por qué las células deberían siquiera molestarse en hacerlo. Después de que la lactosa se escinde, en realidad forma glucosa y galactosa (fácilmente convertida en glucosa). En términos metabólicos, la lactosa es una fuente de carbono y energía tan buena como la glucosa. El nivel de AMPc no está relacionado con la concentración intracelular de glucosa, sino con la velocidad de transporte de glucosa, que influye en la actividad de la adenilato ciclasa. (Además, el transporte de glucosa también conduce a la inhibición directa de la permeasa de la lactosa.) En cuanto a por qué E. coli funciona de esta manera, solo se puede especular. Todas las bacterias entéricas fermentan la glucosa, lo que sugiere que la encuentran con frecuencia. Es posible que una pequeña diferencia en la eficiencia del transporte o metabolismo de la glucosa frente a la lactosa haga que sea ventajoso para las células regular el operón lac de esta manera. [23]
El gen lac y sus derivados se pueden utilizar como gen reportero en varias técnicas de selección basadas en bacterias, como el análisis de dos híbridos , en el que se debe determinar la unión exitosa de un activador transcripcional a una secuencia promotora específica. [16] En placas LB que contienen X-gal , el cambio de color de colonias blancas a un tono de azul corresponde a aproximadamente 20-100 unidades de β-galactosidasa, mientras que los medios de lactosa de tetrazolio y lactosa de MacConkey tienen un rango de 100-1000 unidades, siendo más sensibles en las partes altas y bajas de este rango respectivamente. [16] Dado que los medios de lactosa de MacConkey y lactosa de tetrazolio dependen de los productos de la descomposición de la lactosa, requieren la presencia de los genes lacZ y lacY . Las numerosas técnicas de fusión de lac que incluyen solo el gen lacZ son, por lo tanto, adecuadas para placas X-gal [16] o caldos líquidos ONPG . [24]