Una cuasiespecie viral es una estructura poblacional de virus con una gran cantidad de genomas variantes (relacionados por mutaciones). Las cuasiespecies son el resultado de altas tasas de mutación , ya que los mutantes surgen continuamente y cambian en frecuencia relativa a medida que avanza la replicación y selección viral . [1]
La teoría predice que una cuasiespecie viral en una región baja pero evolutivamente neutral y altamente conectada (es decir, plana) en el paisaje de aptitud superará a una cuasiespecie ubicada en un pico de aptitud más alto pero más estrecho en el que los mutantes circundantes no son aptos. [2] [3] Este fenómeno se ha llamado "el efecto cuasiespecie" o, más recientemente, la "supervivencia del más plano". [4]
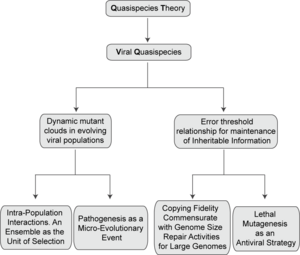
El término cuasiespecies fue adoptado de una teoría del origen de la vida en la que los replicones primitivos consistían en distribuciones mutantes, como las encontradas experimentalmente con los virus de ARN actuales dentro de su huésped . [5] [6] La teoría proporcionó una nueva definición de tipo salvaje al describir los virus, y un marco conceptual para una comprensión más profunda del potencial adaptativo de los virus de ARN que el que ofrecen los estudios clásicos basados en secuencias de consenso simplificadas . [1]
El modelo de cuasiespecies es más aplicable cuando el tamaño del genoma es limitado y la tasa de mutación es alta, y por lo tanto es más relevante para los virus de ARN (incluidos los patógenos importantes ) porque tienen altas tasas de mutación (aproximadamente un error por ronda de replicación) , [7] aunque los conceptos pueden aplicarse a otras entidades biológicas como los virus de ADN con transcripción inversa como el de la hepatitis B. [8] En tales escenarios, las distribuciones complejas de genomas variantes estrechamente relacionados están sujetas a variación genética , competencia y selección , y pueden actuar como una unidad de selección . Por lo tanto, la trayectoria evolutiva de la infección viral no se puede predecir únicamente a partir de las características de la secuencia más apta. Las altas tasas de mutación también colocan un límite superior compatible con la información hereditaria. Cruzar dicho límite conduce a la extinción del virus de ARN , una transición que es la base de un diseño antiviral denominado mutagénesis letal , y de relevancia para la medicina antiviral. [1]
La relevancia de las cuasiespecies en la virología ha sido objeto de un amplio debate. Sin embargo, los análisis clonales estándar y las metodologías de secuenciación profunda han confirmado la presencia de miríadas de genomas mutantes en poblaciones virales y su participación en procesos adaptativos . [1]
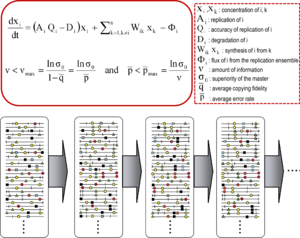
La teoría de las cuasiespecies fue desarrollada en la década de 1970 por Manfred Eigen y Peter Schuster para explicar la autoorganización y adaptabilidad de los replicones primitivos (término utilizado para referirse a cualquier entidad replicante), como ingrediente de organizaciones hipercíclicas que vinculan información genotípica y fenotípica , como un paso esencial en el origen de la vida. [11] [9] La teoría retrató a las poblaciones tempranas de replicones como espectros mutantes organizados dominados por una secuencia maestra, la dotada de mayor aptitud (capacidad replicativa) en la distribución. Introdujo la noción de un conjunto mutante como una unidad de selección, enfatizando así la relevancia de las interacciones intrapoblacionales para comprender la respuesta a las restricciones selectivas . Uno de sus corolarios es la relación del umbral de error , que marca la tasa máxima de mutación a la que la secuencia maestra (o dominante) puede estabilizar el conjunto mutante. La violación del umbral de error resulta en la pérdida de dominancia de la secuencia maestra y la deriva de la población en el espacio de secuencias . [9] [12] [13] [14]
Los conceptos básicos de cuasiespecies se describen mediante dos ecuaciones fundamentales: la replicación con producción de copias erróneas y la relación del umbral de error. Estas ecuaciones reflejan dos características principales de los virus de ARN a nivel de población: la presencia de un espectro mutante y el efecto adverso de un aumento de la tasa de mutación en la supervivencia del virus, cada una de las cuales tiene varias derivaciones. [1]
La existencia de un espectro mutante fue evidenciada experimentalmente primero por análisis clonales de poblaciones de bacteriófagos Qβ de ARN cuya replicación había sido iniciada por una sola partícula viral. Los genomas individuales diferían de la secuencia consenso en un promedio de una a dos mutaciones por genoma individual. [15] La aptitud de los clones biológicos fue inferior a la de la población parental, no clonada, una diferencia también documentada para el virus de la estomatitis vesicular (VSV). [16] La capacidad replicativa de un conjunto de población no necesita coincidir con la de sus componentes individuales. El hallazgo de que una población viral era esencialmente un grupo de mutantes llegó en un momento en que las mutaciones en la genética general se consideraban eventos raros, y los virólogos asociaban un genoma viral con una secuencia de nucleótidos definida , como todavía hoy se implica en el contenido de los bancos de datos . [17] La naturaleza nublada de Qβ se entendió como una consecuencia de su alta tasa de mutación, calculada en 10 −4 mutaciones introducidas por nucleótido copiado, [18] junto con la tolerancia de los genomas individuales a aceptar una proporción indeterminada de las mutaciones que surgen recientemente, a pesar de los costos de adaptación. La tasa de error estimada para el bacteriófago Qβ se ha confirmado y es comparable a los valores calculados para otros virus de ARN. [7] [19]
Se verificaron altas tasas de mutación y cuasiespecies para otros virus de ARN basándose en la disección de poblaciones virales por clonación molecular o biológica, y análisis de secuencia de clones individuales. John Holland y colegas fueron los primeros en reconocer que un mundo de ARN en rápida evolución insertado en una biosfera basada en ADN tenía múltiples implicaciones evolutivas y médicas. [1] [16] [20] [21] [22] La plasticidad genómica de los virus de ARN se había sospechado durante muchas décadas. Las primeras observaciones clave fueron las variaciones en los rasgos virales descritas por Findley en la década de 1930, los estudios de Granoff sobre las transiciones de la morfología de la placa del virus de la enfermedad de Newcastle , o la alta frecuencia de conversiones entre resistencia a fármacos y dependencia en el virus Coxsackie A9 , entre otros estudios con virus animales y vegetales a mediados del siglo XX. [23] Cuando se pone en el contexto del conocimiento actual, nos damos cuenta de que estas observaciones sobre los cambios fenotípicos eran la punta del iceberg de una realidad extremadamente compleja de poblaciones virales. Los virus ARN se caracterizan por unas tasas de mutación elevadas y una heterogeneidad poblacional que tiene consecuencias para la patogénesis viral y el control de las enfermedades virales. Se han realizado estudios detallados sobre la dinámica de las cuasiespecies in vivo con el virus de la inmunodeficiencia humana tipo 1 (VIH-1) y el virus de la hepatitis C. [10] [24] [25]
La primera formulación matemática de las cuasiespecies fue determinista; suponía distribuciones de mutantes en estado estacionario en equilibrio genético sin perturbaciones derivadas de modificaciones del entorno o del tamaño de la población . [26] Estas condiciones son comunes en las formulaciones teóricas iniciales de fenómenos complejos porque confieren manejabilidad matemática. Desde entonces, se han desarrollado varias extensiones de la teoría a condiciones de no equilibrio con componentes estocásticos, con el objetivo de encontrar soluciones generales para paisajes de aptitud de múltiples picos . Estos objetivos aproximan las cuasiespecies al caso real de los virus de ARN, que se ven obligados a lidiar con variaciones dramáticas en el tamaño de la población y el entorno. [27] La investigación sobre cuasiespecies ha avanzado a través de varias vías teóricas y experimentales que incluyen estudios continuos sobre la optimización evolutiva y el origen de la vida, las interacciones ARN-ARN y las redes de replicadores, el umbral de error en paisajes de aptitud variable, la consideración de la mutagénesis química y los mecanismos de corrección, la evolución de células tumorales , poblaciones bacterianas o células madre , la inestabilidad cromosómica , la resistencia a los fármacos y las distribuciones de conformación en priones (una clase de proteínas con potencial patogénico dependiente de la conformación; en este caso la cuasiespecies se define por una distribución de conformaciones). [10] [28] Los nuevos aportes a la investigación experimental de cuasiespecies provienen de la secuenciación profunda para investigar poblaciones virales y celulares, el reconocimiento de interacciones dentro de espectros mutantes, modelos de dinámica de poblaciones virales relacionadas con la progresión de enfermedades y la transmisión de patógenos, y nuevas enseñanzas de variantes de fidelidad de virus. [28] Aquí resumimos los principales aspectos de la dinámica de las cuasiespecies y los desarrollos recientes relevantes para la evolución y patogénesis del virus.
La base molecular de las altas tasas de error es la limitada fidelidad de copia de plantilla de las ARN polimerasas dependientes de ARN (RdRps) y las ADN polimerasas dependientes de ARN (también denominadas transcriptasas inversas, RT). Además, estas enzimas son defectuosas en la corrección de errores [29] porque carecen de un dominio de exonucleasa 3' a 5' presente en las ADN polimerasas celulares replicativas. [30] Además, las vías de reparación posreplicativa, abundantes para corregir lesiones genéticas en el ADN celular replicante, parecen ser ineficaces para el ARN bicatenario o los híbridos ARN-ADN. La presencia de una actividad de corrección de errores-reparación en los coronavirus aumenta su precisión de copia en aproximadamente 15 veces. [31] Esta y otras actividades de reparación, que pueden actuar sobre genomas de ARN estándar o retrovirales , [32] [33] [34] [35] no impiden la formación de espectros mutantes, aunque su amplitud puede ser menor que para otros virus de ARN, al menos en poblaciones cercanas a un origen clonal (genoma único). La dinámica de cuasiespecies operará en cualquier sistema viral o celular en el que debido a altas tasas de mutación (como resultado de polimerasas de ácidos nucleicos de baja fidelidad o alteraciones ambientales) se generen rápidamente espectros mutantes. [10] [36] [37] [38] [39] [40]
Estudios con diferentes sistemas virus-huésped han establecido algunas observaciones generales sobre los mecanismos de generación de mutantes, e implicaciones de la dinámica de cuasiespecies. [10] [41] [42] [43] [44] [45] [46 ] [ 47] [48] [49] [50] En genética de virus ARN cuando hablamos de "un mutante" la entidad que manejamos es una nube de mutantes en la que la mutación específica a la que dirigimos nuestra atención está presente en todos (o la gran mayoría de) los genomas individuales. No existe tal cosa como "un" tipo salvaje o "un" virus mutante. Siempre son nubes de mutantes. Los cambios en la dominancia relativa de los componentes de los espectros mutantes son particularmente severos durante las infecciones in vivo , con dinámicas complejas de heterogeneidad y variaciones intrahuésped. Se han desarrollado procedimientos bioinformáticos para revelar las relaciones entre tipos de genomas diferentes pero estrechamente relacionados que pueden sugerir algún orden jerárquico de adquisición de mutaciones o identificación de grupos de transmisión (algunos ejemplos son el Análisis de Partición de Cuasiespecies , PAQ [51] o la Evolución de Cuasiespecies , Inferencia de Transmisión Basada en Redes , QUENTIN [52] ).
El quid de la cuestión en cuanto a las implicaciones de las cuasiespecies es que, en un momento dado, la población viral incluye un reservorio no solo de variantes genotípicas sino también de variantes fenotípicas , lo que le confiere cierta pluripotencia adaptativa . La acumulación de evidencia clínica y de laboratorio hace insostenible que los componentes minoritarios de los espectros mutantes deban descartarse por ser neutrales . Pueden participar en procesos selectivos y no pueden excluirse de las interpretaciones del comportamiento del virus. La variación implica universalmente mutaciones puntuales y también puede incluir la recombinación (en sus modos replicativo y no replicativo) y la reordenación de segmentos del genoma . [42] Todos los modos de variación molecular son compatibles, solo restringidos por el alcance de los mecanismos accesibles a la maquinaria replicativa y por la necesidad de que los genomas virales sigan siendo funcionales. David Evans y sus colegas identificaron muchos eventos de recombinación asociados con la replicación de enterovirus , y solo unos pocos recombinantes lograron abrirse camino hacia la replicación continua. [53] La recombinación puede mediar la adaptabilidad y la virulencia . [54] Las altas tasas de mutación y recombinación han llevado a la distinción conceptual entre variación mecanísticamente inevitable y variación evolutivamente relevante, en conexión con la cuestión de la naturaleza clonal versus no clonal de la evolución del virus (evolución microbiana en general). [55] [56] Solo una minoría de la variación naciente durante la replicación puede propagarse con éxito. Dentro de los límites establecidos por las restricciones biológicas, cada población está formada por una serie de genomas variantes, con un número total que es proporcional al tamaño de la población del virus. Infectar una planta, un animal o un cultivo celular con 10 3 unidades infecciosas puede tener consecuencias muy diferentes a infectar con 10 10 unidades infecciosas, no solo porque los sistemas de defensa del huésped pueden verse abrumados por la alta dosis infecciosa, sino también porque el repertorio mutante que participa en exploraciones adaptativas es más grande. Parte de las variantes de un espectro mutante, ya sea de forma aislada o en consorcio con otras, [57] puede funcionar mejor que otros miembros de la misma población en caso de un cambio ambiental. Las presiones selectivas favorecen la replicación de algunos componentes de un espectro mutante sobre otros, a pesar de que todos ellos están interconectados por la mutación. El rendimiento diferencial puede darse a nivel de genomas virales (durante la replicación, la expresión génica intracelular, interacción con factores del huésped, etc.) o partículas virales (para estabilidad térmica, entrada o salida de células, para soportar anticuerpos neutralizantes, etc.). [22] [10] [23] [24] [25] [42] [43] [44] La adaptabilidad de los virus de ARN está vinculada a parámetros que facilitan la exploración del espacio de secuencias: tamaño del genoma (1,8 a 33 Kb), tamaño de la población (variable pero que puede alcanzar unos impresionantes 10 12 genomas individuales en un huésped infectado en un momento dado), tasa de replicación, tasa de mutación, fecundidad (rendimiento de partículas virales por célula) y número de mutaciones necesarias para un cambio fenotípico (sorprendentemente bajo para varios rasgos relevantes [58] ).
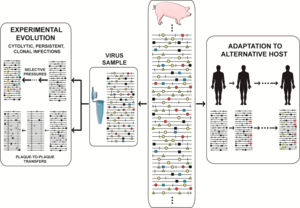
La dinámica del espectro mutante se ha representado de diferentes maneras, y hemos elegido una que abarca eventos frecuentes en poblaciones naturales y diseños de investigación, como el aislamiento del virus de un huésped infectado, la adaptación al cultivo celular para estudios sobre evolución experimental o la adaptación a huéspedes alternativos in vivo. La realidad es aún más compleja, dados los grandes tamaños de población, con una proporción indeterminada de genomas que se replican activamente en un momento dado (a veces equiparada con el tamaño efectivo de la población en genética general), y que albergan múltiples mutaciones por genoma. Los escenarios sugeridos por los datos experimentales actuales desafían nuestra imaginación. La frecuencia relativa de las mutaciones individuales fluctúa en una exploración incesante del espacio de secuencias, [59] [60] [10] con cambios fenotípicos (no solo cambios genotípicos) que son mucho más frecuentes de lo que se pensaba anteriormente. El diseño de evolución experimental que consiste en pasar poblaciones virales durante largos períodos de tiempo (muchas infecciones secuenciales) es a menudo extremadamente revelador. En el caso del virus de la fiebre aftosa (FMDV), este diseño condujo a una notable diversificación fenotípica en subpoblaciones de colonizadores y competidores, que modularon la virulencia del conjunto mutante. [61] En el caso del VHC, este diseño reveló ondas de mutación continuas y una comprensión más precisa de los tipos de paisajes de adaptación ocupados por virus de alta adaptación. [60] [62]
La secuencia de nucleótidos de un genoma individual de una población (sin importar cuál sea el grado de complejidad de la población), puede determinarse ya sea luego de un evento de clonación biológica o molecular o por secuenciación profunda de genomas virales completos, de manera que se pueda establecer el ligamiento de mutaciones (asignación de diferentes mutaciones a la misma molécula del genoma). Cada uno de estos procedimientos implica algunas limitaciones: la clonación biológica puede sesgar la representación a favor de genomas infecciosos, mientras que la clonación molecular puede introducir genomas no infecciosos (defectuosos) en el análisis. [22] [58] [59] La descripción de cuasiespecies de genoma completo aún es técnicamente desafiante debido a la introducción artificial de mutaciones. La mayoría de las plataformas de secuenciación profunda actuales producen secuencias de lecturas cortas para un amplicón dado (secuencia bajo análisis); las mutaciones minoritarias en un amplicón no pueden vincularse de manera confiable a mutaciones en un amplicón diferente del mismo genoma; como máximo, se pueden proponer inferencias estadísticas sobre el ligamiento. A pesar de estas limitaciones, los experimentos de control y las mejoras de los procedimientos bioinformáticos confirman que la mayor parte de la heterogeneidad de secuencias analizada en poblaciones virales refleja de hecho diferencias en las poblaciones de plantilla naturales. Si se puede resolver el vínculo entre mutaciones de forma rutinaria, entrará en escena una nueva oleada de información molecular relevante para las interacciones epistáticas.
Existen niveles adicionales de indeterminación en el análisis secuencial de poblaciones virales, en particular las que se replican in vivo. Los componentes del espectro mutante representados en un momento dado en la muestra tomada para la secuenciación pueden diferir de los del siguiente punto temporal, ya sea debido a incertidumbres en el muestreo o a fluctuaciones genuinas de las frecuencias del genoma. No está justificado aceptar una similitud aproximada porque incluso una única mutación en un contexto de secuencia dado puede afectar las propiedades biológicas. [10] En palabras de John Holland y colegas: "Es importante recordar que cada enjambre de genomas de cuasiespecies en un individuo infectado es único y "nuevo" en el sentido de que nunca antes ha existido una población idéntica de genomas y nunca volverá a existir". [63] Además de la naturaleza fugaz de cualquier distribución mutante, los métodos estándar disponibles para la caracterización de cuasiespecies proporcionan secuencias genómicas de una minoría de la población (estimadas en 10 −8 a 10 −13 para la secuenciación molecular por clonación-Sanger, y en 10 −6 a 10 −11 para la secuenciación profunda). [58] Solo podemos tener una representación aproximada de las poblaciones virales y su dinámica, como lo evidencian muchos estudios experimentales. [15] [20] [10] [43] [44] [58] [60] [64]
Los puntos resumidos en las secciones anteriores justifican plenamente abordar las herramientas analíticas hacia el espectro mutante en lugar de ignorarlo o considerar su presencia como una cuestión secundaria. El uso de secuencias de consenso para describir el genoma de un aislado de virus, a pesar de estar justificado por las dificultades de transmitir la información recapitulada en un espectro mutante, desdibuja y debilita las interpretaciones biológicas. Los resultados experimentales han demostrado que los genomas minoritarios de un espectro mutante (que no se pueden identificar examinando la secuencia de consenso) pueden incluir mutaciones que confieren resistencia a inhibidores antivirales , anticuerpos neutralizantes o células T citotóxicas , o que pueden alterar la capacidad de inducir interferón (IFN) o de responder a IFN, la virulencia o la estabilidad de partículas, entre otros rasgos fenotípicos. [10] [42 ] [46] [60] [64] [65] [66] [67] Los espectros mutantes también pueden mediar la adaptación cíclica a diferentes tipos de células. [41] Un espectro mutante define un consenso, pero el consenso es una abstracción; Puede que no esté representado en la población. Muchos eventos en la patogénesis y evolución viral se deben a modificaciones del espectro mutante o interacciones que no pueden interpretarse adecuadamente basándose únicamente en secuencias de consenso. [15] [10] [22] [23] [28] [41] [40] [43] [44] [53] [54] [57] [60] [63]
Los espectros mutantes no son meros agregados de mutantes que actúan de forma independiente, sino que suelen participar en respuestas colectivas. [1] Los dos tipos principales son los que dependen de la presencia de conjuntos de variantes y los que dependen de interacciones dentro de los espectros mutantes.
En algunos casos de selección de barrido (selección muy fuerte para un rasgo), un individuo (o un número limitado de individuos) que codifica firmas propensas a ser seleccionadas, puede acercarse a la dominancia mientras se convierte en el fundador de una nube de mutantes (porque la formación de una nube es inherente a la replicación). Las condiciones para la dominancia (en este caso en respuesta a la selección) son que el genoma detecte el barrido selectivo y que se permita su replicación en el nuevo entorno selectivo. En otros casos, se selecciona una colección de mutantes. Esto se ilustró con una cuasiespecies de FMDV que se reconstruyó en el laboratorio con múltiples variantes antigénicas (cada una de ellas a baja frecuencia) que pertenecían a dos categorías diferentes y compartían resistencia al mismo anticuerpo monoclonal . [68] Una categoría incluía mutantes con una sustitución de aminoácidos que afectaba el reconocimiento del receptor (ya que el determinante antigénico se superponía con el sitio de reconocimiento del receptor de integrina ); en la otra categoría, las sustituciones afectaban al determinante antigénico pero no al sitio de reconocimiento del receptor. Los pasajes del virus en ausencia del anticuerpo monoclonal dieron como resultado el predominio de variantes antigénicas que mantuvieron la capacidad de reconocimiento del receptor, pero las variantes dominantes estaban rodeadas por una nube de mutantes de la otra categoría de variantes antigénicas. Por el contrario, los pasajes en presencia del anticuerpo dieron lugar a la selección de variantes con un reconocimiento del receptor alterado, rodeadas por una nube de variantes antigénicas que mantuvieron el reconocimiento del receptor. Los resultados subrayaron el papel de las nubes mutantes en los eventos selectivos y revelaron un nuevo mecanismo de flexibilidad antigénica. [68]
La memoria de cuasiespecies es un tipo de memoria molecular que depende de la historia reciente del linaje evolutivo y de la integridad del espectro mutante. [69] [70] La búsqueda de la memoria fue impulsada por el comportamiento complejo del sistema adaptativo de una cuasiespecies viral, sugerido por la presencia de información central (considerada la que define la identidad viral) a pesar de la variación de los elementos constitutivos (el espectro mutante). Un ejemplo bien conocido es la memoria en el sistema inmunológico que moviliza y expande componentes minoritarios en respuesta a estímulos previamente enfrentados por el sistema. [71] En los experimentos diseñados para identificar la memoria en cuasiespecies virales, los miembros del espectro mutante aumentaron en frecuencia como consecuencia de su replicación durante un evento de selección que los llevó hacia el dominio. Cuando se retiró la restricción selectiva, los genomas de memoria permanecieron en niveles que eran 10 a 100 veces más altos que los niveles basales atribuibles únicamente a su generación por mutación, como se documentó con marcadores genéticos independientes de FMDV y con VIH-1 in vivo . [69] [70] [72] [73] Así, la memoria es una propiedad colectiva dependiente de la historia de las cuasiespecies que les confiere una ventaja selectiva para responder a cambios ambientales previamente experimentados por el mismo linaje evolutivo. Puede manifestarse solo si el espectro mutante mantiene su completitud, ya que la memoria se pierde cuando la población sufre un evento de cuello de botella que excluye a las minorías. Un ejemplo relevante de las consecuencias de la memoria se da en la farmacología antiviral con la administración por segunda vez del mismo agente antiviral o de uno relacionado (capaz de evocar mutaciones de resistencia compartidas) utilizado en un tratamiento previo. La segunda intervención puede enfrentarse a genomas de memoria resistentes a inhibidores del tratamiento anterior, contribuyendo así al escape del virus. [69] Este es un aspecto que no ha recibido la atención adecuada en la planificación de intervenciones antivirales para pacientes que fracasan en un primer tratamiento y tienen que ser sometidos a un segundo tratamiento.
Los genomas individuales rodeados por una nube de mutantes relacionados pueden ser suprimidos para mantenerlos en baja frecuencia, o pueden ser ayudados para mantenerlos en la población. Los dos destinos alternativos dependen de varios factores, uno de los cuales es el espectro mutante circundante en aquellos pasos del ciclo infeccioso en los que se establece una competencia efectiva entre variantes, por ejemplo dentro de los complejos de replicación. Este importante concepto fue derivado primero teóricamente, [12] [74] y luego abordado experimentalmente con varios virus. En un estudio temprano, Juan Carlos de la Torre y John Holland describieron la supresión de VSV de alta aptitud por espectros mutantes de aptitud inferior. [12] Desde entonces, se han documentado efectos supresores con poblaciones virales estándar y mutagenizadas. Algunos ejemplos son:
Lo opuesto a la supresión es el mantenimiento de un mutante, ya sea por una posición favorable en un panorama de aptitud o por interacciones de complementación o cooperación con miembros del espectro mutante. La posición en un panorama de aptitud influye en la vulnerabilidad a las mutaciones, como se popularizó con los términos "ventaja del más plano" o "supervivencia del más plano", lo que indica que una variante ubicada en la parte superior de un pico de aptitud agudo tiene mayor probabilidad de disminuir la aptitud como resultado de nuevas mutaciones que la misma variante ubicada en una meseta de aptitud . [3] [81] [82] La supervivencia del más plano también se ha propuesto como un ingrediente en algunos modelos del umbral de error. [83]
El comportamiento colectivo de los virus se ha documentado con virus ARN mutantes resistentes a análogos de nucleótidos. El estudio de esta clase de mutantes ha sido fundamental para la comprensión de la base molecular de la fidelidad de copia de la plantilla y las consecuencias de las alteraciones de la fidelidad en la capacidad adaptativa y el potencial patógeno de los virus ARN. [84] [85] [86] En el primer mutante estudiado, la sustitución del aminoácido G46S en la polimerasa PV resultó en un aumento de aproximadamente cuatro veces en la fidelidad de copia de la plantilla. Esta modificación redujo la adaptabilidad y el potencial infeccioso de PV in vivo. [84] [85] El mutante aislado no se replicó de manera eficiente en el cerebro de ratones susceptibles, pero sí lo hizo cuando su espectro mutante se amplió mediante mutagénesis con 5-fluorouracilo o cuando se co-inoculó con PV de tipo salvaje. [85]
La complementación (que ocurre a menudo cuando una proteína funcional codificada por un conjunto de genomas es utilizada por otro conjunto de genomas cuya proteína codificada no es funcional) puede ser la base de algunas respuestas colectivas de las cuasiespecies, como la aptitud de los individuos aislados de una población que es inferior a la aptitud de la población. [15] [30] Se describió la complementación entre dos formas genómicas truncadas del virus de la fiebre aftosa. [87] Los genomas con deleciones internas se volvieron detectables tras el pasaje de alta multiplicidad de una población clonal del virus de la fiebre aftosa estándar, un virus con un genoma de ARN monocatenario monopartito. La infectividad se generó por complementación de las dos formas truncadas, en ausencia de genomas estándar de longitud completa del virus de la fiebre aftosa. Para que la complementación fuera efectiva, era necesario explorar previamente el espacio de secuencias a través de mutaciones puntuales. [88] El sistema experimentó una notable transición evolutiva similar a la segmentación del genoma. Es difícil observar lesiones genéticas drásticas en los genomas virales a menos que un mecanismo como la complementación venga al rescate de los genomas desviados. Se han informado ejemplos adicionales de complementación entre virus de ARN. [89] [90] [91] [42] [44] La complementación es un medio para mantener genomas defectuosos en frecuencias detectables en poblaciones virales.
Se ha hecho una distinción entre complementación y cooperación, en la que dos genomas diferentes dan lugar a un nuevo fenotipo a través de la interacción entre dos proteínas variantes. [92] Un ejemplo de cooperación se caracterizó durante estudios con el virus del sarampión sobre la fusión de membranas, que es esencial para la entrada del virus en las células. Para este virus, la fusión está mediada por dos proteínas denominadas H y F. Una H truncada era deficiente en la fusión celular, pero la actividad se recuperó cuando la H truncada estaba acompañada por dos formas de F, pero no una de las formas individualmente. [92]
Por lo tanto, la complementación, la cooperación, la interferencia y la supresión pueden surgir de interacciones entre componentes de espectros mutantes que tienen su origen en mutaciones aleatorias. La selección actúa sobre cualquier conjunto de mutantes que pueda proporcionar un rasgo útil, para convertir sucesos aleatorios en significado biológico.

Una forma de interrumpir la participación de genomas individuales en interacciones con su espectro mutante es que el enjambre de cuasiespecies experimente reducciones drásticas en el tamaño de la población que aíslen uno o unos pocos genomas individuales de su entorno. Estas reducciones se denominan cuellos de botella y tienen una participación importante en la conformación de linajes evolutivos para todo tipo de organismos, y también para los virus. Ocurren con frecuencia no solo en la transmisión de huésped a huésped sino también dentro de huéspedes infectados, [93] [94] [95] y pueden perturbar eventos de selección positiva y negativa en procesos que son difíciles de identificar y caracterizar.
Se han reproducido eventos de cuello de botella drásticos con poblaciones de virus de laboratorio en forma de transferencias de placa a placa. [96] [97] Este diseño sirvió para verificar experimentalmente el funcionamiento del trinquete de Müller , o disminución de la aptitud por la incorporación irreversible de mutaciones en organismos asexuales en ausencia de mecanismos compensatorios. [98] Las transferencias de cuello de botella en serie revelaron la presencia de mutaciones raras, no vistas en poblaciones virales naturales o de laboratorio estándar. En ausencia de eventos de cuello de botella forzados, tales mutaciones raras se perderían por selección negativa debido a su costo de aptitud. [99] La investigación de cómo los clones de FMDV debilitados por el trinquete de Müller recuperaron la aptitud replicativa reveló varias vías moleculares alternativas para la recuperación de la aptitud. [100] Las implicaciones de esta observación pasaron en gran medida desapercibidas hasta que resultados recientes con el virus de la hepatitis C (VHC) también sugirieron la accesibilidad de múltiples vías para la ganancia de aptitud. [60] [62] Además, el paso extensivo de un clon biológico de FMDV en células BHK-21 confirió la capacidad de infectar varias líneas celulares humanas además del aumento esperado de la aptitud para la multiplicación en células BHK-21. [101] Por lo tanto, varias líneas de evidencia sugieren que la ganancia de aptitud en un entorno específico puede ampliar paradójicamente el potencial fenotípico de un virus. Será interesante investigar si la adaptación enfocada de otros virus a un entorno específico también puede implicar una ampliación de la diversidad, con muchas variantes fenotípicas alcanzando niveles de aptitud similares. Si se generalizara, esta ampliación del espacio fenotípico proporcionaría una nueva interpretación de la base molecular de la adaptación y explicaría por qué la adaptación a entornos alternativos puede no conducir a la atenuación .
La privación de un virus individual de una posible supresión, complementación o cooperación puede representar una liberación para iniciar un nuevo proceso evolutivo, o una condena a la extinción. Si se libera de la supresión, el genoma aislado debe replicarse y ser capaz de reconstruir una nube mutante para recuperar la capacidad adaptativa. Esto ha llevado a la sugerencia de que las altas tasas de mutación evolucionaron para permitir esa recuperación del espectro mutante después de los cuellos de botella. Otros modelos atribuyen las altas tasas de mutación a la optimización adaptativa independiente de los cuellos de botella, o a una consecuencia mecanicista de la replicación rápida. [58] Cualquiera que sea su origen último, las altas tasas de mutación sirven al propósito de la adaptación en múltiples circunstancias, no solo después de los cuellos de botella. Un virus fundador puede introducir un fenotipo diferente para la evolución subsiguiente. La evolución de los virus en la naturaleza y como agentes patógenos puede verse como una sucesión de alteraciones del espectro mutante, sujetas a expansiones y reducciones del tamaño de la población en una interacción continua de selección positiva y negativa y deriva aleatoria. Si bien la evolución a corto plazo (por ejemplo, intrahuésped) es observable y medible, los virus pueden parecer relativamente estáticos a largo plazo durante décadas (como se ha visto con las variantes antigénicas del virus de la fiebre aftosa [102] ) o más. La evolución intrahuésped es generalmente más rápida que la evolución interhuésped, como se ha documentado con los virus [10] y otros sistemas biológicos. [103] La aparente invariancia puede ser el resultado de la selección para la supervivencia a largo plazo de poblaciones que previamente han probado frenéticamente los resultados evolutivos en procesos a corto plazo. [58]
Poco después de que se descubrieran las cuasiespecies de los virus, se hicieron explícitas algunas implicaciones médicas. [20] [104] A continuación se presentan varios puntos específicos y generales. [10] [43] [66] [28]
En todas las interacciones que conducen a la enfermedad, las células hospedadoras, tanto individualmente como en grupos en tejidos y órganos, desempeñan papeles decisivos. Las consecuencias de una infección viral siempre dependen del hospedador. Sin embargo, el propio virus plantea un importante desafío que una comprensión más profunda de la dinámica de las cuasiespecies está ayudando a afrontar. [28]
Existe una percepción cada vez mayor de que los principios darwinianos deberían ayudar en la planificación de diseños antivirales. [105] El objetivo de la vacunación es provocar una respuesta protectora que impida la replicación del virus o la enfermedad. El objetivo de una intervención farmacológica antiviral es inhibir la replicación del virus para proporcionar al sistema inmunológico la oportunidad de eliminar el virus. Expresado de manera simple, el peligro directo para la vacunación y el tratamiento es que el virus pueda escapar a través de la selección de mutantes resistentes a los componentes de defensa activados por la vacuna o a los inhibidores administrados externamente. Esto ha llevado a varias propuestas para enfrentar la enfermedad viral, que pueden resumirse a continuación. [58]
Las vacunas deben incluir repertorios de epítopos de células B y células T para evocar una amplia respuesta inmune . La amplia respuesta debe minimizar la selección de mutantes de escape que pueden estar presentes como componentes minoritarios en los espectros mutantes, como se documentó repetidamente experimentalmente. [10] [22] [44] [69] Con los tipos actuales de vacunas disponibles, las que mejor cumplen con el requisito de múltiples epítopos son, en el orden de eficacia esperada para conferir protección contra virus altamente variables: atenuado > virus completo inactivado > varias proteínas expresadas > una proteína expresada > múltiples antígenos peptídicos sintéticos > antígeno peptídico único. La escasez de vacunas sintéticas efectivas para patógenos virales de ARN a pesar de enormes esfuerzos científicos y económicos es un reflejo de los problemas subyacentes.
Se debe evitar la monoterapia antiviral (uso de un solo agente antiviral). Se han hecho las siguientes recomendaciones, que en algunos casos se han aplicado con éxito:
Estas estrategias tienen como objetivo principal evitar la selección de mutantes que escapen al tratamiento por múltiples restricciones selectivas que no pueden ser superadas por el virus. [58] [106] El control es efectivo ya sea porque la exploración del espacio de secuencias no puede alcanzar las múltiples mutaciones requeridas (incluso cuando la recombinación está disponible) o porque las múltiples mutaciones infligen un costo de aptitud severo. [106] Las vacunas que exponen múltiples epítopos y las terapias combinadas siguen la misma estrategia cuyo objetivo es limitar las posibles rutas de escape a las cuasiespecies virales frente a la restricción supresora.
La mutagénesis letal es el proceso de extinción de un virus a una tasa de error en la que el virus ya no puede mantener su información genética. [10] [28] [44] [58] [62] [83] [107] [108] La aplicación de la mutagénesis letal como estrategia antiviral merece atención en el contexto del presente artículo porque sus orígenes se encuentran en la teoría de las cuasiespecies, en la forma de la relación del umbral de error. Tanto el umbral de error como la mutagénesis letal dependen en gran medida del entorno de aptitud, pero ambos pueden ocurrir en entornos de aptitud complejos como los pertinentes a las poblaciones virales. [83] El término mutagénesis letal fue acuñado por Lawerence Loeb y colegas, [107] y ahora se usa ampliamente para describir la actividad antiviral de los análogos de bases y nucleósidos que aumentan la tasa de mutación viral. Aunque se han propuesto varios modelos para explicar la extinción del virus por mutaciones excesivas, [83] una extensión de la violación del umbral de error se presenta como un mecanismo probable. [109] [108] Curiosamente, algunos agentes antivirales autorizados para uso humano, que inicialmente se pensaba que actuaban sólo como inhibidores de la replicación viral, en realidad pueden ejercer su actividad antiviral contra algunos virus ARN al menos parcialmente mediante mutagénesis letal. Este es el caso del favipiravir (T-705; 6-fluoro-3-hidroxi-2-pirazinacarboxamida) y la ribavirina (1-β-D-ribofuranosil-1-H-1,2,4-triazol-3-carboxamida) que actualmente se están investigando intensamente como mutágenos letales. [108]
Los mecanismos de defensa basados en la modificación del genoma de parásitos genéticos invasores, como la edición de actividades celulares que se reclutan como parte de la respuesta inmune innata ( ADAR , APOBEC , RIP , etc.) [110] representan una contraparte natural del principio utilizado por la mutagénesis letal. La aplicabilidad a elementos celulares patógenos es una posibilidad real, y la mutagénesis letal para controlar células tumorales es un campo activo de investigación. [111] [112] Por lo tanto, el reconocimiento de la dinámica de las cuasiespecies ha sugerido algunas pautas fundamentales para la prevención y el control de enfermedades que están permeando gradualmente la práctica clínica. Esto está en línea con la necesidad reconocida de aplicar los principios darwinianos al control de las enfermedades infecciosas.
Esto puede definirse como "la incapacidad de un elemento genético de mantenerse en una población a medida que la fidelidad de su maquinaria de replicación disminuye más allá de un cierto valor umbral". [113]
En teoría, si la tasa de mutación fuera lo suficientemente alta, la población viral no sería capaz de mantener el genotipo con la mayor aptitud y, por lo tanto, se vería comprometida la capacidad de la población para adaptarse a su entorno. Una aplicación práctica de esta dinámica es en los medicamentos antivirales que emplean mutagénesis letal. Por ejemplo, el aumento de dosis del mutágeno ribavirina reduce la infectividad del poliovirus. [114]
Sin embargo, estos modelos suponen que sólo las mutaciones que se producen en la secuencia más apta son deletéreas y, además, no letales. Se ha argumentado que, si tenemos en cuenta el efecto deletéreo de las mutaciones en la población de variantes y el hecho de que muchas mutaciones son letales, entonces el umbral de error desaparece, es decir, la secuencia más apta siempre se mantiene. [115] [113] [116] Los datos empíricos sobre el efecto de las mutaciones en los virus son escasos, pero parecen corresponderse con este escenario. [117]
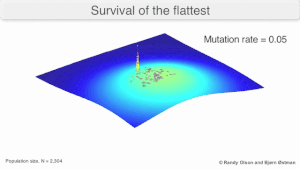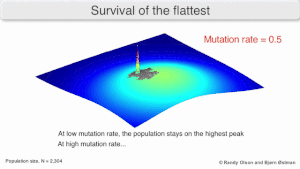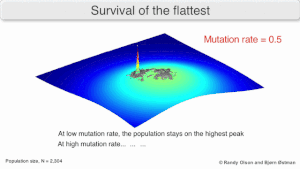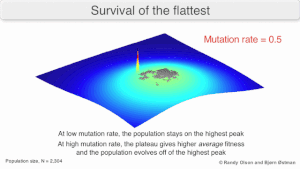
La evolución a largo plazo del virus puede verse influida por el hecho de que puede ser una mejor estrategia evolutivamente estable generar una amplia cuasiespecies con miembros de una aptitud aproximadamente igual que tener un único genotipo "más apto" claramente definido (con vecinos mutacionales sustancialmente menos aptos). Esto se ha denominado "supervivencia del más plano", en referencia a los perfiles de aptitud de las dos estrategias respectivamente. [3]
A largo plazo, un perfil de aptitud más plano podría permitir que una cuasiespecie aproveche mejor los cambios en la presión de selección , de manera análoga a la forma en que los organismos sexuales utilizan la recombinación para preservar la diversidad en una población. Al menos en las simulaciones, se puede demostrar que un replicador más lento puede superar en competencia a uno más rápido en los casos en que es más robusto y la tasa de mutación es alta. [2]
Sin embargo, no está confirmado si la robustez mutacional evolucionó o es intrínseca a los sistemas genéticos, porque el mecanismo básico detrás de la robustez dependería de las peculiaridades de cada sistema. [4]
La manipulación experimental de los poliovirus para darles una polimerasa de mayor fidelidad –y, por lo tanto, reducir su tasa de mutación– mostró que estas variantes tenían una patogenicidad menor que las secuencias de tipo salvaje . [85] La patogenicidad podría entonces ser restaurada mediante la aplicación de mutágenos. Esto se interpretó como que las tasas de mutación más bajas habían reducido la adaptabilidad (o amplitud) de las cuasiespecies. Los virus mutantes extraídos del tejido cerebral no eran patógenos en sí mismos, y los autores especulan que puede haber complementación entre los miembros variantes de las cuasiespecies que podría permitir a los virus colonizar diferentes tejidos y sistemas hospedadores.
![]() Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2000) (informes de los revisores): Esteban Domingo Solans, Celia Perales (17 de octubre de 2019). "Viral quasiespecies". PLOS Genetics . 15 (10): e1008271. doi : 10.1371/JOURNAL.PGEN.1008271 . ISSN 1553-7390. PMC 6797082 . PMID 31622336. Wikidata Q86320171.
Este artículo fue adaptado de la siguiente fuente bajo licencia CC BY 4.0 (2000) (informes de los revisores): Esteban Domingo Solans, Celia Perales (17 de octubre de 2019). "Viral quasiespecies". PLOS Genetics . 15 (10): e1008271. doi : 10.1371/JOURNAL.PGEN.1008271 . ISSN 1553-7390. PMC 6797082 . PMID 31622336. Wikidata Q86320171.
{{cite book}}: |journal=ignorado ( ayuda ){{cite book}}: |journal=ignorado ( ayuda )