Dicer , también conocida como endorribonucleasa Dicer o helicasa con motivo ARNasa , es una enzima que en los humanos está codificada por el gen DICER1 . Al ser parte de la familia de las ARNasas III , Dicer escinde el ARN bicatenario (dsRNA) y el pre-microRNA (pre-miRNA) en fragmentos cortos de ARN bicatenario llamados ARN interferente pequeño y microRNA , respectivamente. Estos fragmentos tienen aproximadamente entre 20 y 25 pares de bases de longitud con un saliente de dos bases en el extremo 3' . Dicer facilita la activación del complejo de silenciamiento inducido por ARN (RISC), que es esencial para la interferencia del ARN . RISC tiene un componente catalítico Argonaute , que es una endonucleasa capaz de degradar el ARN mensajero (ARNm). [5]
Dicer recibió su nombre en 2001 de la mano de Emily Bernstein, estudiante de doctorado de Stony Brook, mientras realizaba una investigación en el laboratorio de Gregory Hannon en Cold Spring Harbor Laboratory . Bernstein buscaba descubrir la enzima responsable de generar pequeños fragmentos de ARN a partir de ARN bicatenario. La capacidad de Dicer para generar alrededor de 22 fragmentos de ARN de nucleótidos se descubrió separándolo del complejo enzimático RISC después de iniciar la vía RNAi con transfección dsRNA . Este experimento mostró que RISC no era responsable de generar los pequeños fragmentos de nucleótidos observables. Experimentos posteriores que probaron las capacidades de las enzimas de la familia RNasa III para crear fragmentos de ARN limitaron la búsqueda a Drosophila CG4792, ahora llamada Dicer. [6]
Los ortólogos de Dicer están presentes en muchos otros organismos. [7] En el musgo Physcomitrella patens, DCL1b, una de las cuatro proteínas DICER, no está involucrada en la biogénesis de miRNA sino en la división de las transcripciones de miRNA objetivo. Por lo tanto, se descubrió un nuevo mecanismo para la regulación de la expresión génica , el silenciamiento epigenético de genes por miRNA. [8]
En términos de estructura cristalina, el primer Dicer que se exploró fue el del protozoo Giardia intestinalis . El trabajo fue realizado por Ian MacRae mientras realizaba una investigación como becario postdoctoral en el laboratorio de Jennifer Doudna en la Universidad de California, Berkeley . Se descubrieron un dominio PAZ y dos dominios de ARNasa III mediante cristalografía de rayos X. El tamaño de la proteína es de 82 kDa , lo que representa el núcleo funcional conservado que posteriormente se ha encontrado en proteínas Dicer más grandes en otros organismos; por ejemplo, es de 219 kDa en humanos. La diferencia de tamaño entre los humanos y los Dicer de G. intestinalis se debe a que hay al menos cinco dominios diferentes presentes en el Dicer humano. Estos dominios son importantes en la regulación de la actividad de Dicer, el procesamiento de dsRNA y el funcionamiento del factor de proteína de interferencia de ARN. [9]

El dicer humano (también conocido como hsDicer o DICER1 ) se clasifica como una ribonucleasa III porque corta el ARN bicatenario. Además de dos dominios de ARNasa III, contiene un dominio de helicasa , un dominio PAZ ( Piwi / Argonaute /Zwille) , [11] [12] y dos dominios de unión al ARN bicatenario (DUF283 y dsRBD). [9] [13]
Las investigaciones actuales sugieren que el dominio PAZ es capaz de unirse al saliente 3' de 2 nucleótidos del dsRNA mientras que los dominios catalíticos de la ARNasa III forman un pseudodímero alrededor del dsRNA para iniciar la escisión de las hebras. Esto da como resultado un acortamiento funcional de la hebra del dsRNA. La distancia entre los dominios PAZ y RNasa III está determinada por el ángulo de la hélice del conector e influye en la longitud del producto de micro ARN. [10] El dominio dsRBD se une al dsRNA, aunque no se ha definido el sitio de unión específico del dominio. Es posible que este dominio funcione como parte de un complejo con otras proteínas reguladoras (TRBP en humanos, R2D2, Loqs en Drosophila ) para posicionar eficazmente los dominios de la ARNasa III y así controlar la especificidad de los productos de sRNA. [14] El dominio de la helicasa se ha implicado en el procesamiento de sustratos largos. [14]
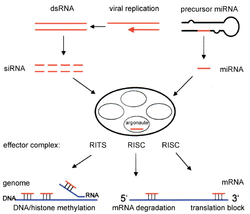
La interferencia de ARN es un proceso en el que la descomposición de las moléculas de ARN en miARN inhibe la expresión génica de secuencias específicas de ARNm del huésped. El miARN se produce dentro de la célula a partir del miARN primario (pri-miARN) en el núcleo . Estas secuencias largas se escinden en miARN precursores más pequeños (pre-miARN), que suelen tener 70 nucleótidos con una estructura de horquilla . Los pri-miARN son identificados por DGCR8 y escindidos por Drosha para formar el pre-miARN, un proceso que ocurre en el núcleo. Estos pre-miARN luego se exportan al citoplasma, donde son escindidos por Dicer para formar miARN maduro. [16]
Los ARN interferentes pequeños (siRNA) se producen y funcionan de manera similar a los miRNA al escindir el ARN bicatenario con Dicer en fragmentos más pequeños, de 21 a 23 nucleótidos de longitud. [14] Tanto los miRNA como los siRNA activan el complejo de silenciamiento inducido por ARN (RISC), que encuentra la secuencia de ARNm diana complementaria y escinde el ARN utilizando ARNasa. [17] Esto a su vez silencia el gen particular por interferencia de ARN. [18] Los siRNA y los miRNA difieren en el hecho de que los siRNA son típicamente específicos de la secuencia de ARNm mientras que los miRNA no son completamente complementarios a la secuencia de ARNm. Los miRNA pueden interactuar con objetivos que tienen secuencias similares, lo que inhibe la traducción de diferentes genes. [19] En general, la interferencia de ARN es una parte esencial de los procesos normales dentro de organismos como los humanos, y es un área que se está investigando como una herramienta diagnóstica y terapéutica para objetivos de cáncer. [16]

La degeneración macular relacionada con la edad es una causa importante de ceguera en los países desarrollados. El papel de Dicer en esta enfermedad se hizo evidente después de que se descubriera que los pacientes afectados mostraban niveles reducidos de Dicer en su epitelio pigmentario de la retina (RPE). Los ratones con Dicer knock out, que carecían de Dicer solo en su RPE, exhibieron síntomas similares. Sin embargo, otros ratones que carecían de proteínas importantes de la vía RNAi como Drosha y Pasha , no tuvieron síntomas de degeneración macular como los ratones con Dicer knock out. Esta observación sugirió un papel específico de Dicer en la salud de la retina que era independiente de la vía RNAi y, por lo tanto, no una función de la generación de si/miRNA. Se encontró que una forma de ARN llamada Alu RNA (las transcripciones de ARN de los elementos alu ) estaba elevada en pacientes con niveles insuficientes de Dicer. Estas hebras no codificantes de ARN pueden formar bucles de estructuras de dsRNA que serían degradadas por Dicer en una retina sana. Sin embargo, con niveles insuficientes de Dicer, la acumulación de ARN alu conduce a la degeneración del EPR como resultado de la inflamación. [20] [21]
Los perfiles de expresión de miRNA alterados en cánceres malignos sugieren un papel fundamental del miRNA y, por lo tanto, del dicer en el desarrollo y el pronóstico del cáncer. Los miRNA pueden funcionar como supresores tumorales y, por lo tanto, su expresión alterada puede dar lugar a la tumorogénesis . [22] En el análisis del cáncer de pulmón y ovario, el mal pronóstico y la disminución de los tiempos de supervivencia del paciente se correlacionan con la disminución de la expresión de dicer y drosha . Los niveles reducidos de ARNm de dicer se correlacionan con un estadio tumoral avanzado. Sin embargo, se ha demostrado que la alta expresión de dicer en otros cánceres, como el de próstata [23] y el de esófago, se correlaciona con un mal pronóstico del paciente. Esta discrepancia entre los tipos de cáncer sugiere que los procesos reguladores únicos de RNAi que involucran a dicer difieren entre los diferentes tipos de tumores. [16]
Dicer también está involucrado en la reparación del ADN . El daño del ADN aumenta en células de mamíferos con una menor expresión de Dicer como resultado de una menor eficiencia de la reparación del daño del ADN y otros mecanismos. Por ejemplo, el ARNi de las roturas de doble cadena (producido por Dicer) puede actuar como guías para los complejos proteicos involucrados en los mecanismos de reparación de roturas de doble cadena y también puede dirigir modificaciones de la cromatina . Además, los patrones de expresión de los miRNA cambian como resultado del daño del ADN causado por la radiación ionizante o ultravioleta . Los mecanismos de ARNi son responsables del silenciamiento de los transposones y en su ausencia, como cuando Dicer es eliminado/desactivado, pueden conducir a transposones activados que causan daño al ADN. La acumulación de daño al ADN puede dar lugar a células con mutaciones oncogénicas y, por lo tanto, al desarrollo de un tumor. [16]
Se ha demostrado que el bocio multinodular con schwannomatosis es una enfermedad autosómica dominante asociada con mutaciones en este gen. [24]
La infección por virus ARN puede desencadenar la cascada de RNAi. Es probable que dicer esté involucrado en la inmunidad viral , ya que los virus que infectan tanto a células vegetales como animales contienen proteínas diseñadas para inhibir la respuesta de RNAi. En humanos, los virus VIH-1 , influenza y vaccinia codifican dichas proteínas supresoras de RNAi. La inhibición de dicer es beneficiosa para el virus, ya que dicer puede escindir el dsRNA viral y cargar el producto en RISC, lo que resulta en la degradación dirigida del mRNA viral; combatiendo así la infección. Otro mecanismo potencial para la patogénesis viral es el bloqueo de dicer como una forma de inhibir las vías de miRNA celulares. [25]
En Drosophila , Dicer-1 genera microARN (miARN) procesando pre-miARN, Dicer-2 es responsable de producir pequeños ARN de interferencia (siARN) a partir de ARN bicatenario largo (dsARN). [26] Los insectos pueden usar Dicer como un potente antiviral . Este hallazgo es especialmente significativo dado que los mosquitos son responsables de la transmisión de muchas enfermedades virales, incluidos los arbovirus potencialmente mortales : virus del Nilo Occidental , dengue y fiebre amarilla . [27] Si bien los mosquitos, más específicamente la especie Aedes aegypti , sirven como vectores de estos virus, no son el huésped previsto del virus. La transmisión ocurre como resultado de la necesidad del mosquito hembra de sangre de vertebrados para desarrollar sus huevos. La vía de RNAi en insectos es muy similar a la de otros animales; Dicer-2 escinde el ARN viral y lo carga en el complejo RISC, donde una hebra sirve como plantilla para la producción de productos de ARNi y la otra se degrada. Los insectos con mutaciones que conducen a componentes no funcionales de su vía de ARNi muestran mayores cargas virales para los virus que portan o mayor susceptibilidad a los virus para los que son huéspedes. De manera similar a los humanos, los virus de insectos han desarrollado mecanismos para evitar la vía de ARNi. Como ejemplo, el virus C de Drosophila codifica la proteína 1A que se une al ARN de doble cadena, protegiéndolo así de la escisión de Dicer y de la carga de RISC. El ascovirus 3a de Heliothis virescens codifica una enzima ARNasa III similar a los dominios de ARNasa III de Dicer que puede competir por el sustrato de ARN de doble cadena y degradar los dúplex de ARNi para evitar la carga de RISC. [28]
Dicer se puede utilizar para identificar la presencia de tumores en el cuerpo en función del nivel de expresión de la enzima. Un estudio demostró que muchos pacientes con cáncer tenían niveles reducidos de expresión de Dicer. El mismo estudio demostró que una menor expresión de Dicer se correlacionaba con una menor supervivencia del paciente. [16] Además de ser una herramienta de diagnóstico , Dicer se puede utilizar para tratar a los pacientes inyectando ARNi extraño por vía intravenosa para provocar el silenciamiento del gen. [29]
Se ha demostrado que el ARNi se administra de dos maneras en especies de mamíferos como los ratones. Una forma sería inyectarlo directamente en el sistema, lo que no requeriría la función Dicer. Otra forma sería introducirlo mediante plásmidos que codifican ARN de horquilla corta, que Dicer corta para formar ARNi. [30]
Una de las ventajas de utilizar Dicer para producir ARNi terapéuticamente sería la especificidad y diversidad de objetivos a los que puede afectar en comparación con lo que se utiliza actualmente, como los anticuerpos o los inhibidores moleculares pequeños . En general, los inhibidores moleculares pequeños son difíciles en términos de especificidad junto con efectos secundarios insoportables. Los anticuerpos son tan específicos como el ARNi, pero están limitados al poder usarse solo contra ligandos o receptores de superficie . Por otro lado, la baja eficiencia de la captación intracelular es el principal obstáculo de la inyección de ARNi. [16] El ARNi inyectado tiene poca estabilidad en la sangre y provoca estimulaciones de la inmunidad no específica . [31] Además, la producción terapéutica de miARN carece de especificidad porque solo se requiere un apareamiento de bases de 6-8 nucleótidos para que el miARN se adhiera al ARNm. [32]
Los genomas de las plantas codifican proteínas similares a las de los dicer con funciones y dominios proteicos similares a los de los dicer de los animales y los insectos. Por ejemplo, en el organismo modelo Arabidopsis thaliana , se crean cuatro proteínas similares a las de los dicer, que se designan DCL1 a DCL4. DCL1 está implicada en la generación de miRNA y la producción de sRNA a partir de repeticiones invertidas. DCL2 crea siRNA a partir de transcripciones antisentido que actúan en cis, lo que ayuda a la inmunidad y defensa virales. DCL3 genera siRNA que ayuda a la modificación de la cromatina y DCL4 está implicado en el metabolismo de siRNA que actúa en trans y el silenciamiento de la transcripción a nivel postranscripcional. Además, DCL 1 y 3 son importantes para la floración de Arabidopsis . En Arabidopsis , la eliminación de DCL no causa problemas graves de desarrollo.
El arroz y las uvas también producen DCL, ya que el mecanismo de dicer es una estrategia de defensa común de muchos organismos. El arroz ha desarrollado otras funciones para los cinco DCL que produce y desempeñan un papel más importante en la función y el desarrollo que en Arabidopsis . Además, los patrones de expresión difieren entre los diferentes tipos de células vegetales del arroz, mientras que la expresión en Arabidopsis es más homogénea . La expresión de DCL en el arroz puede verse afectada por condiciones de estrés biológico, como la sequía, la salinidad y el frío. Por lo tanto, estos factores estresantes pueden disminuir la resistencia viral de una planta. A diferencia de Arabidopsis , la pérdida de función de las proteínas DCL causa defectos de desarrollo en el arroz. [33]