
La genómica comparada es un campo de investigación biológica en el que se comparan las características genómicas de diferentes organismos . [2] [3] Las características genómicas pueden incluir la secuencia de ADN , genes , orden de genes , secuencias reguladoras y otros puntos de referencia estructurales genómicos. [3] En esta rama de la genómica , se comparan partes enteras o grandes de genomas resultantes de proyectos genómicos para estudiar similitudes y diferencias biológicas básicas, así como las relaciones evolutivas entre organismos. [2] [4] [5] El principio principal de la genómica comparada es que las características comunes de dos organismos a menudo estarán codificadas dentro del ADN que se conserva evolutivamente entre ellos. [6] Por lo tanto, los enfoques genómicos comparativos comienzan con algún tipo de alineación de secuencias del genoma y buscando secuencias ortólogas (secuencias que comparten una ascendencia común ) en los genomas alineados y verificando hasta qué punto esas secuencias se conservan. A partir de estos, se infieren la evolución genómica y molecular y esto a su vez puede situarse en el contexto de, por ejemplo, la evolución fenotípica o la genética de poblaciones . [7]
Prácticamente comenzó tan pronto como en 1995 estuvieron disponibles los genomas completos de dos organismos (es decir, los genomas de las bacterias Haemophilus influenzae y Mycoplasma genitalium ), la genómica comparada es ahora un componente estándar del análisis de cada nueva secuencia genómica. [2] [8] Con la explosión en el número de proyectos genómicos debido a los avances en las tecnologías de secuenciación de ADN , particularmente los métodos de secuenciación de próxima generación a finales de la década de 2000, este campo se ha vuelto más sofisticado, haciendo posible tratar con muchos genomas. en un solo estudio. [9] La genómica comparada ha revelado altos niveles de similitud entre organismos estrechamente relacionados, como los humanos y los chimpancés , y, más sorprendentemente, similitud entre organismos aparentemente distantes, como los humanos y la levadura Saccharomyces cerevisiae . [4] También ha demostrado la extrema diversidad de la composición genética en diferentes linajes evolutivos. [8]
Ver también : Historia de la genómica
La genómica comparada tiene sus raíces en la comparación de genomas de virus a principios de la década de 1980. [8] Por ejemplo, se compararon pequeños virus de ARN que infectan animales ( picornavirus ) y aquellos que infectan plantas ( virus del mosaico del caupí ) y resultó que compartían una similitud de secuencia significativa y, en parte, el orden de sus genes. [10] En 1986, se publicó el primer estudio genómico comparativo a mayor escala, comparando los genomas del virus varicela-zóster y el virus de Epstein-Barr que contenían más de 100 genes cada uno. [11]
La primera secuencia genómica completa de un organismo celular, la de Haemophilus influenzae Rd, se publicó en 1995. [12] El segundo artículo sobre secuenciación del genoma fue el de la pequeña bacteria parásita Mycoplasma genitalium, publicado ese mismo año. [13] A partir de este artículo, los informes sobre nuevos genomas se convirtieron inevitablemente en estudios genómicos comparativos. [8]
Genomas microbianos. El primer sistema de comparación de genoma completo de alta resolución de genomas microbianos de 10-15 kbp fue desarrollado en 1998 por Art Delcher, Simon Kasif y Steven Salzberg y aplicado a la comparación de organismos microbianos completos altamente relacionados con sus colaboradores en el Instituto de Investigación Genómica ( TIGR). El sistema se llama MUMMER y se describió en una publicación en Nucleic Acids Research en 1999. El sistema ayuda a los investigadores a identificar grandes reordenamientos, mutaciones de una sola base, inversiones, expansiones de repeticiones en tándem y otros polimorfismos. En bacterias, MUMMER permite la identificación de polimorfismos responsables de la virulencia, la patogenicidad y la resistencia a los antibióticos. El sistema también se aplicó al Proyecto de Organismo Mínimo en TIGR y posteriormente a muchos otros proyectos de genómica comparada.
Genomas de eucariotas. Saccharomyces cerevisiae , la levadura del pan, fue el primer eucariota cuya secuencia genómica completa se publicó en 1996. [14] Después de la publicación del genoma del nematodo Caenorhabditis elegans en 1998 [15] y junto con el genoma de la mosca de la fruta Drosophila melanogaster en 2000, [16] Gerald M. Rubin y su equipo publicaron un artículo titulado "Genómica comparativa de los eucariotas", en el que compararon los genomas de los eucariotas D. melanogaster , C. elegans y S. cerevisiae , así como el procariota H. .influenzae . [17] Al mismo tiempo, Bonnie Berger , Eric Lander y su equipo publicaron un artículo sobre la comparación del genoma completo de humanos y ratones. [18]
Con la publicación de los grandes genomas de vertebrados en la década de 2000, incluidos los humanos , el pez globo japonés Takifugu rubripes y el ratón , se han publicado resultados precalculados de comparaciones de grandes genomas para su descarga o visualización en un navegador de genomas . En lugar de realizar sus propios análisis, la mayoría de los biólogos pueden acceder a estas grandes comparaciones entre especies y evitar la impracticabilidad causada por el tamaño de los genomas. [19]
Los métodos de secuenciación de próxima generación , que se introdujeron por primera vez en 2007, han producido una enorme cantidad de datos genómicos y han permitido a los investigadores generar múltiples borradores de secuencias genómicas (procarióticas) a la vez. Estos métodos también pueden descubrir rápidamente polimorfismos , inserciones y eliminaciones de un solo nucleótido al mapear lecturas no ensambladas con un genoma de referencia bien anotado y, por lo tanto, proporcionar una lista de posibles diferencias genéticas que pueden ser la base de cualquier variación funcional entre cepas. [9]
Una característica de la biología es la evolución, la teoría de la evolución es también la base teórica de la genómica comparada y, al mismo tiempo, los resultados de la genómica comparada enriquecieron y desarrollaron sin precedentes la teoría de la evolución. Cuando se comparan dos o más secuencias del genoma, se pueden deducir las relaciones evolutivas de las secuencias en un árbol filogenético. Basándose en una variedad de datos del genoma biológico y el estudio de los procesos de evolución vertical y horizontal, se pueden comprender partes vitales de la estructura genética y su función reguladora.
La similitud de genomas relacionados es la base de la genómica comparada. Si dos criaturas tienen un ancestro común reciente, las diferencias entre los genomas de las dos especies surgen del genoma de los ancestros. Cuanto más estrecha es la relación entre dos organismos, mayores son las similitudes entre sus genomas. Si existe una relación estrecha entre ellos, entonces su genoma mostrará un comportamiento lineal ( sínteno ), es decir, algunas o todas las secuencias genéticas se conservan. Por tanto, las secuencias del genoma se pueden utilizar para identificar la función genética, analizando su homología (similitud de secuencia) con genes de función conocida.

Las secuencias ortólogas son secuencias relacionadas en diferentes especies: existe un gen en la especie original, la especie se divide en dos especies, por lo que los genes de nuevas especies son ortólogos a la secuencia de la especie original. Las secuencias parálogas se separan mediante clonación de genes (duplicación de genes): si se copia un gen particular del genoma, entonces la copia de las dos secuencias es paráloga al gen original. Un par de secuencias ortólogas se denominan pares ortólogos (ortólogos), un par de secuencias parálogas se denominan pares colaterales (parálogos). Los pares ortólogos suelen tener la misma función o una similar, lo que no es necesariamente el caso de los pares colaterales. En pares colaterales, las secuencias tienden a evolucionar hasta tener diferentes funciones.
La genómica comparada explota tanto las similitudes como las diferencias en las proteínas , el ARN y las regiones reguladoras de diferentes organismos para inferir cómo ha actuado la selección sobre estos elementos. Aquellos elementos que son responsables de las similitudes entre diferentes especies deben conservarse a través del tiempo ( selección estabilizadora ), mientras que aquellos elementos responsables de las diferencias entre especies deben ser divergentes ( selección positiva ). Finalmente, aquellos elementos que no son importantes para el éxito evolutivo del organismo no se conservarán (la selección es neutral).
Uno de los objetivos importantes del campo es la identificación de los mecanismos de evolución del genoma eucariota. Sin embargo, a menudo se complica por la multiplicidad de acontecimientos que han tenido lugar a lo largo de la historia de cada linaje individual, dejando sólo huellas distorsionadas y superpuestas en el genoma de cada organismo vivo. Por esta razón, los estudios genómicos comparativos de pequeños organismos modelo (por ejemplo, el modelo Caenorhabditis elegans y el estrechamente relacionado Caenorhabditis briggsae ) son de gran importancia para avanzar en nuestra comprensión de los mecanismos generales de la evolución. [20] [21]
Los enfoques computacionales son necesarios para las comparaciones de genomas, dada la gran cantidad de datos codificados en los genomas. Actualmente hay muchas herramientas disponibles públicamente, que van desde comparaciones del genoma completo hasta análisis de la expresión genética . [22] Esto incluye enfoques de sistemas y control, teoría de la información, análisis de cadenas y minería de datos. [23] Los enfoques computacionales seguirán siendo críticos para la investigación y la enseñanza, especialmente cuando la ciencia de la información y la biología del genoma se enseñan en conjunto. [24]

La genómica comparada comienza con comparaciones básicas del tamaño del genoma y la densidad genética. Por ejemplo, el tamaño del genoma es importante para la capacidad de codificación y posiblemente por razones regulatorias. La alta densidad genética facilita la anotación del genoma y el análisis de la selección ambiental. Por el contrario, la baja densidad genética dificulta el mapeo de enfermedades genéticas como ocurre en el genoma humano.
Las alineaciones se utilizan para capturar información sobre secuencias similares, como ascendencia, ascendencia evolutiva común o estructura y función comunes. Se pueden realizar alineamientos tanto para secuencias genéticas como de proteínas. [26] [27] Las alineaciones consisten en alineaciones por pares locales o globales y alineaciones de secuencias múltiples. Una forma de encontrar alineaciones globales es utilizar un algoritmo de programación dinámica conocido como algoritmo Needleman-Wunsch . Este algoritmo se puede modificar y utilizar para encontrar alineaciones locales.

Otro método computacional para la genómica comparada es la reconstrucción filogenética. Se utiliza para describir relaciones evolutivas en términos de ancestros comunes. Las relaciones suelen estar representadas en un árbol llamado árbol filogenético . De manera similar, la teoría coalescente es un modelo retrospectivo para rastrear los alelos de un gen en una población hasta una única copia ancestral compartida por los miembros de la población. Este también se conoce como el ancestro común más reciente . El análisis basado en la teoría de la coalescencia intenta predecir la cantidad de tiempo entre la introducción de una mutación y un alelo o distribución genética particular en una población. Este período de tiempo es igual a cuánto tiempo hace que existió el ancestro común más reciente. Las relaciones de herencia se visualizan de forma similar a un árbol filogenético. La coalescencia (o la genealogía de los genes) se puede visualizar mediante dendrogramas . [28]
Un método adicional en genómica comparada es el mapeo genético . En el mapeo genético, visualizar la sintenia es una forma de ver el orden conservado de los genes en los cromosomas. Generalmente se utiliza para cromosomas de especies relacionadas, las cuales resultan de un ancestro común. [30] Este y otros métodos pueden arrojar luz sobre la historia evolutiva. Un estudio reciente utilizó genómica comparada para reconstruir 16 cariotipos ancestrales en la filogenia de los mamíferos. La reconstrucción computacional mostró cómo los cromosomas se reordenaron durante la evolución de los mamíferos. Brindó información sobre la conservación de regiones seleccionadas a menudo asociadas con el control de los procesos de desarrollo. Además, ayudó a comprender la evolución cromosómica y las enfermedades genéticas asociadas con los reordenamientos del ADN. [ cita necesaria ]

Las herramientas computacionales para analizar secuencias y genomas completos se están desarrollando rápidamente debido a la disponibilidad de una gran cantidad de datos genómicos. Al mismo tiempo, se avanzan y mejoran las herramientas de análisis comparativo. En los desafíos de estos análisis, es muy importante visualizar los resultados comparativos. [31]
La visualización de la conservación de secuencias es una tarea difícil del análisis de secuencias comparativo. Como sabemos, es muy ineficaz examinar manualmente la alineación de regiones genómicas largas. Los navegadores de genoma basados en Internet proporcionan muchas herramientas útiles para investigar secuencias genómicas debido a que integran toda la información biológica basada en secuencias en regiones genómicas. Cuando extraemos una gran cantidad de datos biológicos relevantes, pueden ser muy fáciles de usar y consumir menos tiempo. [31]
Una ventaja de utilizar herramientas en línea es que estos sitios web se desarrollan y actualizan constantemente. Hay muchas configuraciones nuevas y se puede utilizar contenido en línea para mejorar la eficiencia. [31]
La agricultura es un campo que cosecha los beneficios de la genómica comparada. Identificar los loci de genes ventajosos es un paso clave en el mejoramiento de cultivos optimizados para lograr un mayor rendimiento , rentabilidad, calidad y resistencia a las enfermedades . Por ejemplo, un estudio de asociación de todo el genoma realizado en 517 variedades locales de arroz reveló 80 loci asociados con varias categorías de desempeño agronómico, como el peso del grano, el contenido de amilosa y la tolerancia a la sequía . Muchos de los loci no estaban caracterizados previamente. [37] Esta metodología no sólo es poderosa, sino que también es rápida. Los métodos anteriores para identificar loci asociados con el desempeño agronómico requirieron varias generaciones de cría cuidadosamente monitoreada de cepas parentales, un esfuerzo que requiere mucho tiempo y que es innecesario para estudios genómicos comparativos. [38]
El campo de la medicina también se beneficia del estudio de la genómica comparada. En un enfoque conocido como vacunología inversa , los investigadores pueden descubrir antígenos candidatos para el desarrollo de vacunas analizando el genoma de un patógeno o una familia de patógenos. [39] La aplicación de un enfoque genómico comparativo mediante el análisis de los genomas de varios patógenos relacionados puede conducir al desarrollo de vacunas que sean multiprotectoras. Un equipo de investigadores empleó este enfoque para crear una vacuna universal para los estreptococos del grupo B , un grupo de bacterias responsables de infecciones neonatales graves . [40] La genómica comparada también se puede utilizar para generar especificidad para vacunas contra patógenos que están estrechamente relacionados con microorganismos comensales. Por ejemplo, los investigadores utilizaron análisis genómicos comparativos de cepas comensales y patógenas de E. coli para identificar genes específicos de patógenos como base para encontrar antígenos que den como resultado una respuesta inmune contra cepas patógenas pero no contra comensales. [41] En mayo de 2019, utilizando el Global Genome Set, un equipo del Reino Unido y Australia secuenció miles de aislados de estreptococos del grupo A recolectados a nivel mundial, lo que proporcionó objetivos potenciales para desarrollar una vacuna contra el patógeno, también conocido como S. pyogenes . [42]

Las células T (también conocidas como linfocitos T o timocitos) son células inmunitarias que crecen a partir de células madre en la médula ósea. Ayudan a defender el cuerpo de infecciones y pueden ayudar en la lucha contra el cáncer. Debido a su parecido morfológico, fisiológico y genético con los humanos, los ratones y las ratas han sido durante mucho tiempo las especies preferidas para los modelos animales de investigación biomédica . La investigación en medicina comparada se basa en la capacidad de utilizar información de una especie para comprender los mismos procesos en otra. Podemos obtener nuevos conocimientos sobre las vías moleculares comparando las células T humanas y de ratón y sus efectos en el sistema inmunológico utilizando genómica comparada. Para comprender sus TCR y sus genes, Glusman realizó una investigación sobre la secuenciación de los loci de los receptores de células T humanas y de ratón. Los genes TCR son bien conocidos y sirven como un recurso importante para respaldar la genómica funcional y comprender cómo los genes y las regiones intergénicas del genoma contribuyen a los procesos biológicos. [43]
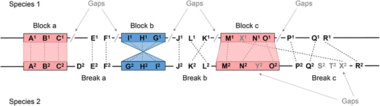
Los receptores inmunitarios de células T son importantes para ver el mundo de los patógenos en el sistema inmunológico celular. Una de las razones para secuenciar los loci TCR humanos y de ratón fue hacer coincidir las secuencias de la familia de genes ortólogos y descubrir áreas conservadas mediante genómica comparada. Se pensaba que éstos reflejarían dos tipos de información biológica: (1) exones y (2) secuencias reguladoras . De hecho, la mayoría de los exones V, D, J y C podrían identificarse con este método. Las regiones variables están codificadas por múltiples elementos de ADN únicos que se reorganizan y conectan durante la diferenciación de las células T (TCR): elementos variables (V), de diversidad (D) y de unión (J) para los polipéptidos y; y elementos V y J para los polipéptidos y. [Figura 1] Sin embargo, se han mostrado varios bloques cortos conservados no codificantes del genoma. Tanto los motivos humanos como los de ratón están agrupados en gran medida en los 200 pb [Figura 2], se identificaron los potenciadores 3 ' conocidos en el TCR/ y posteriormente se demostró que una región conservada de 100 pb en el intrón J del ratón tiene una función reguladora.

Las comparaciones de las secuencias genómicas dentro de cada sitio físico o ubicación de un gen específico en un cromosoma (locs) y entre especies permiten investigar otros mecanismos y otras señales reguladoras. Algunos sugieren nuevas hipótesis sobre la evolución de los TCR, que se probarán (y mejorarán) en comparación con el complemento de genes TCR de otras especies de vertebrados. Una investigación genómica comparativa de humanos y ratones obviamente permitirá el descubrimiento y la anotación de muchos otros genes, así como la identificación de secuencias reguladoras en otras especies. [43]
La genómica comparada también abre nuevas vías en otras áreas de investigación. A medida que la tecnología de secuenciación de ADN se ha vuelto más accesible, ha aumentado el número de genomas secuenciados . Con la creciente reserva de datos genómicos disponibles, la potencia de la inferencia genómica comparada también ha aumentado.
Un caso notable de este aumento de potencia se encuentra en investigaciones recientes con primates . Los métodos genómicos comparativos han permitido a los investigadores recopilar información sobre la variación genética , la expresión genética diferencial y la dinámica evolutiva en primates que era indiscernible utilizando datos y métodos anteriores. [44]
El Proyecto Genoma de los Grandes Simios utilizó métodos genómicos comparativos para investigar la variación genética con referencia a las seis especies de grandes simios , y encontró niveles saludables de variación en su acervo genético a pesar de la reducción del tamaño de la población. [45] Otro estudio demostró que los patrones de metilación del ADN, que son un mecanismo de regulación conocido para la expresión genética, difieren en la corteza prefrontal de los humanos frente a los chimpancés, e implicaron esta diferencia en la divergencia evolutiva de las dos especies. [46]