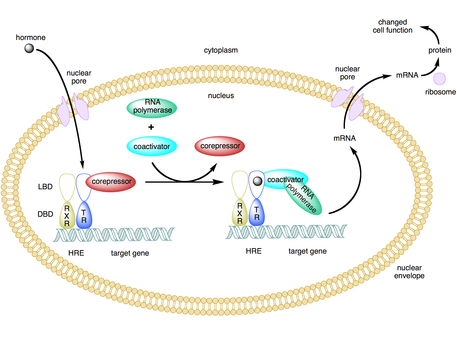
Un coactivador es un tipo de corregulador transcripcional que se une a un activador (un factor de transcripción ) para aumentar la tasa de transcripción de un gen o conjunto de genes. [1] El activador contiene un dominio de unión al ADN que se une a un sitio promotor del ADN o a una secuencia reguladora de ADN específica llamada potenciador . [2] [3] La unión del complejo activador-coactivador aumenta la velocidad de la transcripción al reclutar maquinaria de transcripción general al promotor, aumentando así la expresión genética . [3] [4] [5] El uso de activadores y coactivadores permite una expresión altamente específica de ciertos genes dependiendo del tipo de célula y la etapa de desarrollo. [2]
Algunos coactivadores también tienen actividad de histona acetiltransferasa (HAT). Las HAT forman grandes complejos multiproteicos que debilitan la asociación de las histonas al ADN al acetilar la cola N-terminal de la histona. Esto proporciona más espacio para que la maquinaria de transcripción se una al promotor, aumentando así la expresión génica. [1] [4]
Los activadores se encuentran en todos los organismos vivos , pero las proteínas coactivadoras normalmente solo se encuentran en eucariotas porque son más complejas y requieren un mecanismo más intrincado para la regulación genética. [1] [4] En eucariotas, los coactivadores suelen ser proteínas que se localizan en el núcleo. [1] [6]

Algunos coactivadores regulan indirectamente la expresión genética al unirse a un activador e inducir un cambio conformacional que luego permite que el activador se una a la secuencia promotora o potenciadora del ADN. [2] [7] [8] Una vez que el complejo activador-coactivador se une al potenciador, la ARN polimerasa II y otra maquinaria de transcripción general se reclutan en el ADN y comienza la transcripción. [9]
El ADN nuclear normalmente está envuelto firmemente alrededor de las histonas, lo que dificulta o imposibilita el acceso de la maquinaria de transcripción al ADN. Esta asociación se debe principalmente a la atracción electrostática entre el ADN y las histonas, ya que la cadena principal de fosfato del ADN tiene carga negativa y las histonas son ricas en residuos de lisina, que tienen carga positiva. [10] La estrecha asociación entre el ADN y las histonas impide la transcripción del ADN en ARN.
Muchos coactivadores tienen actividad de acetiltransferasa de histonas (HAT), lo que significa que pueden acetilar residuos de lisina específicos en las colas N-terminales de las histonas. [4] [7] [11] En este método, un activador se une a un sitio potenciador y recluta un complejo HAT que luego acetila las histonas unidas al promotor nucleosómico neutralizando los residuos de lisina cargados positivamente. [7] [11] Esta neutralización de carga hace que las histonas tengan un enlace más débil con el ADN cargado negativamente, lo que relaja la estructura de la cromatina, lo que permite que otros factores de transcripción o maquinaria de transcripción se unan al promotor (iniciación de la transcripción). [4] [11] La acetilación por complejos HAT también puede ayudar a mantener la cromatina abierta durante todo el proceso de elongación, lo que aumenta la velocidad de la transcripción. [4]

La acetilación de la cola de histona N-terminal es una de las modificaciones proteicas más comunes que se encuentran en los eucariotas, y aproximadamente el 85 % de todas las proteínas humanas están acetiladas. [12] La acetilación es crucial para la síntesis, estabilidad, función, regulación y localización de proteínas y transcripciones de ARN. [11] [12]
Las HAT funcionan de manera similar a las acetiltransferasas N-terminales (NAT), pero su acetilación es reversible a diferencia de las NAT. [13] La acetilación de histonas mediada por HAT se revierte utilizando la histona desacetilasa (HDAC), que cataliza la hidrólisis de los residuos de lisina, eliminando el grupo acetilo de las histonas. [4] [7] [11] Esto hace que la cromatina se vuelva a cerrar desde su estado relajado, lo que dificulta que la maquinaria de transcripción se una al promotor, reprimiendo así la expresión génica. [4] [7]
Los ejemplos de coactivadores que muestran actividad HAT incluyen CARM1 , CBP y EP300 . [14] [15]
Muchos coactivadores también funcionan como correpresores en determinadas circunstancias. [5] [9] Los cofactores como TAF1 y BTAF1 pueden iniciar la transcripción en presencia de un activador (actuar como coactivador) y reprimir la transcripción basal en ausencia de un activador (actuar como correpresor). [9]
La regulación transcripcional es una de las formas más comunes en que un organismo altera la expresión genética. [16] El uso de la activación y la coactivación permite un mayor control sobre cuándo, dónde y cuánta proteína se produce. [1] [7] [16] Esto permite que cada célula pueda responder rápidamente a los cambios ambientales o fisiológicos y ayuda a mitigar cualquier daño que pueda ocurrir si no se regula de otra manera. [1] [7]
Las mutaciones en genes coactivadores que conducen a la pérdida o ganancia de la función de la proteína se han relacionado con enfermedades y trastornos como defectos de nacimiento , cáncer (especialmente cánceres dependientes de hormonas), trastornos del desarrollo neurológico y discapacidad intelectual (DI), entre muchos otros. [17] [5] La desregulación que conduce a la sobreexpresión o subexpresión de coactivadores puede interactuar de manera perjudicial con muchos fármacos (especialmente fármacos antihormonales ) y se ha relacionado con el cáncer, problemas de fertilidad y trastornos neuropsiquiátricos y del desarrollo neurológico . [5] Como ejemplo específico, la desregulación de la proteína de unión a CREB (CBP), que actúa como coactivador de numerosos factores de transcripción dentro del sistema nervioso central (SNC), el sistema reproductivo, el timo y los riñones, se ha relacionado con la enfermedad de Huntington , la leucemia , el síndrome de Rubinstein-Taybi , los trastornos del desarrollo neurológico y los déficits del sistema inmunológico , la hematopoyesis y la función del músculo esquelético . [14] [18]
Los coactivadores son objetivos prometedores para terapias farmacológicas en el tratamiento del cáncer, trastornos metabólicos , enfermedades cardiovasculares y diabetes tipo 2 , junto con muchos otros trastornos. [5] [19] Por ejemplo, el coactivador del receptor de esteroides (SCR) NCOA3 a menudo se sobreexpresa en el cáncer de mama , por lo que el desarrollo de una molécula inhibidora que se dirija a este coactivador y disminuya su expresión podría usarse como un posible tratamiento para el cáncer de mama. [15] [20]
Debido a que los factores de transcripción controlan muchos procesos biológicos diferentes, son objetivos ideales para la terapia farmacológica. [14] [21] Los coactivadores que los regulan se pueden reemplazar fácilmente con un ligando sintético que permite controlar el aumento o la disminución de la expresión genética. [14]
Los avances tecnológicos futuros proporcionarán nuevos conocimientos sobre la función y regulación de los coactivadores a nivel de todo el organismo y dilucidarán su papel en las enfermedades humanas, lo que se espera proporcione mejores objetivos para futuras terapias farmacológicas. [14] [15]
Hasta la fecha se conocen más de 300 correguladores. [15] Algunos ejemplos de estos coactivadores incluyen: [22]