Un biosensor es un dispositivo analítico, utilizado para la detección de una sustancia química, que combina un componente biológico con un detector fisicoquímico . [1] [2] [3] [4] El elemento biológico sensible , por ejemplo tejido, microorganismos, orgánulos , receptores celulares , enzimas , anticuerpos , ácidos nucleicos , etc., es un material de origen biológico o componente biomimético que interactúa con, se une con o reconoce el analito en estudio. Los elementos biológicamente sensibles también pueden crearse mediante ingeniería biológica . El transductor o el elemento detector , que transforma una señal en otra, funciona de forma fisicoquímica: óptica, piezoeléctrica , electroquímica, electroquimioluminiscencia etc., resultante de la interacción del analito con el elemento biológico, para medir y cuantificar fácilmente. El dispositivo lector del biosensor se conecta con la electrónica asociada o procesadores de señales que son los principales responsables de la visualización de los resultados de forma sencilla para el usuario. [5] Esto a veces representa la parte más cara del dispositivo sensor, sin embargo es posible generar una pantalla fácil de usar que incluye un transductor y un elemento sensible ( sensor holográfico ). Los lectores suelen estar diseñados y fabricados a medida para adaptarse a los diferentes principios de funcionamiento de los biosensores.
Un biosensor consta típicamente de un biorreceptor (enzima/anticuerpo/célula/ácido nucleico/aptámero), un componente transductor (material semiconductor/nanomaterial) y un sistema electrónico que incluye un amplificador de señal , un procesador y una pantalla. [6] Los transductores y la electrónica se pueden combinar, por ejemplo, en sistemas de microsensores basados en CMOS . [7] [8] El componente de reconocimiento, a menudo llamado biorreceptor, utiliza biomoléculas de organismos o receptores modelados a partir de sistemas biológicos para interactuar con el analito de interés. Esta interacción se mide mediante el biotransductor que emite una señal medible proporcional a la presencia del analito objetivo en la muestra. El objetivo general del diseño de un biosensor es permitir una prueba rápida y conveniente en el punto de interés o atención donde se obtuvo la muestra. [1] [9] [10]
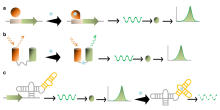
En un biosensor, el biorreceptor está diseñado para interactuar con el analito específico de interés para producir un efecto medible por el transductor. Una alta selectividad para el analito entre una matriz de otros componentes químicos o biológicos es un requisito clave del biorreceptor. Si bien el tipo de biomolécula utilizada puede variar ampliamente, los biosensores se pueden clasificar de acuerdo con los tipos comunes de interacciones de biorreceptores que involucran: anticuerpo/antígeno, [11] enzimas/ligandos, ácidos nucleicos/ADN, estructuras celulares/células o materiales biomiméticos. [12] [13]
Un inmunosensor utiliza la afinidad de unión muy específica de los anticuerpos para un compuesto o antígeno específico . La naturaleza específica de la interacción anticuerpo-antígeno es análoga a un ajuste de llave y cerradura en el que el antígeno solo se unirá al anticuerpo si tiene la conformación correcta. Los eventos de unión dan como resultado un cambio fisicoquímico que, en combinación con un trazador, como moléculas fluorescentes, enzimas o radioisótopos, puede generar una señal. Existen limitaciones con el uso de anticuerpos en sensores: 1. La capacidad de unión del anticuerpo depende en gran medida de las condiciones del ensayo (por ejemplo, pH y temperatura), y 2. la interacción anticuerpo-antígeno es generalmente robusta, sin embargo, la unión puede verse alterada por reactivos caotrópicos , solventes orgánicos o incluso radiación ultrasónica. [14] [15]
Las interacciones anticuerpo-antígeno también se pueden utilizar para las pruebas serológicas o la detección de anticuerpos circulantes en respuesta a una enfermedad específica. Es importante destacar que las pruebas serológicas se han convertido en una parte importante de la respuesta global a la pandemia de COVID-19 . [16]
El uso de anticuerpos como componente de bioreconocimiento de biosensores tiene varios inconvenientes. Tienen pesos moleculares altos y estabilidad limitada, contienen enlaces disulfuro esenciales y son costosos de producir. En un enfoque para superar estas limitaciones, se han diseñado fragmentos de unión recombinantes ( Fab , Fv o scFv ) o dominios (VH, VHH ) de anticuerpos. [17] En otro enfoque, se han diseñado pequeños andamios de proteínas con propiedades biofísicas favorables para generar familias artificiales de proteínas de unión a antígeno (AgBP), capaces de unirse específicamente a diferentes proteínas objetivo mientras conservan las propiedades favorables de la molécula original. Los elementos de la familia que se unen específicamente a un antígeno objetivo dado, a menudo se seleccionan in vitro mediante técnicas de visualización: visualización de fagos , visualización de ribosomas , visualización de levaduras o visualización de ARNm . Las proteínas de unión artificiales son mucho más pequeñas que los anticuerpos (normalmente menos de 100 residuos de aminoácidos), tienen una fuerte estabilidad, carecen de enlaces disulfuro y pueden expresarse con un alto rendimiento en entornos celulares reductores como el citoplasma bacteriano, al contrario de los anticuerpos y sus derivados. [18] [19] Por lo tanto, son especialmente adecuadas para crear biosensores. [20] [21]
Las capacidades de unión específicas y la actividad catalítica de las enzimas las convierten en biorreceptores populares. El reconocimiento de analitos se habilita a través de varios mecanismos posibles: 1) la enzima convierte el analito en un producto que es detectable por el sensor, 2) detecta la inhibición o activación enzimática por el analito, o 3) monitorea la modificación de las propiedades de la enzima resultante de la interacción con el analito. [15] Las principales razones para el uso común de enzimas en biosensores son: 1) capacidad para catalizar una gran cantidad de reacciones; 2) potencial para detectar un grupo de analitos (sustratos, productos, inhibidores y moduladores de la actividad catalítica); y 3) idoneidad con varios métodos de transducción diferentes para detectar el analito. En particular, dado que las enzimas no se consumen en las reacciones, el biosensor se puede usar fácilmente de forma continua. La actividad catalítica de las enzimas también permite límites de detección más bajos en comparación con las técnicas de unión comunes. Sin embargo, la vida útil del sensor está limitada por la estabilidad de la enzima.
Los anticuerpos tienen una constante de unión alta , superior a 10^8 L/mol, lo que representa una asociación casi irreversible una vez que se ha formado la pareja antígeno-anticuerpo. Para ciertas moléculas de analito, como la glucosa, existen proteínas de unión por afinidad que se unen a su ligando con una alta especificidad como un anticuerpo, pero con una constante de unión mucho menor, del orden de 10^2 a 10^4 L/mol. La asociación entre analito y receptor es entonces de naturaleza reversible y, junto a la pareja entre ambos, también sus moléculas libres se presentan en una concentración medible. En el caso de la glucosa, por ejemplo, la concanavalina A puede funcionar como receptor de afinidad que exhibe una constante de unión de 4x10^2 L/mol. [22] El uso de receptores de unión por afinidad para fines de biodetección ha sido propuesto por Schultz y Sims en 1979 [23] y posteriormente se configuró en un ensayo fluorescente para medir la glucosa en el rango fisiológico relevante entre 4,4 y 6,1 mmol/L. [24] El principio del sensor tiene la ventaja de que no consume el analito en una reacción química como ocurre en los ensayos enzimáticos.
Los biosensores que emplean receptores basados en ácidos nucleicos pueden basarse en interacciones de apareamiento de bases complementarias denominadas genosensores o imitadores de anticuerpos basados en ácidos nucleicos específicos (aptámeros) como aptasensores. [25] En el primero, el proceso de reconocimiento se basa en el principio de apareamiento de bases complementarias , adenina:timina y citosina:guanina en ADN . Si se conoce la secuencia de ácido nucleico diana, se pueden sintetizar secuencias complementarias, etiquetarlas y luego inmovilizarlas en el sensor. El evento de hibridación se puede detectar ópticamente y determinar la presencia de ADN/ARN diana. En el último, los aptámeros generados contra la diana lo reconocen a través de la interacción de interacciones no covalentes específicas y ajuste inducido. Estos aptámeros se pueden etiquetar con un fluoróforo/nanopartículas metálicas fácilmente para la detección óptica o se pueden emplear para plataformas de detección electroquímicas o basadas en voladizos sin etiqueta para una amplia gama de moléculas diana o dianas complejas como células y virus. [26] [27] Además, los aptámeros se pueden combinar con enzimas de ácidos nucleicos, como las ADNzimas que escinden ARN, lo que proporciona tanto reconocimiento de objetivos como generación de señales en una sola molécula, lo que muestra posibles aplicaciones en el desarrollo de biosensores multiplex. [28]
Se ha propuesto que los resonadores ópticos integrados optimizados adecuadamente se pueden explotar para detectar modificaciones epigenéticas (por ejemplo, metilación del ADN, modificaciones postraduccionales de histonas) en fluidos corporales de pacientes afectados por cáncer u otras enfermedades. [29] En la actualidad, se están desarrollando biosensores fotónicos con ultrasensibilidad a nivel de investigación para detectar fácilmente células cancerosas dentro de la orina del paciente. [30] Diferentes proyectos de investigación tienen como objetivo desarrollar nuevos dispositivos portátiles que utilicen cartuchos desechables económicos y respetuosos con el medio ambiente que solo requieran un manejo simple sin necesidad de procesamiento, lavado o manipulación adicionales por parte de técnicos expertos. [31]
Los orgánulos forman compartimentos separados dentro de las células y generalmente realizan funciones de forma independiente. Diferentes tipos de orgánulos tienen varias vías metabólicas y contienen enzimas para cumplir su función. Los orgánulos comúnmente utilizados incluyen lisosomas, cloroplastos y mitocondrias. El patrón de distribución espacio-temporal del calcio está estrechamente relacionado con la vía de señalización ubicua. Las mitocondrias participan activamente en el metabolismo de los iones de calcio para controlar la función y también modular las vías de señalización relacionadas con el calcio. Los experimentos han demostrado que las mitocondrias tienen la capacidad de responder a altas concentraciones de calcio generadas en su proximidad abriendo los canales de calcio. [32] De esta manera, las mitocondrias se pueden utilizar para detectar la concentración de calcio en el medio y la detección es muy sensible debido a la alta resolución espacial. Otra aplicación de las mitocondrias se utiliza para la detección de la contaminación del agua. La toxicidad de los compuestos detergentes dañará la célula y la estructura subcelular, incluidas las mitocondrias. Los detergentes causarán un efecto de hinchazón que podría medirse mediante un cambio de absorbancia. Los datos experimentales muestran que la tasa de cambio es proporcional a la concentración de detergente, lo que proporciona un alto estándar para la precisión de detección. [33]
Las células se utilizan a menudo en biorreceptores porque son sensibles al entorno circundante y pueden responder a todo tipo de estimulantes. Las células tienden a adherirse a la superficie para que puedan inmovilizarse fácilmente. En comparación con los orgánulos, permanecen activas durante un período más largo y la reproducibilidad las hace reutilizables. Se utilizan comúnmente para detectar parámetros globales como condiciones de estrés, toxicidad y derivados orgánicos. También se pueden utilizar para monitorear el efecto del tratamiento de medicamentos. Una aplicación es utilizar células para determinar los herbicidas que son los principales contaminantes acuáticos. [34] Las microalgas se atrapan en una microfibra de cuarzo y la fluorescencia de clorofila modificada por los herbicidas se recoge en la punta de un haz de fibras ópticas y se transmite a un fluorímetro. Las algas se cultivan continuamente para obtener una medición optimizada. Los resultados muestran que el límite de detección de ciertos herbicidas puede alcanzar un nivel de concentración inferior a ppb. Algunas células también se pueden utilizar para monitorear la corrosión microbiana. [35] Pseudomonas sp. se aísla de la superficie del material corroído y se inmoviliza en una membrana de acetilcelulosa. La actividad respiratoria se determina midiendo el consumo de oxígeno. Existe una relación lineal entre la corriente generada y la concentración de ácido sulfúrico . El tiempo de respuesta está relacionado con la carga de las células y el entorno circundante y se puede controlar a no más de 5 minutos.
Los tejidos se utilizan como biosensores para la abundancia de enzimas existentes. Las ventajas de los tejidos como biosensores incluyen las siguientes: [36]
También existen algunas desventajas de los tejidos, como la falta de especificidad debido a la interferencia de otras enzimas y un mayor tiempo de respuesta debido a la barrera de transporte.
Los biosensores microbianos aprovechan la respuesta de las bacterias a una sustancia determinada. Por ejemplo, el arsénico se puede detectar utilizando el operón ars presente en varios taxones bacterianos. [37]

Una parte importante de un biosensor es la fijación de los elementos biológicos (pequeñas moléculas/proteínas/células) a la superficie del sensor (ya sea de metal, polímero o vidrio). La forma más sencilla es funcionalizar la superficie para recubrirla con los elementos biológicos. Esto se puede hacer con polilisina, aminosilano, epoxisilano o nitrocelulosa en el caso de chips de silicio/vidrio de sílice. Posteriormente, el agente biológico unido también se puede fijar, por ejemplo, mediante la deposición capa por capa de recubrimientos de polímeros con carga alternativa. [38]
Como alternativa, se pueden utilizar redes tridimensionales ( hidrogel / xerogel ) para atraparlos química o físicamente (por lo que químicamente atrapado se entiende que el elemento biológico se mantiene en su lugar mediante un enlace fuerte, mientras que físicamente se mantiene en su lugar al no poder pasar a través de los poros de la matriz del gel). El hidrogel más comúnmente utilizado es el sol-gel , sílice vítrea generada por polimerización de monómeros de silicato (añadidos como ortosilicatos de tetraalquilo, como TMOS o TEOS ) en presencia de los elementos biológicos (junto con otros polímeros estabilizadores, como PEG ) en el caso de atrapamiento físico. [39]
Otro grupo de hidrogeles, que se fijan en condiciones adecuadas para las células o las proteínas, son los hidrogeles de acrilato , que se polimerizan tras la iniciación radicalaria . Un tipo de iniciador radical es un radical peróxido , normalmente generado mediante la combinación de un persulfato con TEMED ( los geles de poliacrilamida también se utilizan habitualmente para la electroforesis de proteínas ), [40] alternativamente, se puede utilizar luz en combinación con un fotoiniciador, como DMPA ( 2,2-dimetoxi-2-fenilacetofenona ). [41] Los materiales inteligentes que imitan los componentes biológicos de un sensor también se pueden clasificar como biosensores utilizando solo el sitio activo o catalítico o configuraciones análogas de una biomolécula. [42]

Los biosensores se pueden clasificar según el tipo de biotransductor que contienen. Los tipos de biotransductores más comunes que se utilizan en los biosensores son:
Los biosensores electroquímicos se basan normalmente en la catálisis enzimática de una reacción que produce o consume electrones (tales enzimas se denominan correctamente enzimas redox). El sustrato del sensor normalmente contiene tres electrodos : un electrodo de referencia , un electrodo de trabajo y un contraelectrodo. El analito objetivo está involucrado en la reacción que tiene lugar en la superficie del electrodo activo, y la reacción puede causar la transferencia de electrones a través de la doble capa (produciendo una corriente) o puede contribuir al potencial de doble capa (produciendo un voltaje). Podemos medir la corriente (la tasa de flujo de electrones ahora es proporcional a la concentración de analito) a un potencial fijo o el potencial puede medirse a corriente cero (esto da una respuesta logarítmica). Tenga en cuenta que el potencial del electrodo de trabajo o activo es sensible a la carga espacial y esto se usa a menudo. Además, la detección eléctrica directa y sin etiqueta de pequeños péptidos y proteínas es posible por sus cargas intrínsecas utilizando transistores de efecto de campo sensibles a iones biofuncionalizados . [43]
Otro ejemplo, el biosensor potenciométrico (potencial producido a corriente cero) da una respuesta logarítmica con un alto rango dinámico. Estos biosensores se fabrican a menudo mediante la serigrafía de los patrones de electrodos sobre un sustrato de plástico, recubierto con un polímero conductor y luego se le adhiere una proteína (enzima o anticuerpo). Tienen solo dos electrodos y son extremadamente sensibles y robustos. Permiten la detección de analitos a niveles que antes solo se podían lograr mediante HPLC y LC/MS y sin una preparación rigurosa de la muestra. Todos los biosensores suelen implicar una preparación mínima de la muestra, ya que el componente de detección biológica es altamente selectivo para el analito en cuestión. La señal se produce por cambios electroquímicos y físicos en la capa de polímero conductor debido a los cambios que ocurren en la superficie del sensor. Dichos cambios pueden atribuirse a la fuerza iónica, el pH, la hidratación y las reacciones redox, estas últimas debidas a la etiqueta enzimática que gira sobre un sustrato. [44] Los transistores de efecto de campo, en los que la región de compuerta se ha modificado con una enzima o anticuerpo, también pueden detectar concentraciones muy bajas de varios analitos, ya que la unión del analito a la región de compuerta del FET provoca un cambio en la corriente de drenaje-fuente.
El desarrollo de biosensores basados en espectroscopia de impedancia ha ido ganando terreno en la actualidad y muchos de estos dispositivos/desarrollos se encuentran en el ámbito académico y en la industria. Se ha demostrado que un dispositivo de este tipo, basado en una celda electroquímica de 4 electrodos que utiliza una membrana de alúmina nanoporosa, detecta bajas concentraciones de trombina alfa humana en presencia de un alto nivel de albúmina sérica. [45] También se han utilizado electrodos entrelazados para biosensores de impedancia. [46]


Se ha demostrado que el uso de canales iónicos ofrece una detección altamente sensible de moléculas biológicas objetivo. [47] Al incorporar los canales iónicos en membranas bicapa soportadas o ancladas (t-BLM) unidas a un electrodo de oro, se crea un circuito eléctrico. Las moléculas de captura, como los anticuerpos, se pueden unir al canal iónico de modo que la unión de la molécula objetivo controle el flujo de iones a través del canal. Esto da como resultado un cambio mensurable en la conducción eléctrica que es proporcional a la concentración del objetivo.
Se puede crear un biosensor de cambio de canal iónico (ICS) utilizando gramicidina, un canal peptídico dimérico, en una membrana bicapa anclada. [48] Un péptido de gramicidina, con anticuerpo unido, es móvil y el otro es fijo. La ruptura del dímero detiene la corriente iónica a través de la membrana. La magnitud del cambio en la señal eléctrica aumenta considerablemente al separar la membrana de la superficie metálica utilizando un espaciador hidrofílico.
Se ha demostrado la detección cuantitativa de una amplia clase de especies objetivo, incluidas proteínas, bacterias, fármacos y toxinas, utilizando diferentes configuraciones de membrana y captura. [49] [50] El proyecto de investigación europeo Greensense desarrolla un biosensor para realizar una detección cuantitativa de drogas de abuso como el THC, la morfina y la cocaína [51] en saliva y orina.
Un biosensor sin reactivos puede monitorizar un analito objetivo en una mezcla biológica compleja sin reactivo adicional. Por lo tanto, puede funcionar de forma continua si se inmoviliza sobre un soporte sólido. Un biosensor fluorescente reacciona a la interacción con su analito objetivo mediante un cambio de sus propiedades de fluorescencia. Un biosensor fluorescente sin reactivos (biosensor RF) se puede obtener integrando un receptor biológico, que se dirige contra el analito objetivo, y un fluoróforo solvatocrómico , cuyas propiedades de emisión son sensibles a la naturaleza de su entorno local, en una única macromolécula. El fluoróforo transduce el evento de reconocimiento en una señal óptica medible. El uso de fluoróforos extrínsecos, cuyas propiedades de emisión difieren ampliamente de las de los fluoróforos intrínsecos de las proteínas, el triptófano y la tirosina, permite detectar y cuantificar inmediatamente el analito en mezclas biológicas complejas. La integración del fluoróforo debe realizarse en un sitio donde sea sensible a la unión del analito sin perturbar la afinidad del receptor.
Los anticuerpos y las familias artificiales de proteínas de unión a antígenos (AgBP) son muy adecuados para proporcionar el módulo de reconocimiento de los biosensores de RF, ya que pueden dirigirse contra cualquier antígeno (véase el párrafo sobre biorreceptores). Se ha descrito un enfoque general para integrar un fluoróforo solvatocrómico en una AgBP cuando se conoce la estructura atómica del complejo con su antígeno, y así transformarlo en un biosensor de RF. [20] Se identifica un residuo de la AgBP en la vecindad del antígeno en su complejo. Este residuo se transforma en una cisteína mediante mutagénesis dirigida al sitio. El fluoróforo se acopla químicamente a la cisteína mutante. Cuando el diseño es exitoso, el fluoróforo acoplado no impide la unión del antígeno, esta unión protege al fluoróforo del solvente y puede detectarse mediante un cambio de fluorescencia. Esta estrategia también es válida para fragmentos de anticuerpos. [52] [53]
Sin embargo, en ausencia de datos estructurales específicos, se deben aplicar otras estrategias. Los anticuerpos y las familias artificiales de AgBPs están constituidas por un conjunto de posiciones de residuos hipervariables (o aleatorizadas), ubicadas en una única subregión de la proteína, y soportadas por un andamiaje polipeptídico constante. Los residuos que forman el sitio de unión para un antígeno dado, se seleccionan entre los residuos hipervariables. Es posible transformar cualquier AgBP de estas familias en un biosensor de RF, específico del antígeno diana, simplemente acoplando un fluoróforo solvatocrómico a uno de los residuos hipervariables que tienen poca o ninguna importancia para la interacción con el antígeno, después de convertir este residuo en cisteína por mutagénesis. Más específicamente, la estrategia consiste en convertir individualmente los residuos de las posiciones hipervariables en cisteína a nivel genético, en acoplar químicamente un fluoróforo solvatocrómico con la cisteína mutante, y luego en mantener los conjugados resultantes que tienen la mayor sensibilidad (un parámetro que involucra tanto la afinidad como la variación de la señal de fluorescencia). [21] Este enfoque también es válido para familias de fragmentos de anticuerpos. [54]
Estudios a posteriori han demostrado que los mejores biosensores fluorescentes sin reactivos se obtienen cuando el fluoróforo no realiza interacciones no covalentes con la superficie del biorreceptor, lo que aumentaría la señal de fondo, y cuando interactúa con un bolsillo de unión en la superficie del antígeno objetivo. [55] Los biosensores de RF que se obtienen mediante los métodos anteriores pueden funcionar y detectar analitos objetivo dentro de células vivas. [56]
Los biosensores magnéticos utilizan partículas paramagnéticas o supraparamagnéticas, o cristales, para detectar interacciones biológicas. Algunos ejemplos podrían ser la inductancia de la bobina, la resistencia u otras propiedades magnéticas. Es común utilizar nanopartículas o micropartículas magnéticas. En la superficie de dichas partículas se encuentran los biorreceptores, que pueden ser ADN (complementario a una secuencia o aptámeros), anticuerpos u otros. La unión del biorreceptor afectará algunas de las propiedades de las partículas magnéticas que se pueden medir mediante susceptometría de CA, [57] un sensor de efecto Hall, [58] un dispositivo de magnetorresistencia gigante, [59] u otros.
Los sensores piezoeléctricos utilizan cristales que sufren una deformación elástica cuando se les aplica un potencial eléctrico. Un potencial alterno (CA) produce una onda estacionaria en el cristal a una frecuencia característica. Esta frecuencia depende en gran medida de las propiedades elásticas del cristal, de modo que si un cristal está recubierto con un elemento de reconocimiento biológico, la unión de un analito objetivo (de gran tamaño) a un receptor producirá un cambio en la frecuencia de resonancia, lo que da una señal de unión. En un modo que utiliza ondas acústicas de superficie (SAW), la sensibilidad aumenta considerablemente. Esta es una aplicación especializada de la microbalanza de cristal de cuarzo como biosensor.
La electroquimioluminiscencia (ECL) es hoy en día una técnica líder en biosensores. [60] [61] [62] Dado que las especies excitadas se producen con un estímulo electroquímico en lugar de con una fuente de excitación luminosa, la ECL muestra una relación señal-ruido mejorada en comparación con la fotoluminiscencia, con efectos minimizados debido a la dispersión de la luz y al fondo de luminiscencia. En particular, la ECL correactiva que opera en una solución acuosa tamponada en la región de potenciales positivos (mecanismo de reducción oxidativa) impulsó definitivamente la ECL para el inmunoensayo, como lo confirman muchas aplicaciones de investigación y, aún más, por la presencia de importantes empresas que desarrollaron hardware comercial para análisis de inmunoensayos de alto rendimiento en un mercado que vale miles de millones de dólares cada año.
Los biosensores termométricos son raros.
El MOSFET inventado en Bell Labs entre 1955 y 1960, [63] [64 ] [65 ] [66] [67] [68] Más tarde, Leland C. Clark y Champ Lyons inventaron el primer biosensor en 1962. [69] [70] Los MOSFET biosensores (BioFET) se desarrollaron más tarde y desde entonces se han utilizado ampliamente para medir parámetros físicos , químicos , biológicos y ambientales . [71]
El primer BioFET fue el transistor de efecto de campo sensible a iones (ISFET), inventado por Piet Bergveld para aplicaciones electroquímicas y biológicas en 1970. [72] [73] el FET de adsorción (ADFET) fue patentado por PF Cox en 1974, y un MOSFET sensible al hidrógeno fue demostrado por I. Lundstrom, MS Shivaraman, CS Svenson y L. Lundkvist en 1975. [71] El ISFET es un tipo especial de MOSFET con una compuerta a una cierta distancia, [71] y donde la compuerta de metal se reemplaza por una membrana sensible a iones , una solución electrolítica y un electrodo de referencia . [74] El ISFET se usa ampliamente en aplicaciones biomédicas , como la detección de hibridación de ADN , detección de biomarcadores en sangre , detección de anticuerpos , medición de glucosa , detección de pH y tecnología genética . [74]
A mediados de la década de 1980, se habían desarrollado otros BioFET, incluido el FET de sensor de gas (GASFET), el FET de sensor de presión (PRESSFET), el transistor de efecto de campo químico (ChemFET), el ISFET de referencia (REFET), el FET modificado por enzimas (ENFET) y el FET modificado inmunológicamente (IMFET). [71] A principios de la década de 2000, se habían desarrollado BioFET como el transistor de efecto de campo de ADN (DNAFET), el FET modificado genéticamente (GenFET) y el BioFET de potencial celular (CPFET). [74]
La colocación adecuada de los biosensores depende de su campo de aplicación, que puede dividirse a grandes rasgos en biotecnología , agricultura , tecnología alimentaria y biomedicina .
En biotecnología, el análisis de la composición química del caldo de cultivo se puede realizar en línea, en línea, en línea y fuera de línea. Como lo describe la Administración de Alimentos y Medicamentos de los EE. UU. ( FDA ), la muestra no se retira del flujo de proceso para los sensores en línea, mientras que se desvía del proceso de fabricación para las mediciones en línea. Para los sensores en línea, la muestra se puede retirar y analizar muy cerca del flujo de proceso. [75] Un ejemplo de esto último es el monitoreo de la lactosa en una planta de procesamiento de lácteos. [76] Los biosensores fuera de línea se comparan con las técnicas bioanalíticas que no operan en el campo, sino en el laboratorio. Estas técnicas se utilizan principalmente en agricultura, tecnología alimentaria y biomedicina.
En aplicaciones médicas, los biosensores generalmente se clasifican como sistemas in vitro e in vivo . Una medición in vitro del biosensor se lleva a cabo en un tubo de ensayo, una placa de cultivo, una placa de microtitulación o en otro lugar fuera de un organismo vivo. El sensor utiliza un biorreceptor y un transductor como se describe anteriormente. Un ejemplo de un biosensor in vitro es un biosensor enzimático-conductimétrico para el control de la glucosa en sangre . Existe un desafío para crear un biosensor que funcione según el principio de prueba en el punto de atención , es decir, en el lugar donde se necesita la prueba. [77] [78] El desarrollo de biosensores portátiles se encuentra entre dichos estudios. [79] La eliminación de las pruebas de laboratorio puede ahorrar tiempo y dinero. Una aplicación de un biosensor POCT puede ser la prueba del VIH en áreas donde es difícil que los pacientes se hagan la prueba. Un biosensor se puede enviar directamente a la ubicación y se puede utilizar una prueba rápida y sencilla.

Un biosensor in vivo es un dispositivo implantable que funciona dentro del cuerpo. Por supuesto, los implantes de biosensores tienen que cumplir con las estrictas regulaciones sobre esterilización para evitar una respuesta inflamatoria inicial después de la implantación. La segunda preocupación se relaciona con la biocompatibilidad a largo plazo , es decir, la interacción inocua con el entorno corporal durante el período de uso previsto. [81] Otro problema que surge es el fallo. Si hay un fallo, el dispositivo debe retirarse y reemplazarse, lo que provoca una cirugía adicional. Un ejemplo de aplicación de un biosensor in vivo sería el control de la insulina dentro del cuerpo, que aún no está disponible.
Los implantes de biosensores más avanzados se han desarrollado para el control continuo de la glucosa. [82] [83] La figura muestra un dispositivo, para el cual se utiliza una carcasa de Ti y una batería como las establecidas para implantes cardiovasculares como marcapasos y desfibriladores . [80] Su tamaño está determinado por la batería, ya que se requiere para una vida útil de un año. Los datos de glucosa medidos se transmitirán de forma inalámbrica fuera del cuerpo dentro de la banda MICS 402-405 MHz, tal como se aprobó para implantes médicos.
Los biosensores también pueden integrarse en sistemas de telefonía móvil, haciéndolos fáciles de usar y accesibles para un gran número de usuarios. [84]

Existen muchas aplicaciones potenciales para los biosensores de distintos tipos. Los principales requisitos para que un método basado en biosensores sea valioso en términos de investigación y aplicaciones comerciales son la identificación de una molécula objetivo, la disponibilidad de un elemento de reconocimiento biológico adecuado y la posibilidad de que los sistemas de detección portátiles desechables sean preferibles a las técnicas sensibles de laboratorio en algunas situaciones. Algunos ejemplos son:
Un ejemplo común de un biosensor comercial es el biosensor de glucosa en sangre , que utiliza la enzima glucosa oxidasa para descomponer la glucosa en sangre. Para ello, primero oxida la glucosa y utiliza dos electrones para reducir el FAD (un componente de la enzima) a FADH 2 . Este, a su vez, es oxidado por el electrodo en una serie de pasos. La corriente resultante es una medida de la concentración de glucosa. En este caso, el electrodo es el transductor y la enzima es el componente biológicamente activo.
Un canario en una jaula , como los que utilizan los mineros para advertir de la presencia de gases, podría considerarse un biosensor. Muchas de las aplicaciones actuales de los biosensores son similares, ya que utilizan organismos que reaccionan ante sustancias tóxicas en concentraciones mucho más bajas que las que los humanos pueden detectar para advertir de su presencia. Dichos dispositivos se pueden utilizar en la vigilancia medioambiental , [86] la detección de trazas de gases y en instalaciones de tratamiento de agua.
Los monitores de glucosa disponibles comercialmente se basan en la detección amperométrica de la glucosa por medio de la glucosa oxidasa , que oxida la glucosa produciendo peróxido de hidrógeno que es detectado por el electrodo. Para superar la limitación de los sensores amperométricos, existe una oleada de investigaciones sobre nuevos métodos de detección, como los biosensores de glucosa fluorescentes . [88]
El sensor de imágenes por reflectancia interferométrica (IRIS) se basa en los principios de la interferencia óptica y consta de un sustrato de silicio-óxido de silicio, óptica estándar y LED coherentes de baja potencia. Cuando se ilumina la luz a través de un objetivo de bajo aumento sobre el sustrato de silicio-óxido de silicio en capas, se produce una firma interferométrica. A medida que la biomasa, que tiene un índice de refracción similar al óxido de silicio, se acumula en la superficie del sustrato, se produce un cambio en la firma interferométrica y el cambio se puede correlacionar con una masa cuantificable. Daaboul et al. utilizaron IRIS para producir una sensibilidad sin etiqueta de aproximadamente 19 ng/mL. [89] Ahn et al. mejoraron la sensibilidad de IRIS mediante una técnica de etiquetado de masa. [90]
Desde su publicación inicial, IRIS se ha adaptado para realizar varias funciones. En primer lugar, IRIS integró una capacidad de obtención de imágenes de fluorescencia en el instrumento de obtención de imágenes interferométricas como una forma potencial de abordar la variabilidad de los microarrays de proteínas de fluorescencia. [91] En resumen, la variación en los microarrays de fluorescencia se deriva principalmente de la inmovilización inconsistente de las proteínas en las superficies y puede causar diagnósticos erróneos en los microarrays de alergia. [92] Para corregir cualquier variación en la inmovilización de las proteínas, los datos adquiridos en la modalidad de fluorescencia se normalizan a continuación con los datos adquiridos en la modalidad sin etiqueta. [92] IRIS también se ha adaptado para realizar el recuento de nanopartículas individuales simplemente cambiando el objetivo de bajo aumento utilizado para la cuantificación de biomasa sin etiqueta a un objetivo de mayor aumento. [93] [94] Esta modalidad permite la discriminación de tamaño en muestras biológicas humanas complejas. Monroe et al. utilizaron IRIS para cuantificar los niveles de proteínas añadidos a la sangre completa y el suero humanos y determinaron la sensibilización a los alérgenos en muestras de sangre humana caracterizadas utilizando el procesamiento de muestra cero. [95] Otros usos prácticos de este dispositivo incluyen la detección de virus y patógenos. [96]
Existen diversas aplicaciones de los biosensores en el análisis de alimentos. [97] [98] [99] [100] En la industria alimentaria, se utilizan comúnmente ópticas recubiertas con anticuerpos para detectar patógenos y toxinas alimentarias. Comúnmente, el sistema de luz en estos biosensores es la fluorescencia, ya que este tipo de medición óptica puede amplificar en gran medida la señal.
Se ha desarrollado una gama de ensayos de unión de ligandos e inmunoensayos para la detección y medición de moléculas pequeñas, como vitaminas solubles en agua y contaminantes químicos ( residuos de fármacos ), como sulfonamidas y beta-agonistas , para su uso en sistemas de sensores basados en SPR , a menudo adaptados de ELISA existentes u otros ensayos inmunológicos. Estos se utilizan ampliamente en la industria alimentaria.
Los biosensores podrían utilizarse para monitorear contaminantes del aire , el agua y el suelo, como pesticidas, sustancias potencialmente cancerígenas, mutagénicas y/o tóxicas y productos químicos disruptores endocrinos. [101] [102]
Por ejemplo, los bionanotecnólogos desarrollaron un biosensor viable, ROSALIND 2.0 , que puede detectar niveles de diversos contaminantes del agua . [103] [104]
Debido a que el ozono filtra la radiación ultravioleta dañina, el descubrimiento de agujeros en la capa de ozono de la atmósfera terrestre ha suscitado preocupación sobre la cantidad de luz ultravioleta que llega a la superficie terrestre. Son de especial preocupación las cuestiones de hasta qué profundidad penetra la radiación ultravioleta en el agua del mar y cómo afecta a los organismos marinos , especialmente al plancton (microorganismos flotantes) y a los virus que atacan al plancton. El plancton forma la base de las cadenas alimentarias marinas y se cree que afecta a la temperatura y al clima de nuestro planeta mediante la absorción de CO2 para la fotosíntesis.
Deneb Karentz, investigadora del Laboratorio de Radiobiología y Salud Ambiental ( Universidad de California, San Francisco ), ha ideado un método sencillo para medir la penetración y la intensidad de la radiación ultravioleta. En el océano Antártico, sumergió a distintas profundidades unas bolsas de plástico delgadas que contenían cepas especiales de E. coli que son casi totalmente incapaces de reparar los daños causados por la radiación ultravioleta en su ADN. Se compararon las tasas de muerte bacteriana en estas bolsas con las tasas en bolsas de control no expuestas del mismo organismo. Los "biosensores" bacterianos revelaron un daño ultravioleta significativo y constante a profundidades de 10 m y, con frecuencia, a 20 y 30 m. Karentz planea realizar estudios adicionales sobre cómo la radiación ultravioleta puede afectar a las floraciones estacionales de plancton (brotes de crecimiento) en los océanos. [105]
La metástasis es la propagación del cáncer de una parte del cuerpo a otra a través del sistema circulatorio o del sistema linfático. [106] A diferencia de las pruebas de imagenología radiológica (mamografías), que envían formas de energía (rayos X, campos magnéticos, etc.) a través del cuerpo para tomar solo imágenes interiores, los biosensores tienen el potencial de probar directamente el poder maligno del tumor. La combinación de un elemento biológico y detector permite un pequeño requisito de muestra, un diseño compacto, señales rápidas, detección rápida, alta selectividad y alta sensibilidad para el analito que se está estudiando. En comparación con las pruebas de imagenología radiológica habituales, los biosensores tienen la ventaja no solo de descubrir cuánto se ha propagado el cáncer y verificar si el tratamiento es efectivo, sino que también son formas más económicas y eficientes (en tiempo, costo y productividad) de evaluar la metástasis en etapas tempranas del cáncer.
Los investigadores en ingeniería biológica han creado biosensores oncológicos para el cáncer de mama. [107] El cáncer de mama es el cáncer más común entre las mujeres en todo el mundo. [108] Un ejemplo sería una microbalanza de cristal de cuarzo (QCM) de transferrina. Como biosensor, las microbalanzas de cristal de cuarzo producen oscilaciones en la frecuencia de la onda estacionaria del cristal a partir de un potencial alterno para detectar cambios de masa de nanogramos. Estos biosensores están diseñados específicamente para interactuar y tienen una alta selectividad para los receptores en las superficies de las células (cancerosas y normales). Idealmente, esto proporciona una detección cuantitativa de células con este receptor por área de superficie en lugar de una detección de imagen cualitativa dada por las mamografías.
Seda Atay, investigadora de biotecnología en la Universidad Hacettepe, observó experimentalmente esta especificidad y selectividad entre un QCM y células de mama MDA-MB 231 , células MCF 7 y células MDA-MB 231 hambrientas in vitro. [107] Con otros investigadores, ideó un método para lavar estas células metastásicas de diferentes niveles sobre los sensores para medir los cambios de masa debido a diferentes cantidades de receptores de transferrina. En particular, el poder metastásico de las células de cáncer de mama se puede determinar mediante microbalanzas de cristal de cuarzo con nanopartículas y transferrina que potencialmente se unirían a los receptores de transferrina en las superficies de las células cancerosas. Existe una selectividad muy alta para los receptores de transferrina porque están sobreexpresados en las células cancerosas. Si las células tienen una alta expresión de receptores de transferrina, lo que muestra su alto poder metastásico, tienen mayor afinidad y se unen más al QCM que mide el aumento de masa. Dependiendo de la magnitud del cambio de masa en nanogramos, se puede determinar el poder metastásico.
Además, en los últimos años, se ha prestado una atención significativa a la detección de biomarcadores de cáncer de pulmón sin biopsia. En este sentido, los biosensores son herramientas muy atractivas y aplicables para proporcionar detecciones rápidas, sensibles, específicas, estables, rentables y no invasivas para el diagnóstico temprano del cáncer de pulmón. Por lo tanto, los biosensores de cáncer consisten en moléculas de biorreconocimiento específicas, como anticuerpos, sondas de ácidos nucleicos complementarios u otras biomoléculas inmovilizadas en la superficie de un transductor. Las moléculas de biorreconocimiento interactúan específicamente con los biomarcadores (objetivos) y las respuestas biológicas generadas son convertidas por el transductor en una señal analítica medible. Dependiendo del tipo de respuesta biológica, se utilizan varios transductores en la fabricación de biosensores de cáncer, como transductores electroquímicos, ópticos y basados en masa. [109]
Los biosensores podrían utilizarse para la detección de organismos patógenos. [102]
Se han desarrollado biosensores integrados para firmas patógenas, como el SARS-CoV-2 , que son portátiles , como mascarillas faciales con pruebas incorporadas . [110] [111] Véase también: I+D del transporte público contra la COVID-19
Los nuevos tipos de chips biosensores podrían permitir métodos novedosos, como sensores de patógenos desplegados por drones que supervisen activamente el aire o las aguas residuales. Los aptámeros de unión a proteínas podrían usarse para realizar pruebas de patógenos de enfermedades infecciosas. [112] Los sistemas de pieles electrónicas (o pieles robóticas) con biosensores (o sensores químicos) incorporados e interfaces hombre-máquina pueden permitir la detección de patógenos (así como de varios materiales peligrosos y percepciones táctiles ) mediante dispositivos portátiles o de detección remota o robóticos . [113] [ cita(s) adicional(es) necesaria(s) ]
Muchos biosensores ópticos se basan en el fenómeno de las técnicas de resonancia plasmónica de superficie (SPR). [114] [115] Esto utiliza una propiedad del oro y otros materiales (metales); [116] específicamente, que una capa delgada de oro sobre una superficie de vidrio de alto índice de refracción puede absorber la luz láser, produciendo ondas de electrones (plasmones de superficie) en la superficie del oro. Esto ocurre solo en un ángulo y longitud de onda específicos de luz incidente y depende en gran medida de la superficie del oro, de modo que la unión de un analito objetivo a un receptor en la superficie del oro produce una señal medible.
Los sensores de resonancia de plasmón superficial funcionan utilizando un chip sensor que consiste en un casete de plástico que soporta una placa de vidrio, uno de cuyos lados está recubierto con una capa microscópica de oro. Este lado entra en contacto con el aparato de detección óptica del instrumento. El lado opuesto se pone en contacto con un sistema de flujo microfluídico. El contacto con el sistema de flujo crea canales a través de los cuales se pueden pasar reactivos en solución. Este lado del chip sensor de vidrio se puede modificar de varias maneras para permitir la fácil adhesión de moléculas de interés. Normalmente está recubierto de carboximetil dextrano o un compuesto similar.
El índice de refracción en el lado de flujo de la superficie del chip tiene una influencia directa en el comportamiento de la luz reflejada por el lado dorado. La unión al lado de flujo del chip tiene un efecto en el índice de refracción y de esta manera las interacciones biológicas se pueden medir con un alto grado de sensibilidad con algún tipo de energía. El índice de refracción del medio cerca de la superficie cambia cuando las biomoléculas se adhieren a la superficie y el ángulo SPR varía en función de este cambio.
La luz de una longitud de onda fija se refleja en el lado dorado del chip en el ángulo de reflexión interna total y se detecta dentro del instrumento. El ángulo de la luz incidente se varía para que coincida con la velocidad de propagación de la onda evanescente con la velocidad de propagación de los polaritones plasmónicos de la superficie. [117] Esto induce a la onda evanescente a penetrar a través de la placa de vidrio y cierta distancia dentro del líquido que fluye sobre la superficie.
Otros biosensores ópticos se basan principalmente en cambios en la absorbancia o fluorescencia de un compuesto indicador apropiado y no necesitan una geometría de reflexión interna total. Por ejemplo, se ha fabricado un prototipo de dispositivo totalmente operativo que detecta caseína en la leche. El dispositivo se basa en la detección de cambios en la absorción de una capa de oro. [118] Una herramienta de investigación ampliamente utilizada, la micromatriz, también puede considerarse un biosensor.
Los biosensores biológicos, también conocidos como sensores optogenéticos , a menudo incorporan una forma genéticamente modificada de una proteína o enzima nativa. La proteína está configurada para detectar un analito específico y la señal resultante es leída por un instrumento de detección como un fluorómetro o luminómetro. Un ejemplo de un biosensor desarrollado recientemente es uno para detectar la concentración citosólica del analito cAMP (monofosfato de adenosina cíclico), un segundo mensajero involucrado en la señalización celular desencadenada por ligandos que interactúan con receptores en la membrana celular. [119] Se han creado sistemas similares para estudiar las respuestas celulares a ligandos nativos o xenobióticos (toxinas o inhibidores de moléculas pequeñas). Tales "ensayos" se utilizan comúnmente en el desarrollo del descubrimiento de fármacos por parte de empresas farmacéuticas y biotecnológicas. La mayoría de los ensayos de cAMP en uso actual requieren la lisis de las células antes de la medición de cAMP. Un biosensor de células vivas para cAMP se puede utilizar en células no lisadas con la ventaja adicional de múltiples lecturas para estudiar la cinética de la respuesta del receptor.
Los nanobiosensores utilizan una sonda biorreceptora inmovilizada que es selectiva para las moléculas de analito objetivo. Los nanomateriales son sensores químicos y biológicos sumamente sensibles. Los materiales a escala nanométrica presentan propiedades únicas. Su gran relación área-volumen permite lograr reacciones rápidas y de bajo costo, utilizando una variedad de diseños. [120]
Se han comercializado otros biosensores de ondas evanescentes que utilizan guías de ondas en las que la constante de propagación a través de la guía de ondas cambia con la absorción de moléculas en su superficie. Un ejemplo de ello es la interferometría de polarización dual , que utiliza una guía de ondas enterrada como referencia con la que se mide el cambio en la constante de propagación. Otras configuraciones, como la de Mach-Zehnder, tienen brazos de referencia definidos litográficamente sobre un sustrato. Se pueden lograr niveles más altos de integración utilizando geometrías de resonador en las que la frecuencia de resonancia de un resonador de anillo cambia cuando se absorben moléculas. [121] [122]
Recientemente, se han aplicado conjuntos de muchas moléculas detectoras diferentes en los llamados dispositivos de nariz electrónica , donde el patrón de respuesta de los detectores se utiliza para identificar una sustancia. [123] En el detector de olores Wasp Hound , el elemento mecánico es una cámara de vídeo y el elemento biológico son cinco avispas parásitas que han sido condicionadas a agruparse en respuesta a la presencia de una sustancia química específica. [124] Sin embargo, las narices electrónicas comerciales actuales no utilizan elementos biológicos.
El ADN puede ser el analito de un biosensor, siendo detectado a través de medios específicos, pero también puede utilizarse como parte de un biosensor o, teóricamente, incluso como un biosensor completo.
Existen muchas técnicas para detectar ADN, que normalmente es un medio para detectar organismos que tienen ese ADN en particular. También se pueden utilizar secuencias de ADN como se describió anteriormente. Pero existen enfoques más avanzados, en los que se puede sintetizar ADN para contener enzimas en un gel biológico estable. [125] Otras aplicaciones son el diseño de aptámeros, secuencias de ADN que tienen una forma específica para unirse a una molécula deseada. Los procesos más innovadores utilizan origami de ADN para esto, creando secuencias que se pliegan en una estructura predecible que es útil para la detección. [126] [127]
Los científicos han construido prototipos de sensores para detectar el ADN de los animales a partir del aire aspirado, el "ADN transportado por el aire". [128]
Las "nanoantenas" hechas de ADN –un nuevo tipo de antena óptica a escala nanométrica– pueden unirse a las proteínas y producir una señal a través de fluorescencia cuando estas realizan sus funciones biológicas, en particular para cambios conformacionales específicos . [129] [130]
El grafeno es una sustancia bidimensional basada en carbono con propiedades ópticas, eléctricas, mecánicas, térmicas y mecánicas superiores. La capacidad de absorber e inmovilizar una variedad de proteínas, particularmente algunas con estructuras de anillo de carbono, ha demostrado que el grafeno es un excelente candidato como transductor de biosensores. Como resultado, se han explorado y desarrollado varios biosensores basados en grafeno en los últimos tiempos. [14] [131] El grafeno se ha empleado como biosensor en varios formatos, especialmente sensores electroquímicos y transistores de efecto de campo. Entre ellos, los transistores de efecto de campo de grafeno (GFET) en particular han demostrado un excelente rendimiento como diagnósticos rápidos en el punto de atención (PoC), como se observa a través de un aumento en el número de artículos de investigación que informan sobre diagnósticos de COVID-19 utilizando GFET. Se ha informado que tienen uno de los límites de detección más bajos, al mismo tiempo que tienen un tiempo de respuesta rápido de unos pocos segundos junto con capacidades de multiplexación. [132] Estas capacidades permiten la detección inmediata de enfermedades, especialmente en casos con síntomas superpuestos que son difíciles de distinguir al principio, lo que permite obtener mejores resultados para los pacientes, especialmente en entornos médicos con recursos limitados.
{{cite web}}: CS1 maint: copia archivada como título ( enlace )(consultado el 30 de enero de 2013).