
La biología de sistemas es el análisis y modelado computacional y matemático de sistemas biológicos complejos . Es un campo de estudio interdisciplinario basado en la biología que se centra en las interacciones complejas dentro de los sistemas biológicos, utilizando un enfoque holístico ( holismo en lugar del reduccionismo más tradicional ) para la investigación biológica. [1]
En particular, a partir del año 2000, el concepto se ha utilizado ampliamente en biología en una variedad de contextos. El Proyecto Genoma Humano es un ejemplo de pensamiento sistémico aplicado en biología que ha llevado a nuevas formas colaborativas de trabajar en problemas en el campo biológico de la genética. [2] Uno de los objetivos de la biología de sistemas es modelar y descubrir propiedades emergentes , propiedades de células , tejidos y organismos que funcionan como un sistema cuya descripción teórica solo es posible utilizando técnicas de biología de sistemas. [1] [3] Estas típicamente involucran redes metabólicas o redes de señalización celular . [1] [4]
La biología de sistemas puede considerarse desde varios aspectos diferentes.
Como campo de estudio, en particular, el estudio de las interacciones entre los componentes de los sistemas biológicos, y cómo estas interacciones dan lugar a la función y comportamiento de ese sistema (por ejemplo, las enzimas y metabolitos en una vía metabólica o los latidos del corazón). [5] [6] [7]
Como paradigma , la biología de sistemas suele definirse en antítesis del llamado paradigma reduccionista ( organización biológica ), aunque es coherente con el método científico . La distinción entre ambos paradigmas se hace referencia en estas citas: "el enfoque reduccionista ha identificado con éxito la mayoría de los componentes y muchas de las interacciones pero, desafortunadamente, no ofrece conceptos ni métodos convincentes para entender cómo surgen las propiedades del sistema... el pluralismo de causas y efectos en las redes biológicas se aborda mejor observando, mediante medidas cuantitativas, múltiples componentes simultáneamente y mediante una integración rigurosa de datos con modelos matemáticos". (Sauer et al. ) [8] "La biología de sistemas... trata de unir en lugar de separar, de integrar en lugar de reducir. Requiere que desarrollemos formas de pensar sobre la integración que sean tan rigurosas como nuestros programas reduccionistas, pero diferentes... Significa cambiar nuestra filosofía, en el pleno sentido del término". ( Denis Noble ) [7]
Como una serie de protocolos operativos utilizados para realizar investigaciones, es decir, un ciclo compuesto por teoría, modelado analítico o computacional para proponer hipótesis específicas comprobables sobre un sistema biológico, validación experimental y luego usar la descripción cuantitativa recién adquirida de células o procesos celulares para refinar el modelo computacional o teoría. [9] Dado que el objetivo es un modelo de las interacciones en un sistema, las técnicas experimentales que más se adaptan a la biología de sistemas son aquellas que abarcan todo el sistema e intentan ser lo más completas posible. Por lo tanto, se utilizan la transcriptómica , la metabolómica , la proteómica y las técnicas de alto rendimiento para recopilar datos cuantitativos para la construcción y validación de modelos. [10]
Como la aplicación de la teoría de los sistemas dinámicos a la biología molecular .
Como un fenómeno sociocientífico definido por la estrategia de buscar la integración de datos complejos sobre las interacciones en sistemas biológicos de diversas fuentes experimentales utilizando herramientas y personal interdisciplinarios. [12]
Aunque el concepto de una visión sistémica de la función celular se ha entendido bien desde al menos la década de 1930, [13] las limitaciones tecnológicas hicieron que fuera difícil hacer mediciones de todo el sistema. La llegada de la tecnología de microarrays en la década de 1990 abrió una nueva vía para estudiar las células a nivel de sistemas. En 2000, se estableció el Instituto de Biología de Sistemas en Seattle en un esfuerzo por atraer a personas de tipo "computacional" que se pensaba que no se sentían atraídas por los entornos académicos de la universidad. El instituto no tenía una definición clara de lo que realmente era el campo: reunir a personas de diversos campos para usar computadoras para estudiar la biología de manera holística de nuevas maneras. [14] En 2003 se lanzó un Departamento de Biología de Sistemas en la Facultad de Medicina de Harvard. [15] En 2006 se predijo que el revuelo generado por el nuevo concepto "muy de moda" haría que todas las universidades importantes necesitaran un departamento de biología de sistemas, por lo que habría carreras disponibles para graduados con un mínimo de habilidad en programación informática y biología. [14] En 2006, la National Science Foundation propuso el reto de construir un modelo matemático de la célula completa. [ cita requerida ] En 2012, el Laboratorio Covert de la Universidad de Stanford logró el primer modelo de célula completa de Mycoplasma genitalium . El modelo de célula completa es capaz de predecir la viabilidad de las células de M. genitalium en respuesta a mutaciones genéticas. [16]
Un precursor temprano de la biología de sistemas, como disciplina distinta, puede haber sido el teórico de sistemas Mihajlo Mesarovic en 1966 con un simposio internacional en el Instituto Case de Tecnología en Cleveland , Ohio, titulado Teoría de sistemas y biología . Mesarovic predijo que tal vez en el futuro existiría algo así como "biología de sistemas". [17] [18] Otros precursores tempranos que se centraron en la visión de que la biología debe analizarse como un sistema, en lugar de una simple colección de partes, fueron el Análisis de control metabólico , desarrollado por Henrik Kacser y Jim Burns [19] posteriormente revisado a fondo, [20] y Reinhart Heinrich y Tom Rapoport , [21] y la Teoría de sistemas bioquímicos desarrollada por Michael Savageau [22] [23] [24]
Según Robert Rosen , en la década de 1960, la biología holística había pasado de moda a principios del siglo XX, a medida que se popularizaba una ciencia más empírica dominada por la química molecular. [18] Haciéndose eco de sus palabras cuarenta años después, en 2006, Kling escribe que el éxito de la biología molecular a lo largo del siglo XX había suprimido los métodos computacionales holísticos. [14] En 2011, los Institutos Nacionales de Salud habían puesto a disposición dinero de subvención para apoyar a más de diez centros de biología de sistemas en los Estados Unidos, [25] pero en 2012 Hunter escribe que la biología de sistemas todavía tiene mucho camino por recorrer para alcanzar su máximo potencial. No obstante, los defensores esperaban que alguna vez pudiera resultar más útil en el futuro. [26]
Un hito importante en el desarrollo de la biología de sistemas ha sido el proyecto internacional Physiome . [ cita requerida ]
Según la interpretación de la biología de sistemas como el uso de grandes conjuntos de datos mediante herramientas interdisciplinarias, una aplicación típica es la metabolómica , que es el conjunto completo de todos los productos metabólicos, metabolitos , en el sistema a nivel de organismo, célula o tejido. [28]
Los elementos que pueden ser una base de datos informática incluyen: fenómica , variación organismal en el fenotipo a medida que cambia durante su vida útil; genómica , secuencia de ácido desoxirribonucleico (ADN) organismal , incluida la variación específica de la célula intraorganismo (es decir, variación de la longitud de los telómeros ); epigenómica / epigenética , factores reguladores transcriptómicos específicos del organismo y de la célula correspondiente no codificados empíricamente en la secuencia genómica (es decir, metilación del ADN , acetilación y desacetilación de histonas , etc.); transcriptómica , mediciones de la expresión génica del organismo, tejido o célula completa mediante microarreglos de ADN o análisis en serie de la expresión génica ; interferómica , factores correctores de transcripción a nivel de organismo, tejido o célula (es decir, interferencia de ARN ), proteómica , mediciones a nivel de organismo, tejido o célula de proteínas y péptidos mediante electroforesis en gel bidimensional , espectrometría de masas o técnicas de identificación de proteínas multidimensionales ( sistemas HPLC avanzados acoplados a espectrometría de masas ). Las subdisciplinas incluyen fosfoproteómica , glicoproteómica y otros métodos para detectar proteínas modificadas químicamente; glicomica , mediciones a nivel de organismo, tejido o célula de carbohidratos ; lipidómica , mediciones a nivel de organismo, tejido o célula de lípidos . [ cita requerida ]
También se estudian las interacciones moleculares dentro de la célula, a esto se le llama interactómica . [29] Una disciplina en este campo de estudio son las interacciones proteína-proteína , aunque la interactómica incluye las interacciones de otras moléculas. [ cita requerida ] Neuroelectrodinámica , donde se estudia la función computacional de la computadora o del cerebro como un sistema dinámico junto con sus mecanismos (bio)físicos; [30] y fluxómica , mediciones de las tasas de reacciones metabólicas en un sistema biológico (célula, tejido u organismo). [28]
Para abordar un problema de biología de sistemas existen dos enfoques principales: el enfoque de arriba hacia abajo y el de abajo hacia arriba. El enfoque de arriba hacia abajo tiene en cuenta la mayor parte posible del sistema y se basa en gran medida en resultados experimentales. La técnica RNA-Seq es un ejemplo de un enfoque experimental de arriba hacia abajo. Por el contrario, el enfoque de abajo hacia arriba se utiliza para crear modelos detallados a la vez que se incorporan datos experimentales. Un ejemplo del enfoque de abajo hacia arriba es el uso de modelos de circuitos para describir una red genética simple. [31]
Diversas tecnologías utilizadas para capturar cambios dinámicos en el ARNm, las proteínas y las modificaciones postraduccionales. Mecanobiología , fuerzas y propiedades físicas en todas las escalas, su interacción con otros mecanismos reguladores; [32] biosemiótica , análisis del sistema de relaciones de signos de un organismo u otros biosistemas; Fisiómica , un estudio sistemático del fisioma en biología.
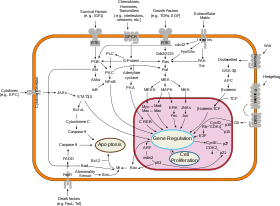
La biología de sistemas del cáncer es un ejemplo del enfoque de la biología de sistemas, que se puede distinguir por el objeto específico de estudio ( tumorigénesis y tratamiento del cáncer ). Trabaja con datos específicos (muestras de pacientes, datos de alto rendimiento con especial atención a la caracterización del genoma del cáncer en muestras de tumores de pacientes) y herramientas ( líneas celulares de cáncer inmortalizadas , modelos de ratón de tumorigénesis, modelos de xenoinjerto , métodos de secuenciación de alto rendimiento , derribo de genes basado en ARNi en pruebas de alto rendimiento , modelado computacional de las consecuencias de las mutaciones somáticas y la inestabilidad del genoma ). [33] El objetivo a largo plazo de la biología de sistemas del cáncer es la capacidad de diagnosticar mejor el cáncer, clasificarlo y predecir mejor el resultado de un tratamiento sugerido, que es una base para la medicina personalizada del cáncer y el paciente virtual con cáncer en una perspectiva más lejana. Se han realizado esfuerzos significativos en la biología de sistemas computacionales del cáncer para crear modelos in silico realistas a múltiples escalas de varios tumores. [34]
El enfoque de la biología de sistemas a menudo implica el desarrollo de modelos mecanicistas , como la reconstrucción de sistemas dinámicos a partir de las propiedades cuantitativas de sus bloques de construcción elementales. [35] [36] [37] [38] Por ejemplo, una red celular se puede modelar matemáticamente utilizando métodos provenientes de la cinética química [39] y la teoría de control . Debido a la gran cantidad de parámetros, variables y restricciones en las redes celulares, a menudo se utilizan técnicas numéricas y computacionales (por ejemplo, análisis de balance de flujo ). [37] [39]
Otros aspectos de la informática, la informática y la estadística también se utilizan en la biología de sistemas. Estos incluyen nuevas formas de modelos computacionales, como el uso de cálculos de procesos para modelar procesos biológicos (los enfoques notables incluyen el cálculo π estocástico , BioAmbients, Beta Binders, BioPEPA y cálculo de branas) y el modelado basado en restricciones ; integración de información de la literatura, utilizando técnicas de extracción de información y minería de texto ; [40] desarrollo de bases de datos y repositorios en línea para compartir datos y modelos, enfoques para la integración de bases de datos e interoperabilidad de software mediante el acoplamiento flexible de software, sitios web y bases de datos, o trajes comerciales; enfoques basados en redes para analizar conjuntos de datos genómicos de alta dimensión. Por ejemplo, el análisis de redes de correlación ponderada se utiliza a menudo para identificar clústeres (denominados módulos), modelar la relación entre clústeres, calcular medidas difusas de pertenencia a clústeres (módulos), identificar centros intramodulares y para estudiar la preservación de clústeres en otros conjuntos de datos; Métodos basados en vías para el análisis de datos ómicos, por ejemplo, enfoques para identificar y puntuar vías con actividad diferencial de sus genes, proteínas o metabolitos. [41] Gran parte del análisis de conjuntos de datos genómicos también incluye la identificación de correlaciones. Además, como gran parte de la información proviene de diferentes campos, es necesario el desarrollo de formas sintáctica y semánticamente sólidas de representar modelos biológicos. [42]

Los investigadores comienzan eligiendo una vía biológica y diagramando todas las vías de proteínas, genes y/o metabólicas. Después de determinar todas las interacciones, se utilizan leyes de velocidad cinética de acción de masas o de enzimas para describir la velocidad de las reacciones en el sistema. Utilizando la conservación de masa, se pueden construir las ecuaciones diferenciales para el sistema biológico. Se pueden realizar experimentos o ajustes de parámetros para determinar los valores de los parámetros que se utilizarán en las ecuaciones diferenciales . [44] Estos valores de parámetros serán las diversas constantes cinéticas necesarias para describir completamente el modelo. Este modelo determina el comportamiento de las especies en los sistemas biológicos y aporta nuevos conocimientos sobre las actividades específicas del sistema. A veces no es posible recopilar todas las velocidades de reacción de un sistema. Las velocidades de reacción desconocidas se determinan simulando el modelo de parámetros conocidos y el comportamiento objetivo que proporciona posibles valores de parámetros. [45] [43]
El uso de métodos de reconstrucción y análisis basados en restricciones (COBRA) se ha vuelto popular entre los biólogos de sistemas para simular y predecir los fenotipos metabólicos, utilizando modelos a escala del genoma. Uno de los métodos es el enfoque de análisis de balance de flujo (FBA), mediante el cual se pueden estudiar las redes bioquímicas y analizar el flujo de metabolitos a través de una red metabólica particular, optimizando la función objetivo de interés (por ejemplo, maximizando la producción de biomasa para predecir el crecimiento). [46]

{{cite journal}}: Requiere citar revista |journal=( ayuda ){{cite journal}}: Requiere citar revista |journal=( ayuda ){{cite journal}}: Requiere citar revista |journal=( ayuda )