
Los sistemas de secreción bacteriana son complejos proteicos presentes en las membranas celulares de las bacterias para la secreción de sustancias. En concreto, son los dispositivos celulares utilizados por las bacterias patógenas para secretar sus factores de virulencia (principalmente proteínas) para invadir las células huésped. Se pueden clasificar en diferentes tipos en función de su estructura, composición y actividad específicas. En general, las proteínas se pueden secretar a través de dos procesos diferentes. Un proceso es un mecanismo de un solo paso en el que las proteínas del citoplasma de las bacterias se transportan y se entregan directamente a través de la membrana celular a la célula huésped. Otro implica una actividad de dos pasos en la que las proteínas se transportan primero fuera de la membrana celular interna, luego se depositan en el periplasma y, finalmente, a través de la membrana celular externa a la célula huésped. [2]
Estas diferencias importantes se pueden distinguir entre las bacterias didérmicas gramnegativas y las bacterias monodérmicas grampositivas . Pero la clasificación no es en absoluto clara ni completa. Hay al menos ocho tipos específicos de bacterias gramnegativas, cuatro de bacterias grampositivas, mientras que dos son comunes a ambas. [3] Además, existe una diferencia apreciable entre las bacterias didérmicas con lipopolisacárido en la membrana externa (didermo-LPS) y las que tienen ácido micólico (didermo-micolato). [4]
La vía de exportación es responsable de atravesar la membrana celular interna en los didermos y la única membrana celular en los monodermos. [4]
La secreción general (Sec) implica la secreción de proteínas desplegadas que primero permanecen dentro de las células. En las bacterias Gram-negativas, la proteína secretada se envía a la membrana interna o al periplasma. Pero en las bacterias Gram-positivas, la proteína puede permanecer en la célula o es transportada principalmente fuera de la bacteria utilizando otros sistemas de secreción. Entre las bacterias Gram-negativas, Escherichia coli , Vibrio cholerae , Klebsiella pneumoniae y Yersinia enterocolitica utilizan el sistema Sec. Staphylococcus aureus y Listeria monocytogenes son bacterias Gram-positivas que utilizan el sistema Sec. [5]
El sistema Sec utiliza dos vías diferentes para la secreción: la vía SecA y la vía de la partícula de reconocimiento de señal (SRP). SecA es una proteína motora de ATPasa y tiene muchas proteínas relacionadas, incluidas SecD, SecE, SecF, SegG, SecM y SecY. SRP es una ribonucleoproteína (complejo proteína-ARN) que reconoce y dirige proteínas específicas al retículo endoplásmico en eucariotas y a la membrana celular en procariotas. Las dos vías requieren diferentes chaperonas moleculares y, en última instancia, utilizan un canal de transporte de proteínas SecYEG para transportar las proteínas a través de la membrana celular interna. [6] En la vía SecA, SecB actúa como chaperona, ayudando al transporte de proteínas al periplasma después de la síntesis completa de las cadenas de péptidos. Mientras que en la vía SRP, YidC es la chaperona y transporta proteínas a la membrana celular mientras aún están experimentando la síntesis de péptidos. [7] En Escherichia coli , las proteínas de membrana interna son el objetivo principal de la vía SRP y las proteínas de membrana externa o periplásmicas son el objetivo de la vía SecA. [8] Sin embargo, un estudio reciente de perfil selectivo de ribosomas sugiere que las proteínas de membrana interna con grandes bucles periplásmicos son el objetivo de la vía SecA. [9]
Las proteínas se sintetizan en los ribosomas mediante un proceso de adición seriada de aminoácidos, denominado traducción. En la vía SecA, un factor desencadenante (TF) chaperona se une primero a la secuencia señal N-terminal expuesta de la cadena peptídica. A medida que continúa la elongación de la cadena peptídica, el TF es reemplazado por SecB. SecB mantiene específicamente el péptido en un estado desplegado y ayuda a la unión de SecA. El complejo puede entonces unirse a SecYEG, por lo que SecA se activa al unirse con ATP. Impulsada por la energía del ATP, SecA empuja la proteína a través del canal secYEG. El complejo SecD/F también ayuda a extraer la proteína del otro lado de la membrana celular. [10]
En los últimos años, también se ha sugerido que la vía SecA tiene un mecanismo cotraduccional, lo que significa que el polipéptido sería el objetivo directo de SecA durante su síntesis. [11]
En esta vía, el SRP compite con el TF y se une a la secuencia señal N-terminal. Las proteínas de la membrana interna detienen el proceso de elongación de la cadena. El SRP se une entonces a un receptor de membrana, FtsY. El complejo cadena peptídica-SRP-FtsY se transporta entonces a SecY, donde se reanuda la elongación del péptido. [7]
La vía de translocación de arginina gemela (vía Tat) es similar a Sec en el proceso de secreción de proteínas, sin embargo, envía proteínas solo en su estado plegado (terciario). Es utilizada por todos los tipos de bacterias, así como por arqueas y cloroplastos y mitocondrias de plantas. [12] En las bacterias, el sistema Tat exporta proteínas desde el citoplasma a través de la membrana celular interna; mientras que en los cloroplastos, está presente en la membrana tilacoide donde ayuda a la importación de proteínas desde el estroma. [13] Las proteínas Tat son muy variables en diferentes bacterias y se clasifican en tres tipos principales, a saber, TatA, TatB y TatC. Por ejemplo, mientras que solo hay dos proteínas Tat funcionales en Bacillus subtilis , [14] puede haber más de cien en Streptomyces coelicolor . [15] Los péptidos señal que pueden reconocer las proteínas Tat se caracterizan por un motivo de consenso Ser/Thr-Arg-Arg-X-Phe-Leu-Lys (donde X puede ser cualquier aminoácido polar). Son las dos argininas sucesivas de las que proviene el nombre de translocación de arginina gemela. La sustitución de cualquiera de las argininas conduce a la disminución o la falla de la secreción. [16]
La vía Wss/Esx ( sistema ESAT-6 ) a veces se denomina sistema de secreción tipo VII (T7SS) a pesar de ser una vía de exportación. [4] Está presente en bacterias Gram-positivas (como WSS) y Mycobacteria (como Esx en todos los didermos-micolatos) como M. tuberculosis , M. bovis , Streptomyces coelicolor y S. aureus . También se denomina sistema T7b en Bacillus subtilis y S. aureus . Está compuesto por dos componentes básicos: una ATPasa hexamérica unida a la membrana que es miembro de la familia de proteínas FtsK /SpoIIIE, [17] y cualquiera de las proteínas relacionadas con EsxA/EsxB como EsaA, EsaD, EsxB, EsxD, así como el sistema Ess (EssA, EssB y EsxC encontrados en S. aureus ). [18] EsxA y EsxB pertenecen a una superfamilia de proteínas WXG100 que forman horquillas helicoidales diméricas.
En S. aureus , T7SS secreta una toxina grande llamada EsaD, que es un miembro de las enzimas nucleasas . EsaD se vuelve inofensiva (desintoxicada) durante su biosíntesis con la ayuda de su contraparte antitoxina EsaG. El complejo EsaD-EsaG luego se une con EsaE. La porción EsaE se une a EssC, que es una enzima ATPasa del complejo T7SS. Durante la secreción, EsaG queda en el citoplasma, y solo EsaD y EsaE se secretan juntas. Pero en algunas cepas de S. aureus , EsaD no se produce, sino que se forman dos copias de proteínas similares a EsaG. Esto podría explicar la aparición de T7SS en especies no patógenas como B. subtilis y S. coelicolor . [19]
Los sistemas de secreción son los responsables de atravesar la membrana celular externa o ambas membranas en los didermos. La nomenclatura actual se aplica únicamente a los didermos-LPS, ya que no se sabe nada sobre lo que utilizan las bacterias didermos-micolatos para atravesar su membrana externa. [4]

El sistema de secreción de tipo I (T1SS o TOSS) se encuentra en las bacterias Gram negativas. Depende de la actividad de las chaperonas que utilizan las proteínas Hly y Tol. El sistema se activa cuando una secuencia señal HlyA se une a HlyB en la membrana celular. Esta secuencia señal es un transportador ABC. El complejo HlyAB activa a HlyD, que se desenrolla y se desplaza hacia la membrana celular externa. La señal terminal es reconocida por TolC en la membrana interna. La HlyA se secreta fuera de la membrana externa a través de un canal proteico en forma de túnel.
El T1SS transporta diversas moléculas, entre ellas iones, carbohidratos, fármacos y proteínas. Las moléculas secretadas varían en tamaño, desde el pequeño péptido colicina V de Escherichia coli , que pesa 10 kDa, hasta la proteína de adhesión celular LapA de Pseudomonas fluorescens , que pesa 520 kDa. [20] Entre las moléculas más conocidas se encuentran las toxinas RTX y las enzimas lipasas.

El sistema de secreción de tipo II (T2SS) depende del sistema Sec o Tat para la secreción inicial dentro de la célula bacteriana. Desde el periplasma, las proteínas se secretan a partir de las secretinas de la membrana externa. Las secretinas son complejos multiméricos (12-14 subunidades) de proteínas formadoras de poros. La secretina está respaldada por otras 10-15 proteínas de membrana interna y externa para constituir el aparato de secreción completo. [21]

El sistema de secreción de tipo III (T3SS o TTSS) es estructuralmente similar y está relacionado con el cuerpo basal de los flagelos bacterianos . Se observa en algunas de las bacterias gramnegativas más virulentas, como Salmonella , Shigella , Yersinia y Vibrio , y se utiliza para inyectar proteínas tóxicas en células eucariotas. La estructura del T3SS se describe a menudo como un inyectoma o un aparato similar a una aguja o jeringa. Descubierto en Yersinia pestis , se encontró que el T3SS puede inyectar toxinas directamente desde el citoplasma bacteriano al citoplasma de las células de su huésped. [22]
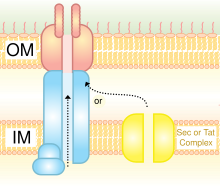
El sistema de secreción de tipo IV (T4SS o TFSS) está relacionado con el sistema de conjugación bacteriana , por el cual diferentes bacterias pueden intercambiar sus ADN. Las bacterias participantes pueden ser de la misma especie bacteriana Gram-negativa o de diferentes especies. Puede transportar proteínas individuales, así como complejos proteína-proteína y ADN-proteína. La secreción se transfiere directamente desde la célula receptora a través de las membranas celulares. Agrobacterium tumefaciens , a partir del cual se descubrió originalmente, utiliza este sistema para enviar la porción de T-ADN del plásmido Ti a las células vegetales, en las que se produce una agalla de corona (tumor) como resultado. Helicobacter pylori lo utiliza para entregar CagA a las células epiteliales gástricas, para inducir cáncer gástrico. [23] Bordetella pertussis , la bacteria causante de la tos ferina, secreta su toxina pertussis en parte a través de T4SS. Legionella pneumophila que causa legionelosis (enfermedad del legionario) tiene un T4SS llamado icm/dot ( genes de multiplicación intracelular / defecto en el tráfico de orgánulos ) que transporta muchas proteínas bacterianas a su huésped eucariota. [24] Más recientemente, se ha demostrado que el fitopatógeno Xanthomonas citri utiliza su T4SS para secretar efectores que son letales para otras especies bacterianas, lo que coloca a este sistema como un determinante principal de la aptitud de la competencia bacteriana entre especies. [25] [26] El sistema de secreción tipo IVA prototípico es el complejo VirB de Agrobacterium tumefaciens . [27]
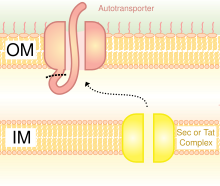
Los sistemas de secreción de tipo V (T5SS) se diferencian de otros sistemas de secreción en que se secretan a sí mismos y solo involucran la membrana celular externa. Para que la proteína secretada pase a través de la membrana celular interna, los T5SS dependen del sistema Sec. Tienen un dominio de barril β, que se inserta en la membrana celular externa y forma un canal que puede transportar la proteína secretada junto con ella. Por esta actividad, también se denominan sistemas autotransportadores. [28] Cuando las proteínas secretadas se exponen al exterior, los autotransportadores se cortan (se escinden), liberando la proteína del dominio de barril β. Un ejemplo de autotransportador son las adhesinas autotransportadoras triméricas . [29]
Los sistemas de secreción de tipo VI (T6SS) fueron descubiertos por el equipo de John Mekalanos en la Escuela de Medicina de Harvard en 2006 a partir de Vibrio cholerae y Pseudomonas aeruginosa . [30] [31] Fueron reconocidos cuando las mutaciones en los genes Hcp y VrgG de Vibrio Cholerae causaron una disminución de la virulencia y la patogenicidad. [32] [33] Además de su papel clásico como factor de patogenicidad, los T6SS también están involucrados en la defensa contra depredadores eucariotas simples y en interacciones entre bacterias. [34] [35] El gen para T6SS forma un grupo de genes que consta de más de 15 genes. Los genes Hcp y VgrG son los genes más universales. La similitud estructural de T6SS con la espiga de la cola del fago T4 sugiere que el proceso de infección es similar al del fago. [36]

El T7SS de las bacterias diderm-LPS es la vía chaperona-usher . [4]
En las bacterias didermo-micoladas este sistema de secreción es el ESAT-6 . [4]
El T8SS de las bacterias diderm-LPS es la vía de nucleación-precipitación extracelular. [4] [37]

Los sistemas de secreción de tipo IX (T9SS) se encuentran regularmente en el linaje de bacterias Fibrobacteres-Chlorobi-Bacteroidetes , donde las especies miembro incluyen una membrana externa. El sistema está involucrado de forma variable en un tipo de motilidad de deslizamiento, en la orientación adecuada de ciertos factores de virulencia a la superficie celular y en la degradación de complejos de biopolímeros. [39] El T9SS también se ha conocido como secreción de Por (acumulación de porfirina en la superficie celular), [4] en honor al patógeno oral Porphyromonas gingivalis . Se han descrito al menos dieciséis componentes estructurales del sistema, incluida PorU, una transpeptidasa de clasificación de proteínas que elimina la señal de clasificación C-terminal de las proteínas de carga y media su unión en su lugar al lipopolisacárido .