
La adenililación , [1] [2] más comúnmente conocida como AMPilación , es un proceso en el que una molécula de monofosfato de adenosina (AMP) se une covalentemente a la cadena lateral de aminoácidos de una proteína . [3] Esta adición covalente de AMP a una cadena lateral de hidroxilo de la proteína es una modificación postraduccional . [4] La adenililación implica un enlace fosfodiéster entre un grupo hidroxilo de la molécula que experimenta adenililación y el grupo fosfato del nucleótido monofosfato de adenosina (es decir, ácido adenílico). Las enzimas que son capaces de catalizar este proceso se denominan AMPiladores.
Los aminoácidos conocidos que se dirigen a la proteína son tirosina y treonina , y a veces serina . [5] Cuando las cargas de una proteína sufren un cambio, afecta las características de la proteína, normalmente alterando su forma a través de interacciones de los aminoácidos que componen la proteína. La AMPilación puede tener varios efectos en la proteína. Estas son propiedades de la proteína como la estabilidad, la actividad enzimática, la unión de cofactores y muchas otras capacidades funcionales de una proteína. Otra función de la adenililación es la activación de aminoácidos, que es catalizada por la ARNt aminoacil sintetasa. [3] Las proteínas más comúnmente identificadas para recibir AMPilación son las GTPasas y la glutamina sintetasa .
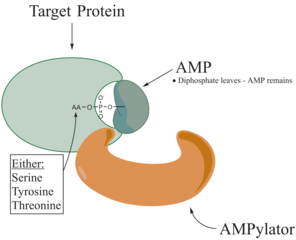
Las enzimas responsables de la AMPilación, llamadas AMPylators o Adenylyltransferase , se dividen en dos familias diferentes, todas dependiendo de sus propiedades estructurales y el mecanismo utilizado. AMPylator es creado por dos mitades catalíticas homólogas. Una mitad es responsable de catalizar la reacción de adenililación, mientras que la otra mitad cataliza la reacción de deadenilación fosforolítica [2] . Estas dos familias son la familia de las polimerasas tipo ADN- β y la familia Fic. [6]
La familia de las nucleótidos similares a la β -polimerasa de ADN es una familia de nucleótidos transferasas . [4] Se la conoce más específicamente como la familia GlnE. Existe un motivo específico que se utiliza para aclarar esta familia en particular. El motivo consiste en una lámina β de tres cadenas que forma parte de la coordinación de iones de magnesio y la unión de fosfato. El aspartato es esencial para que se produzca la actividad en esta familia.
El dominio Fic pertenece a la superfamilia Fido (Fic/Doc) La familia Fic , que es una filamentación inducida por el dominio AMP cíclico, es conocida por realizar AMPilación. Este término fue acuñado cuando se descubrió que VopS de Vibrio parahaemolyticus modificaba las RhoGTPasas con AMP en una serina. Esta familia de proteínas se encuentra en todos los dominios de la vida en la Tierra. Está mediada por un mecanismo de motivo de hélice alfa del sitio de unión de ATP. Las bacterias infecciosas utilizan este dominio para interrumpir la fagocitosis y causar la muerte celular. Los dominios Fic son dominios conservados evolutivamente en procariotas y eucariotas que pertenecen a la superfamilia de dominios Fido. [4]
Se ha demostrado que los AMPiladores son comparables a las quinasas debido a su actividad de hidrólisis de ATP y la transferencia reversible del metabolito a una cadena lateral de hidroxilo del sustrato proteico. Sin embargo, la AMPilación cataliza un ataque nucleofílico al grupo α-fosfato, mientras que la quinasa en la reacción de fosforilación se dirige al γ-fosfato. El ataque nucleofílico de la AMPilación conduce a la liberación de pirofosfato y la proteína modificada con AMP son los productos de la reacción de AMPilación. [5]

La desAMPilación es la reacción inversa en la que la molécula de AMP se separa del lado del aminoácido de una proteína de cadena.
Existen tres mecanismos conocidos para esta reacción. La GS-ATasa bacteriana (GlnE) codifica una proteína bipartita con dominios separados de AMPilación N-terminal y desAMPilación C-terminal cuya actividad está regulada por P II y modificaciones postraduccionales asociadas. La desAMPilación de su sustrato, la glutamina sintetasa AMPilada, se produce mediante una reacción fosforolítica entre la adenil-tirosina de GS y el ortofosfato , lo que conduce a la formación de ADP y glutamina sintetasa no modificada. [4]
La proteína SidD, introducida en la célula huésped por la bacteria patógena Legionella pneumophila , des-AMPila a Rab1, una proteína huésped AMPilada por una enzima distinta de Legionella pneumophila , la AMPilasa SidM. Aunque el beneficio para el patógeno de introducir estos dos efectores antagónicos en el huésped sigue sin estar claro, la reacción bioquímica llevada a cabo por SidD implica el uso de un dominio similar a la fosfatasa para catalizar la eliminación hidrolítica del AMP de la tirosina 77 de la proteína Rab1 del huésped. [7]
En las células animales, la eliminación de AMP de la treonina 518 de BiP/Grp78 es catalizada por la misma enzima, FICD, que AMPila BiP. A diferencia de la GS-ATasa bacteriana, FICD lleva a cabo ambas reacciones con el mismo dominio catalítico. [8]
La AMPilación está implicada en la homeostasis bacteriana. El ejemplo más famoso es la AMPiladora GS-ATasa (GlnE), que contribuye a la compleja regulación del metabolismo del nitrógeno a través de la AMPilación de la glutamina sintetasa que se introdujo en las partes de AMPilación y DeAMPilación.
Otro ejemplo de AMPiladores que desempeñan un papel en la homeostasis bacteriana son los AMPiladores Fic de clase I (FicT), que modifican la subunidad GyrB de la ADN girasa, el residuo de tirosina conservado para la unión de ATP de la subunidad ParE en la topoisomerasa IV. Esta inactivación de la ADN girasa por AMPilación conduce a la activación de la respuesta SOS, que es la respuesta celular al daño del ADN. La actividad de AMPilación de FicT es reversible y solo conduce a la detención del crecimiento, pero no a la muerte celular. Por lo tanto, la AMPilación de FicT desempeña un papel en la regulación del estrés celular, lo que se demuestra en la bacteria Wolbachia, donde el nivel de FicT aumenta en respuesta a la doxiciclina.
También se ha descubierto que un AMPilador de Fic de clase III, NmFic de N. meningtidis, modifica el AMPilato GyrB en la tirosina conservada para la unión de ATP. Esto demuestra que los dominios Fic están altamente conservados, lo que indica el importante papel de la AMPilación en la regulación del estrés celular en las bacterias. La regulación de NmFic implica la monomerización y la autoAMPilación dependientes de la concentración para la activación de la actividad de NmFic. [5]
Se ha demostrado que las proteínas bacterianas, también conocidas como efectores, utilizan la AMPilación. Se ha demostrado que los efectores como VopS, IbpA y DrrA, AMPilan las GTPasas del huésped y provocan cambios en el citoesqueleto de actina. Las GTPasas son objetivos comunes de los AMPiladores. Las familias de GTPasas Rho , Rab y Arf están involucradas en la dinámica del citoesqueleto de actina y el tráfico vesicular. También desempeñan funciones en los mecanismos de control celular como la fagocitosis en la célula huésped.
El patógeno mejora o previene su internalización induciendo o inhibiendo la fagocitosis de la célula huésped [4] . Vibrio parahaemolyticus es una bacteria Gram-negativa que causa intoxicación alimentaria como resultado del consumo de mariscos crudos o poco cocidos en humanos. [9] VopS, un efector tipo III encontrado en Vibrio parahaemolyticus , contiene un dominio Fic que tiene un motivo HPFx(D/E)GN(G/K)R conservado que contiene un residuo de histidina esencial para la AMPilación. VopS bloquea el ensamblaje de actina modificando el residuo de treonina en la región switch 1 de las Rho GTPasas. La transferencia de una fracción de AMP usando ATP al residuo de treonina resulta en un impedimento estérico y, por lo tanto, evita que las Rho GTPasas interactúen con los efectores posteriores. VopS también adenila RhoA y el ciclo de división celular 42 (CDC42), lo que lleva a una desagregación de la red de filamentos de actina. [3] [5] Como resultado, el control del citoesqueleto de actina de la célula huésped se desactiva, lo que conduce al redondeo celular. [4] [9]
La IbpA se secreta en células eucariotas de H. somni , una bacteria gramnegativa del ganado que causa infecciones del epitelio respiratorio. Este efector contiene dos dominios Fic en la región C-terminal. La AMPilación del dominio Fic de IbpA de las GTPasas de la familia Rho es responsable de su citotoxicidad. Ambos dominios Fic tienen efectos similares en el citoesqueleto de las células huésped como VopS. [3] [5] La AMPilación en un residuo de tirosina de la región switch 1 bloquea la interacción de las GTPasas con sustratos posteriores como PAK.
DrrA es el sustrato del sistema de translocación Dot/Icm tipo IV DrrA de Legionella pneumophila . Es el efector secretado por L. pneumophila para modificar las GTPasas de las células huésped. Esta modificación aumenta la supervivencia de las bacterias en las células huésped. DrrA está compuesto por el dominio del factor de intercambio de nucleótidos de guanina (GEF) específico de Rab1b , un dominio de unión a lípidos C-terminal y un dominio N-terminal con propiedades citotóxicas poco claras. Los trabajos de investigación muestran que DrrA N-terminal y de longitud completa muestra actividad AMPylators hacia la proteína Rab1b del huésped (proteína relacionada con Ras), que también es el sustrato del dominio GEF de Rab1b. La proteína Rab1b es la GTPasa Rab para regular el transporte de vesículas y la fusión de membranas. La adenilación por AMPylators de bacterias prolonga el estado unido a GTP de Rab1b. Por lo tanto, el papel del efector DrrA está conectado con los beneficios de las vacuolas de las bacterias para su replicación durante la infección. [3] [5]
Las plantas y las levaduras no tienen enzimas AMPilantes endógenas conocidas, pero los genomas animales están dotados de una única copia de un gen que codifica una AMPilasa de dominio Fic, [10] que probablemente fue adquirida por un ancestro temprano de los animales a través de la transferencia horizontal de genes desde un procariota. La proteína humana conocida comúnmente como FICD, había sido identificada previamente como proteína E asociada a Huntingtin (HypE; una asignación que surge de una pantalla de dos híbridos de levadura, pero de relevancia cuestionable, ya que Huntingtin y HypE/FICD están localizados en diferentes compartimentos celulares). Los homólogos de CG9523 en Drosophila melanogaster (CG9523) y C. elegans (Fic-1) también han recibido atención. En todos los animales, FICD tiene una estructura similar. Es una proteína de dominio transmembrana tipo II , con un dominio citoplasmático corto seguido de un ancla de membrana que sostiene la proteína en el retículo endoplásmico (RE) y una porción C-terminal larga que reside en el RE y abarca repeticiones de tetratricopéptidos (TPR) seguidas de un dominio catalítico Fic. [11]
El descubrimiento de una AMPilasa en células animales [10], seguido por el descubrimiento de su localización en el RE y de que la BiP es un sustrato importante para su actividad [12], fueron avances importantes. Se sabía desde hacía tiempo que la BiP (también conocida como Grp78) sufría una modificación postraduccional inactivadora [13] [14] , pero su naturaleza sigue siendo esquiva. Aunque se creía ampliamente que se trataba de ADP-ribosilación , resultó ser una AMPilación mediada por FICD, ya que la inactivación del gen FICD en las células abolió toda modificación postraduccional mensurable de la BiP [15] .
BiP es una proteína chaperona localizada en el RE cuya actividad está estrechamente regulada a nivel transcripcional a través de un programa de expresión génica conocido como la Respuesta de Proteínas Desplegadas (UPR). La UPR es un proceso homeostático que acopla la tasa de transcripción de BiP (y muchas otras proteínas) a la carga de proteínas desplegadas en el RE (el llamado estrés del RE) para ayudar a mantener la proteostasis del RE . La AMPilación agrega otra capa rápida de control postraduccional de la actividad de BiP, ya que la modificación de Thr518 del dominio de unión al sustrato de BiP con un AMP bloquea la chaperona en una conformación inactiva. [16] [17] Esta modificación se implementa selectivamente a medida que el estrés del RE disminuye, para inactivar el exceso de BiP. Sin embargo, a medida que el estrés del RE aumenta nuevamente, la misma enzima, FICD, cataliza la reacción opuesta, la des-AMPilación de BiP. [8]
Está surgiendo gradualmente una comprensión de la base estructural de la AMPilación y desAMPilación de BiP, [18] [19] al igual que pistas sobre el alosterio que podría regular el cambio en la actividad de FICD [20] pero aún quedan por descubrir detalles importantes de este proceso tal como ocurre en las células.
El papel del FICD en la AMPilación (y desAMPilación) de BiP en Thr518 está bien respaldado por estudios bioquímicos y estructurales. También se ha presentado evidencia de que en algunas circunstancias el FICD puede AMPilar un residuo diferente, Thr366 en el dominio de unión de nucleótidos de BiP. [12]
Fic-1 es la única proteína Fic presente en el código genético de C. elegans . Se encuentra principalmente en la envoltura nuclear del RE de las células germinales adultas y las células embrionarias, pero se pueden encontrar pequeñas cantidades dentro del citoplasma. A este grupo de FICD-1 extra-RE se le atribuye la AMPilación de las histonas centrales y los factores de traducción de tipo eEF1-A dentro del nematodo. [21]
Aunque los diferentes niveles de AMPilación no crearon ningún efecto notable en el comportamiento o la fisiología del nematodo, los gusanos knock out de Fic-1 fueron más susceptibles a la infección por Pseudomonas aeruginosa en comparación con sus contrapartes con dominios Fic-1 activos, lo que implica un vínculo entre la AMPilación de objetivos celulares y las respuestas inmunes dentro de los nematodos. [11]
Las moscas que carecen de FICD (CG9523) han sido descritas como ciegas. Inicialmente, este defecto se atribuyó a una función del FICD en la superficie celular de las proyecciones capitadas, un supuesto sitio de reciclaje de neurotransmisores [22]; sin embargo, un estudio posterior implicó la AMPilación de BiP Thr366 mediada por FICD en el problema visual [23].
Se descubrió que la proteína presináptica α-sinucleína es un objetivo para la AMPilación de FICD. Durante la adenililación de αSyn mediada por HypE , la agregación de αSyn disminuye y se descubrió que tanto la neurotoxicidad como el estrés del RE disminuyen in vitro . Por lo tanto, la adenililación de αSyn es posiblemente una respuesta protectora al estrés del RE y la agregación de αSyn. Sin embargo, como aSyn y FICD residen en compartimentos diferentes, se necesita realizar más investigaciones para confirmar la importancia de estas afirmaciones. [24]
Los marcadores químicos se utilizan para detectar proteínas modificadas postraduccionalmente. Recientemente, existe un N6pATP que contiene una etiqueta de alquinilo (propargilo) en la posición N6 de la adenina del ATP. Este N6pATP se combina con la reacción de clic para detectar proteínas AMPiladas. Para detectar proteínas modificadas no reconocidas y etiquetar sustratos VopS, se utilizan derivados de ATP con un fluoróforo en la posición N6 de la adenina NH2. [5] [6]
Los anticuerpos son famosos por su alta afinidad y selectividad, por lo que son una buena forma de detectar proteínas AMPiladas. Recientemente, los anticuerpos ɑ-AMP se utilizan para detectar y aislar directamente las proteínas AMPiladas (especialmente tirosina AMPilada y treonina AMPilada) de las células y los lisados celulares. La AMPilación es una modificación postraduccional, por lo que modificará las propiedades de las proteínas al otorgarle el carácter polar de AMP e hidrofobicidad. Por lo tanto, en lugar de utilizar anticuerpos que detecten una secuencia peptídica completa, se prefiere generar anticuerpos AMP directamente dirigidos a aminoácidos específicos. [5] [6]
Anteriormente, muchos trabajos científicos utilizaban la espectrometría de masas (MS) en diferentes modos de fragmentación para detectar péptidos AMPilados. En respuesta a las técnicas de fragmentación distintivas, las secuencias de proteínas AMPiladas se desintegraban en diferentes partes del AMP. Mientras que la disociación por transferencia de electrones (ETD) crea fragmentos mínimos y espectros menos complicados, la disociación inducida por colisión (CID) y la fragmentación por colisión de alta energía (HCD) generan iones característicos adecuados para la identificación de proteínas AMPiladas mediante la generación de múltiples fragmentos de AMP. Debido a la estabilidad del AMP, los espectros de fragmentación de péptidos son fáciles de leer manualmente o con motores de búsqueda. [5] [6]
Se han descubierto inhibidores de la AMPilación de proteínas con una constante inhibitoria (K i ) que varía entre 6 y 50 μM y una selectividad al menos 30 veces mayor que la de HypE. [25] [5] [6]