Viroma
La palabra se usa con frecuencia para describir los metagenomas virales de escopeta ambientales.
[18] A continuación, los ácidos nucleicos se secuencian y analizan utilizando métodos metagenómicos .
Alternativamente, existen métodos computacionales recientes que utilizan secuencias ensambladas directamente metagenómicas para descubrir virus.
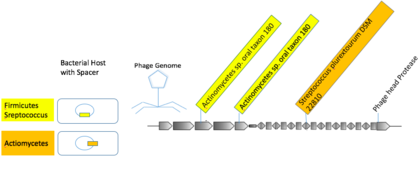
[21] Se utilizó un conjunto de métodos computacionales para identificar las conexiones putativas del virus huésped.
Algunos mVC se vincularon a múltiples huéspedes de taxones superiores.
Un grupo viral compuesto por macs de muestras orales humanas contenía tres foto-espaciadores distintos con coincidencias casi exactas con los espaciadores en Actionbacteria y Firmicutes.