Vacunología inversa
Esto emplea técnica bioinformática, iniciada por Rino Rappuoli y utilizada por primera vez contra el meningococo del serogrupo B.
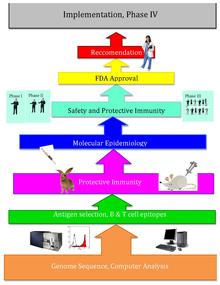
[3] La idea básica detrás de la vacunación inversa es que se puede cribar un genoma patógeno completo utilizando enfoques bioinformáticos para encontrar genes.
Como resultado, Rino Rappuoli y otros científicos recurrieron a la bioinformática para diseñar una vacuna funcional.
[6] Rappuoli y otros del Instituto J. Craig Venter secuenciaron por primera vez el genoma de MenB.
Debido a que esas cepas bacterianas inducen anticuerpos que reaccionan con antígenos humanos, las vacunas para esas bacterias debían no contener homologaciones con proteínas codificadas en el genoma humano para no causar reacciones adversas, estableciendo así la necesidad de una vacunación inversa basada en dicho genoma.
Los métodos tradicionales pueden tardar décadas en desentrañar patógenos y antígenos, enfermedades e inmunidad.
Sin embargo, In silico puede ser muy rápido, lo que permite identificar nuevas vacunas para probar en solo unos pocos años.
Considerando que, los enfoques de vacunología convencionales pueden encontrar otras dianas biomoleculares como los polisacáridos .