
Un vector de clonación es un pequeño fragmento de ADN que se puede mantener de forma estable en un organismo y en el que se puede insertar un fragmento de ADN extraño con fines de clonación . [1] El vector de clonación puede ser ADN tomado de un virus , la célula de un organismo superior o puede ser el plásmido de una bacteria. El vector contiene características que permiten la inserción conveniente de un fragmento de ADN en el vector o su eliminación del vector, por ejemplo, a través de la presencia de sitios de restricción . El vector y el ADN extraño pueden tratarse con una enzima de restricción que corta el ADN, y los fragmentos de ADN así generados contienen extremos romos o salientes conocidos como extremos pegajosos, y el ADN del vector y el ADN extraño con extremos compatibles pueden unirse entonces mediante ligadura molecular . Después de que un fragmento de ADN se ha clonado en un vector de clonación, puede subclonarse aún más en otro vector diseñado para un uso más específico.
Existen muchos tipos de vectores de clonación, pero los más utilizados son los plásmidos modificados genéticamente . La clonación se realiza generalmente primero utilizando Escherichia coli , y los vectores de clonación en E. coli incluyen plásmidos, bacteriófagos (como el fago λ ), cósmidos y cromosomas artificiales bacterianos (BAC). Sin embargo, algunos ADN no se pueden mantener de forma estable en E. coli , por ejemplo fragmentos de ADN muy grandes, y se pueden utilizar otros organismos como la levadura. Los vectores de clonación en levadura incluyen cromosomas artificiales de levadura (YAC).
Todos los vectores de clonación comúnmente utilizados en biología molecular tienen características clave necesarias para su función, como un sitio de clonación adecuado y un marcador seleccionable. Otros pueden tener características adicionales específicas para su uso. Por razones de facilidad y conveniencia, la clonación a menudo se realiza utilizando E. coli . Por lo tanto, los vectores de clonación utilizados a menudo tienen elementos necesarios para su propagación y mantenimiento en E. coli , como un origen de replicación funcional (ori). El origen de replicación ColE1 se encuentra en muchos plásmidos. Algunos vectores también incluyen elementos que les permiten mantenerse en otro organismo además de E. coli , y estos vectores se denominan vector lanzadera .
Todos los vectores de clonación tienen características que permiten insertar o extraer un gen de manera conveniente. Puede tratarse de un sitio de clonación múltiple (MCS) o un polienlazador, que contiene muchos sitios de restricción únicos . Los sitios de restricción en el MCS se escinden primero mediante enzimas de restricción y luego se liga un gen diana amplificado por PCR también digerido con las mismas enzimas a los vectores utilizando la ADN ligasa . La secuencia de ADN diana se puede insertar en el vector en una dirección específica si así se desea. Los sitios de restricción se pueden utilizar además para la subclonación en otro vector si es necesario. [ cita requerida ]
Otros vectores de clonación pueden utilizar topoisomerasa en lugar de ligasa y la clonación puede realizarse más rápidamente sin la necesidad de digestión de restricción del vector o inserto. En este método de clonación TOPO, se activa un vector linealizado uniendo la topoisomerasa I a sus extremos, y este vector "activado por TOPO" puede entonces aceptar un producto de PCR ligando ambos extremos 5' del producto de PCR, liberando la topoisomerasa y formando un vector circular en el proceso. [2] Otro método de clonación sin el uso de digestión de ADN y ligasa es mediante recombinación de ADN , por ejemplo, como se utiliza en el sistema de clonación Gateway . [3] [4] El gen, una vez clonado en el vector de clonación (llamado clon de entrada en este método), puede introducirse convenientemente en una variedad de vectores de expresión mediante recombinación. [5]
El vector lleva un marcador seleccionable para permitir la selección de células transformadas positivamente. La resistencia a los antibióticos se utiliza a menudo como marcador, un ejemplo es el gen de la beta-lactamasa , que confiere resistencia al grupo de penicilina de antibióticos beta-lactámicos como la ampicilina . Algunos vectores contienen dos marcadores seleccionables, por ejemplo, el plásmido pACYC177 tiene genes de resistencia a la ampicilina y a la kanamicina . [6] El vector lanzadera que está diseñado para mantenerse en dos organismos diferentes también puede requerir dos marcadores seleccionables, aunque algunos marcadores seleccionables como la resistencia a la zeocina y la higromicina B son eficaces en diferentes tipos de células. También se pueden utilizar marcadores de selección auxotróficos que permiten que un organismo auxotrófico crezca en un medio de crecimiento mínimo ; ejemplos de estos son LEU2 y URA3 que se utilizan con sus cepas auxotróficas correspondientes de levadura. [7]
Otro tipo de marcador seleccionable permite la selección positiva de plásmidos con genes clonados. Esto puede implicar el uso de un gen letal para las células huésped, como la barnasa , [8] Ccda , [9] y las toxinas parD/parE . [10] [11] Esto normalmente funciona alterando o eliminando el gen letal durante el proceso de clonación, y los clones fallidos en los que el gen letal aún permanece intacto matarían a las células huésped, por lo tanto, solo se seleccionan los clones exitosos.
Los genes reporteros se utilizan en algunos vectores de clonación para facilitar la selección de clones exitosos mediante el uso de características de estos genes que permiten identificar fácilmente el clon exitoso. Dichas características presentes en los vectores de clonación pueden ser el fragmento α lacZ para la complementación α en la selección azul-blanca , y/o genes marcadores o genes reporteros en marco con y flanqueando el MCS para facilitar la producción de proteínas de fusión . Ejemplos de socios de fusión que pueden usarse para la selección son la proteína fluorescente verde (GFP) y la luciferasa .
Un vector de clonación no necesita contener elementos adecuados para la expresión de un gen diana clonado, como un promotor y un sitio de unión ribosomal (RBS), sin embargo muchos sí los contienen, y pueden funcionar entonces como un vector de expresión . El ADN diana puede insertarse en un sitio que esté bajo el control de un promotor particular necesario para la expresión del gen diana en el huésped elegido. Cuando el promotor está presente, la expresión del gen está preferiblemente controlada de forma estricta e inducible de modo que las proteínas solo se produzcan cuando sea necesario. Algunos promotores de uso común son los promotores T7 y lac . La presencia de un promotor es necesaria cuando se utilizan técnicas de cribado como la selección azul-blanca .
A veces se utilizan vectores de clonación sin promotor ni RBS para la secuencia de ADN clonada, por ejemplo, cuando se clonan genes cuyos productos son tóxicos para las células de E. coli . El promotor y el RBS para la secuencia de ADN clonada también son innecesarios cuando se crea por primera vez una biblioteca genómica o de ADNc de clones, ya que los genes clonados normalmente se subclonan en un vector de expresión más apropiado si se requiere su expresión.
Algunos vectores están diseñados únicamente para la transcripción sin expresión de proteínas heterólogas, por ejemplo, para la producción de ARNm in vitro . Estos vectores se denominan vectores de transcripción. Pueden carecer de las secuencias necesarias para la poliadenilación y la terminación, por lo que no pueden utilizarse para la producción de proteínas.
Existe una gran cantidad de vectores de clonación disponibles y la elección del vector puede depender de varios factores, como el tamaño del inserto, el número de copias y el método de clonación. Es posible que un inserto grande no se mantenga de manera estable en un vector de clonación general, especialmente en el caso de aquellos con un alto número de copias, por lo que la clonación de fragmentos grandes puede requerir un vector de clonación más especializado. [6]
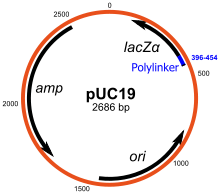
Los plásmidos son ADN extracromosómico circular que se replica de forma autónoma. Son los vectores de clonación estándar y los más utilizados. La mayoría de los plásmidos generales se pueden utilizar para clonar insertos de ADN de hasta 15 kb de tamaño. Uno de los primeros vectores de clonación utilizados comúnmente es el plásmido pBR322 . Otros vectores de clonación incluyen la serie de plásmidos pUC , y hay una gran cantidad de diferentes vectores de plásmidos de clonación disponibles. Muchos plásmidos tienen un alto número de copias, por ejemplo, pUC19 tiene un número de copias de 500 a 700 copias por célula, [6] y un alto número de copias es útil ya que produce un mayor rendimiento de plásmido recombinante para la manipulación posterior. Sin embargo, los plásmidos con un bajo número de copias se pueden utilizar preferiblemente en ciertas circunstancias, por ejemplo, cuando la proteína del gen clonado es tóxica para las células. [12]
Algunos plásmidos contienen un origen de replicación del bacteriófago M13 y pueden utilizarse para generar ADN monocatenario. Se denominan fagémidos y algunos ejemplos son la serie de vectores de clonación pBluescript.
Los bacteriófagos utilizados para la clonación son el fago λ y el fago M13 . [13] Hay un límite superior en la cantidad de ADN que se puede empaquetar en un fago (un máximo de 53 kb), por lo tanto, para permitir que se inserte ADN extraño en el ADN del fago, los vectores de clonación de fagos pueden necesitar que se eliminen algunos genes no esenciales, por ejemplo, los genes para la lisogenia , ya que el uso del fago λ como vector de clonación involucra solo el ciclo lítico. [14] Hay dos tipos de vectores de fago λ: vector de inserción y vector de reemplazo. Los vectores de inserción contienen un sitio de escisión único mediante el cual se puede insertar ADN extraño con un tamaño de 5-11 kb. En los vectores de reemplazo, los sitios de escisión flanquean una región que contiene genes no esenciales para el ciclo lítico, y esta región puede eliminarse y reemplazarse por el inserto de ADN en el proceso de clonación, y se puede insertar un ADN de mayor tamaño de 8-24 kb. [6]
También existe un límite de tamaño inferior para el ADN que se puede empaquetar en un fago, y el ADN del vector que es demasiado pequeño no se puede empaquetar adecuadamente en el fago. Esta propiedad se puede utilizar para la selección: el vector sin inserto puede ser demasiado pequeño, por lo tanto, solo los vectores con inserto se pueden seleccionar para la propagación. [15]
Los cósmidos son plásmidos que incorporan un segmento de ADN del bacteriófago λ que tiene el sitio final cohesivo ( cos ) que contiene elementos necesarios para empaquetar el ADN en partículas λ. Bajo el origen de replicación adecuado (ori), puede replicarse como un plásmido. Normalmente se utiliza para clonar fragmentos grandes de ADN entre 28 y 45 Kb. [6]
Se pueden clonar insertos de hasta 350 kb en cromosomas artificiales bacterianos (BAC). Los BAC se mantienen en E. coli con un número de copias de solo 1 por célula. [6] Los BAC se basan en el plásmido F , otro cromosoma artificial llamado PAC se basa en el fago P1 .
Los cromosomas artificiales de levadura se utilizan como vectores para clonar fragmentos de ADN de más de 1 megabase (1 Mb = 1000 kb) de tamaño. Son útiles para clonar fragmentos de ADN más grandes, como se requiere en el mapeo de genomas, como en el Proyecto Genoma Humano . Contienen una secuencia telomérica, una secuencia de replicación autónoma (características necesarias para replicar cromosomas lineales en células de levadura). Estos vectores también contienen sitios de restricción adecuados para clonar ADN extraño, así como genes que se pueden usar como marcadores seleccionables.
Los cromosomas artificiales humanos pueden ser potencialmente útiles como vectores de transferencia de genes para la administración de genes a células humanas y como herramienta para estudios de expresión y determinación de la función cromosómica humana. Pueden transportar fragmentos de ADN muy grandes (no hay un límite superior de tamaño para fines prácticos), por lo que no tienen el problema de la capacidad de clonación limitada de otros vectores, y también evitan la posible mutagénesis insercional causada por la integración en los cromosomas del huésped por el vector viral. [16] [17]
Los virus que infectan células animales y vegetales también han sido manipulados para introducir genes extraños en ellas. La capacidad natural de los virus de adsorberse en las células, introducir su ADN y replicarse los ha convertido en vehículos ideales para transferir ADN extraño a células eucariotas en cultivo. En el primer experimento de clonación con células de mamíferos se utilizó un vector basado en el virus de los simios 40 (SV40). Se han utilizado varios vectores basados en otros tipos de virus, como los adenovirus y el virus del papiloma, para clonar genes en mamíferos. En la actualidad, los vectores retrovirales son populares para clonar genes en células de mamíferos. En el caso de las plantas, se han utilizado con éxito limitado el virus del mosaico de la coliflor , el virus del mosaico del tabaco y los virus Gemini .

Muchos vectores de uso general, como pUC19, suelen incluir un sistema para detectar la presencia de un fragmento de ADN clonado, basándose en la pérdida de un fenotipo fácilmente puntuo- rado. El más utilizado es el gen que codifica la β-galactosidasa de E. coli , cuya actividad puede detectarse fácilmente por la capacidad de la enzima que codifica para hidrolizar el sustrato soluble e incoloro X-gal (5-bromo-4-cloro-3-indolil-beta-d-galactosido) en un producto insoluble y azul (5,5'-dibromo-4,4'-dicloro índigo). La clonación de un fragmento de ADN dentro de la secuencia lacZα basada en el vector de la β-galactosidasa impide la producción de una enzima activa. Si se incluye X-gal en las placas de agar selectivas, las colonias transformantes son generalmente azules en el caso de un vector sin ADN insertado y blancas en el caso de un vector que contiene un fragmento de ADN clonado.