En biología, un modelo de sustitución , también llamado modelo de evolución de secuencia , son modelos de Markov que describen cambios a lo largo del tiempo evolutivo. Estos modelos describen cambios evolutivos en macromoléculas, como secuencias de ADN o secuencias de proteínas , que pueden representarse como secuencias de símbolos (p. ej., A, C, G y T en el caso del ADN o los 20 aminoácidos proteinogénicos "estándar" en el caso de las proteínas ). Los modelos de sustitución se utilizan para calcular la probabilidad de que existan árboles filogenéticos utilizando datos de alineación de secuencias múltiples . Por lo tanto, los modelos de sustitución son fundamentales para la estimación de máxima verosimilitud de la filogenia, así como para la inferencia bayesiana en filogenia . Las estimaciones de distancias evolutivas (número de sustituciones que han ocurrido desde que un par de secuencias divergieron de un ancestro común) generalmente se calculan utilizando modelos de sustitución (las distancias evolutivas se utilizan como entrada para métodos de distancia como la unión de vecinos ). Los modelos de sustitución también son fundamentales para los invariantes filogenéticos porque son necesarios para predecir las frecuencias de patrones de sitios dada una topología de árbol. Los modelos de sustitución también son necesarios para simular datos de secuencia de un grupo de organismos relacionados por un árbol específico.
Las topologías de árboles filogenéticos suelen ser el parámetro de interés; [1] por lo tanto, las longitudes de las ramas y cualquier otro parámetro que describa el proceso de sustitución a menudo se consideran parámetros molestos . Sin embargo, los biólogos a veces se interesan por otros aspectos del modelo. Por ejemplo, la longitud de las ramas, especialmente cuando esas longitudes se combinan con información del registro fósil y un modelo para estimar el marco temporal de la evolución. [2] Se han utilizado otros parámetros del modelo para obtener información sobre diversos aspectos del proceso de evolución. La relación K a /K s (también llamada ω en los modelos de sustitución de codones) es un parámetro de interés en muchos estudios. La relación K a /K s se puede utilizar para examinar la acción de la selección natural en las regiones codificantes de proteínas, [3] proporciona información sobre las tasas relativas de sustituciones de nucleótidos que cambian los aminoácidos (sustituciones no sinónimos) a aquellos que sí lo hacen. no cambiar el aminoácido codificado (sustituciones sinónimas).
La mayor parte del trabajo sobre modelos de sustitución se ha centrado en la evolución de secuencias de ADN/ ARN y proteínas . Los modelos de evolución de la secuencia de ADN, donde el alfabeto corresponde a los cuatro nucleótidos (A, C, G y T), son probablemente los modelos más fáciles de entender. Los modelos de ADN también se pueden utilizar para examinar la evolución de los virus de ARN ; esto refleja el hecho de que el ARN también tiene un alfabeto de cuatro nucleótidos (A, C, G y U). Sin embargo, los modelos de sustitución se pueden utilizar para alfabetos de cualquier tamaño; el alfabeto son los 20 aminoácidos proteinogénicos para las proteínas y los codones sentidos (es decir, los 61 codones que codifican los aminoácidos en el código genético estándar ) para las secuencias de genes codificadores de proteínas alineadas. De hecho, se pueden desarrollar modelos de sustitución para cualquier carácter biológico que pueda codificarse utilizando un alfabeto específico (por ejemplo, secuencias de aminoácidos combinadas con información sobre la conformación de esos aminoácidos en estructuras proteicas tridimensionales [4] ).
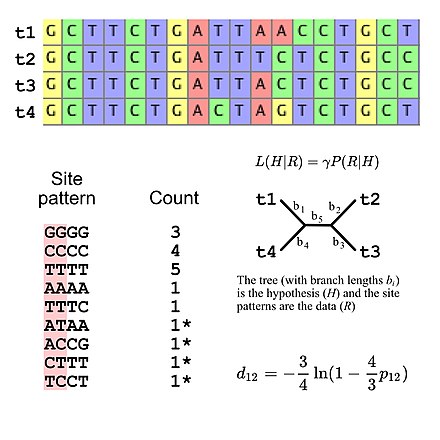
La mayoría de los modelos de sustitución utilizados para la investigación evolutiva asumen independencia entre sitios (es decir, la probabilidad de observar cualquier patrón de sitio específico es idéntica independientemente de dónde se encuentre el patrón de sitio en la alineación de secuencia). Esto simplifica los cálculos de probabilidad porque sólo es necesario calcular la probabilidad de todos los patrones de sitios que aparecen en la alineación y luego usar esos valores para calcular la probabilidad general de la alineación (por ejemplo, la probabilidad de tres patrones de sitios "GGGG" dado algún modelo de La evolución de la secuencia de ADN es simplemente la probabilidad de un único patrón de sitio "GGGG" elevado a la tercera potencia). Esto significa que se puede considerar que los modelos de sustitución implican una distribución multinomial específica para las frecuencias de patrones de sitio. Si consideramos un alineamiento de secuencia múltiple de cuatro secuencias de ADN, hay 256 patrones de sitios posibles, por lo que hay 255 grados de libertad para las frecuencias de los patrones de sitios. Sin embargo, es posible especificar las frecuencias esperadas del patrón de sitio usando cinco grados de libertad si se utiliza el modelo de evolución del ADN de Jukes-Cantor, [5] que es un modelo de sustitución simple que permite calcular las frecuencias esperadas del patrón de sitio solo en el árbol. topología y longitud de las ramas (dados cuatro taxones, un árbol bifurcado sin raíces tiene cinco longitudes de ramas).
Los modelos de sustitución también permiten simular datos de secuencia utilizando métodos de Monte Carlo . Se pueden utilizar alineamientos de secuencias múltiples simulados para evaluar el rendimiento de los métodos filogenéticos [6] y generar la distribución nula para ciertas pruebas estadísticas en los campos de la evolución molecular y la filogenética molecular. Ejemplos de estas pruebas incluyen pruebas de ajuste del modelo [7] y la "prueba SOWH" que se puede utilizar para examinar topologías de árboles. [8] [9]
El hecho de que los modelos de sustitución puedan usarse para analizar cualquier alfabeto biológico ha hecho posible desarrollar modelos de evolución para conjuntos de datos fenotípicos [10] (por ejemplo, rasgos morfológicos y de comportamiento). Normalmente, "0" es. se usa para indicar la ausencia de un rasgo y "1" se usa para indicar la presencia de un rasgo, aunque también es posible calificar personajes usando múltiples estados. Usando este marco, podríamos codificar un conjunto de fenotipos como cadenas binarias (esto podría generalizarse a cadenas de k estados para caracteres con más de dos estados) antes de realizar análisis usando un modo apropiado. Esto se puede ilustrar usando un ejemplo de "juguete": podemos usar un alfabeto binario para calificar los siguientes rasgos fenotípicos "tiene plumas", "pone huevos", "tiene pelaje", "tiene sangre caliente" y "es capaz de generar energía". vuelo". En este ejemplo de juguete, los colibríes tendrían la secuencia 11011 (la mayoría de las demás aves tendrían la misma cuerda), los avestruces tendrían la secuencia 11010, el ganado (y la mayoría de los demás mamíferos terrestres ) tendrían 00110 y los murciélagos tendrían 00111. La probabilidad de una secuencia filogenética Luego, el árbol se puede calcular utilizando esas secuencias binarias y un modelo de sustitución apropiado. La existencia de estos modelos morfológicos hace posible analizar matrices de datos con taxones fósiles, ya sea utilizando datos morfológicos solos [11] o una combinación de datos morfológicos y moleculares [12] (este último puntuado como datos faltantes para los taxones fósiles) .
Existe una similitud obvia entre el uso de datos moleculares o fenotípicos en el campo de la cladística y el análisis de caracteres morfológicos utilizando un modelo de sustitución. Sin embargo, ha habido un debate ruidoso [a] en la comunidad sistemática sobre la cuestión de si los análisis cladísticos deben considerarse "libres de modelos". El campo de la cladística (definido en el sentido más estricto) favorece el uso del criterio de máxima parsimonia para la inferencia filogenética. [13] Muchos cladistas rechazan la posición de que la máxima parsimonia se base en un modelo de sustitución y (en muchos casos) justifican el uso de la parsimonia utilizando la filosofía de Karl Popper . [14] Sin embargo, la existencia de modelos "equivalentes a la parsimonia" [15] (es decir, modelos de sustitución que producen el árbol de parsimonia máxima cuando se utilizan para análisis) hace posible ver la parsimonia como un modelo de sustitución. [1]
Normalmente, la longitud de una rama de un árbol filogenético se expresa como el número esperado de sustituciones por sitio; Si el modelo evolutivo indica que cada sitio dentro de una secuencia ancestral normalmente experimentará x sustituciones en el momento en que evolucione a una secuencia de descendiente particular, entonces se considera que el ancestro y el descendiente están separados por la longitud de la rama x .
A veces, la longitud de una rama se mide en términos de años geológicos. Por ejemplo, un registro fósil puede permitir determinar el número de años entre una especie ancestral y una especie descendiente. Debido a que algunas especies evolucionan a un ritmo más rápido que otras, estas dos medidas de longitud de las ramas no siempre están en proporción directa. El número esperado de sustituciones por sitio por año a menudo se indica con la letra griega mu (μ).
Se dice que un modelo tiene un reloj molecular estricto si el número esperado de sustituciones por año μ es constante independientemente de la evolución de la especie que se esté examinando. Una implicación importante de un reloj molecular estricto es que el número de sustituciones esperadas entre una especie ancestral y cualquiera de sus descendientes actuales debe ser independiente de qué especie descendiente se examine.
Tenga en cuenta que la suposición de un reloj molecular estricto suele ser poco realista, especialmente durante largos períodos de evolución. Por ejemplo, aunque los roedores son genéticamente muy similares a los primates , han sufrido un número mucho mayor de sustituciones en el tiempo estimado desde la divergencia en algunas regiones del genoma . [16] Esto podría deberse a su menor tiempo de generación , [17] mayor tasa metabólica , mayor estructuración de la población, mayor tasa de especiación o menor tamaño corporal. [18] [19] Cuando se estudian eventos antiguos como la explosión del Cámbrico bajo el supuesto de un reloj molecular, a menudo se observa poca concurrencia entre los datos cladísticos y filogenéticos. Se han realizado algunos trabajos sobre modelos que permiten una tasa de evolución variable. [20] [21]
Los modelos que pueden tener en cuenta la variabilidad del ritmo del reloj molecular entre diferentes linajes evolutivos en la filogenia se denominan "relajados" en oposición a "estrictos". En tales modelos se puede suponer que la tasa está correlacionada o no entre ancestros y descendientes y la variación de la tasa entre linajes se puede extraer de muchas distribuciones, pero generalmente se aplican distribuciones exponenciales y lognormales. Existe un caso especial, llamado “reloj molecular local”, cuando una filogenia se divide en al menos dos particiones (conjuntos de linajes) y en cada una se aplica un reloj molecular estricto, pero con ritmos diferentes.
Muchos modelos de sustitución útiles son reversibles en el tiempo ; En términos matemáticos, al modelo no le importa qué secuencia es el ancestro y cuál es el descendiente, siempre y cuando todos los demás parámetros (como el número de sustituciones por sitio que se espera entre las dos secuencias) se mantengan constantes.
Cuando se realiza un análisis de datos biológicos reales, generalmente no se tiene acceso a las secuencias de especies ancestrales, sólo a las especies actuales. Sin embargo, cuando un modelo es reversible en el tiempo, es irrelevante qué especie era la especie ancestral. En cambio, el árbol filogenético puede enraizarse utilizando cualquiera de las especies, volver a enraizarse más tarde basándose en nuevos conocimientos o dejarse sin enraizar. Esto se debe a que no existe una especie "especial", todas las especies eventualmente derivarán unas de otras con la misma probabilidad.
Un modelo es reversible en el tiempo si y sólo si satisface la propiedad (la notación se explica a continuación)
o, de manera equivalente, la propiedad del saldo detallado ,
para cada i , j y t .
La reversibilidad del tiempo no debe confundirse con la estacionariedad . Un modelo es estacionario si Q no cambia con el tiempo. El siguiente análisis supone un modelo estacionario.
Los modelos de sitios finitos, neutrales, independientes y estacionarios (suponiendo una tasa de evolución constante) tienen dos parámetros, π , un vector de equilibrio de frecuencias de bases (o caracteres) y una matriz de tasas, Q , que describe la tasa a la que las bases de un tipo cambiar a bases de otro tipo; El elemento para i ≠ j es la velocidad a la que la base i pasa a la base j . Las diagonales de la matriz Q se eligen de modo que las filas sumen cero:
El vector fila de equilibrio π debe ser aniquilado por la matriz de tasas Q :
La función de matriz de transición es una función de las longitudes de las ramas (en algunas unidades de tiempo, posiblemente en sustituciones) a una matriz de probabilidades condicionales. Se denota . La entrada en la i -ésima columna y la j -ésima fila , es la probabilidad, después del tiempo t , de que haya una base j en una posición dada, condicionada a que haya una base i en esa posición en el tiempo 0. Cuando el modelo Es reversible en el tiempo, esto se puede realizar entre dos secuencias cualesquiera, incluso si una no es el antepasado de la otra, si se conoce la longitud total de la rama entre ellas.
Las propiedades asintóticas de P ij (t) son tales que P ij (0) = δ ij , donde δ ij es la función delta de Kronecker . Es decir, no hay ningún cambio en la composición de bases entre una secuencia y ella misma. En el otro extremo, o, en otras palabras, a medida que el tiempo llega al infinito, la probabilidad de encontrar la base j en una posición dada que originalmente había una base i en esa posición va a la probabilidad de equilibrio de que haya una base j en esa posición, independientemente de de la base original. Además, se deduce que para todo t .
La matriz de transición se puede calcular a partir de la matriz de tasas mediante la exponenciación matricial :
donde Q n es la matriz Q multiplicada por sí misma suficientes veces para dar su enésima potencia .
Si Q es diagonalizable , la matriz exponencial se puede calcular directamente: sea Q = U −1 Λ U una diagonalización de Q , con
donde Λ es una matriz diagonal y donde están los valores propios de Q , cada uno repetido según su multiplicidad. Entonces
donde la matriz diagonal e Λt está dada por
El tiempo generalizado reversible (GTR) es el modelo neutral, independiente, de sitios finitos y reversible en el tiempo más general posible. Fue descrito por primera vez de forma general por Simon Tavaré en 1986. [22] El modelo GTR a menudo se denomina modelo general reversible en el tiempo en las publicaciones; [23] también se le ha llamado modelo REV. [24]
Los parámetros de GTR para nucleótidos consisten en un vector de frecuencia de bases de equilibrio, que proporciona la frecuencia a la que ocurre cada base en cada sitio, y la matriz de tasas.
Debido a que el modelo debe ser reversible en el tiempo y debe acercarse a las frecuencias de nucleótidos (bases) de equilibrio en tiempos prolongados, cada tasa por debajo de la diagonal es igual a la tasa recíproca por encima de la diagonal multiplicada por la relación de equilibrio de las dos bases. Como tal, el nucleótido GTR requiere 6 parámetros de tasa de sustitución y 4 parámetros de frecuencia de base de equilibrio. Dado que los 4 parámetros de frecuencia deben sumar 1, solo hay 3 parámetros de frecuencia libres. El total de 9 parámetros libres a menudo se reduce aún más a 8 parámetros más el número total de sustituciones por unidad de tiempo. Al medir el tiempo en sustituciones ( =1) sólo quedan 8 parámetros libres.
En general, para calcular el número de parámetros, se cuenta el número de entradas por encima de la diagonal en la matriz, es decir, para n valores de rasgos por sitio , y luego se suma n-1 para las frecuencias de equilibrio y se resta 1 porque es fijo. obtienes
Por ejemplo, para una secuencia de aminoácidos (hay 20 aminoácidos "estándar" que forman las proteínas ), encontrará que hay 208 parámetros. Sin embargo, cuando se estudian regiones codificantes del genoma, es más común trabajar con un modelo de sustitución de codones (un codón tiene tres bases y codifica un aminoácido en una proteína). Hay codones, lo que da como resultado 2078 parámetros libres. Sin embargo, a menudo se supone que las tasas de transiciones entre codones que difieren en más de una base son cero, lo que reduce el número de parámetros libres a sólo parámetros. Otra práctica común es reducir el número de codones prohibiendo los codones de terminación (o sin sentido ). Esta es una suposición biológicamente razonable porque incluir los codones de terminación significaría que uno está calculando la probabilidad de encontrar un codón de sentido después de un tiempo, dado que el codón ancestral implicaría la posibilidad de pasar por un estado con un codón de terminación prematuro.
Una forma alternativa (y comúnmente utilizada [23] [25] [26] [27] ) de escribir la matriz de tasas instantáneas ( matriz) para el modelo GTR de nucleótidos es:
La matriz está normalizada entonces .
Esta notación es más fácil de entender que la notación utilizada originalmente por Tavaré , porque todos los parámetros del modelo corresponden a parámetros de "intercambiabilidad" ( a través de , que también se pueden escribir usando la notación ) o a frecuencias de nucleótidos de equilibrio . Tenga en cuenta que los nucleótidos de la matriz se han escrito en orden alfabético. En otras palabras, la matriz de probabilidad de transición para la matriz anterior sería:
Algunas publicaciones escriben los nucleótidos en un orden diferente (p. ej., algunos autores optan por agrupar dos purinas y dos pirimidinas juntas; ver también modelos de evolución del ADN ). Estas diferencias de notación hacen que sea importante tener claro el orden de los estados al escribir la matriz.
El valor de esta notación es que la tasa de cambio instantáneo de un nucleótido a otro siempre se puede escribir como , donde es la intercambiabilidad de los nucleótidos y y es la frecuencia de equilibrio del nucleótido. La matriz que se muestra arriba utiliza las letras para los parámetros de intercambiabilidad en aras de la legibilidad, pero esos parámetros también podrían escribirse de manera sistemática usando la notación (por ejemplo, , etc.).
Tenga en cuenta que el orden de los subíndices de nucleótidos para los parámetros de intercambiabilidad es irrelevante (por ejemplo, ), pero los valores de la matriz de probabilidad de transición no lo son (es decir, es la probabilidad de observar A en la secuencia 1 y C en la secuencia 2 cuando la distancia evolutiva entre esas secuencias es mientras que es la probabilidad de observar C en la secuencia 1 y A en la secuencia 2 a la misma distancia evolutiva).
Un parámetro de intercambiabilidad elegido arbitrariamente (por ejemplo, ) normalmente se establece en un valor de 1 para aumentar la legibilidad de las estimaciones de los parámetros de intercambiabilidad (ya que permite a los usuarios expresar esos valores en relación con el parámetro de intercambiabilidad elegido). La práctica de expresar los parámetros de intercambiabilidad en términos relativos no es problemática porque la matriz está normalizada. La normalización permite que (el tiempo) en la exponenciación de la matriz se exprese en unidades de sustituciones esperadas por sitio (práctica estándar en filogenética molecular). Esto equivale a decir que se establece la tasa de mutación en 1) y se reduce el número de parámetros libres a ocho. Específicamente, hay cinco parámetros de intercambiabilidad libre ( a través de , que se expresan en relación con el fijo en este ejemplo) y tres parámetros de frecuencia base de equilibrio (como se describió anteriormente, solo es necesario especificar tres valores porque deben sumar 1).
La notación alternativa también facilita la comprensión de los submodelos del modelo GTR, que simplemente corresponden a casos en los que los parámetros de intercambiabilidad y/o frecuencia base de equilibrio están obligados a tomar valores iguales. Se han nombrado varios submodelos específicos, en gran parte basándose en sus publicaciones originales:
Hay 203 formas posibles de restringir los parámetros de intercambiabilidad para formar submodelos de GTR, [34] que van desde los modelos JC69 [5] y F81 [28] (donde todos los parámetros de intercambiabilidad son iguales) hasta el SYM [33]. modelo y el modelo GTR [22] (o REV [24] ) completo (donde todos los parámetros de intercambiabilidad son gratuitos). Las frecuencias base de equilibrio normalmente se tratan de dos maneras diferentes: 1) todos los valores están obligados a ser iguales (es decir, ); o 2) todos los valores se tratan como parámetros libres. Aunque las frecuencias base de equilibrio pueden restringirse de otras maneras, la mayoría de las restricciones que vinculan algunos pero no todos los valores no son realistas desde un punto de vista biológico. La posible excepción es imponer la simetría de las hebras [35] (es decir, restringir pero permitir ).
La notación alternativa también hace que sea sencillo ver cómo se puede aplicar el modelo GTR a alfabetos biológicos con un espacio de estados más grande (por ejemplo, aminoácidos o codones ). Es posible escribir un conjunto de frecuencias de estados de equilibrio como , , ... y un conjunto de parámetros de intercambiabilidad ( ) para cualquier alfabeto de estados de caracteres. Luego, estos valores se pueden usar para completar la matriz estableciendo los elementos fuera de la diagonal como se muestra arriba (la notación general sería ), estableciendo los elementos diagonales en la suma negativa de los elementos fuera de la diagonal en la misma fila y normalizando. Obviamente, para aminoácidos y codones ( asumiendo el código genético estándar ). Sin embargo, la generalidad de esta notación es beneficiosa porque se pueden utilizar alfabetos reducidos para los aminoácidos. Por ejemplo, se pueden utilizar y codificar aminoácidos recodificando los aminoácidos utilizando las seis categorías propuestas por Margaret Dayhoff . Los alfabetos de aminoácidos reducidos se consideran una forma de reducir el impacto de la variación y saturación de la composición. [36]
Es importante destacar que los patrones evolutivos pueden variar entre regiones genómicas y, por lo tanto, diferentes regiones genómicas pueden encajar con diferentes modelos de sustitución. [37] En realidad, ignorar los patrones evolutivos heterogéneos a lo largo de las secuencias puede generar sesgos en la estimación de los parámetros evolutivos, incluida la relación K a /K s . En este sentido, el uso de modelos mixtos en marcos filogenénticos es conveniente para imitar mejor la evolución molecular observada en datos reales. [38]
Una diferencia principal entre los modelos evolutivos es cuántos parámetros se estiman cada vez para el conjunto de datos considerado y cuántos de ellos se estiman una vez en un conjunto de datos grande. Los modelos mecanicistas describen todas las sustituciones en función de una serie de parámetros que se estiman para cada conjunto de datos analizado, preferiblemente utilizando la máxima verosimilitud . Esto tiene la ventaja de que el modelo puede ajustarse a las particularidades de un conjunto de datos específico (por ejemplo, diferentes sesgos de composición en el ADN). Pueden surgir problemas cuando se utilizan demasiados parámetros, especialmente si pueden compensarse entre sí (esto puede conducir a la no identificabilidad [39] ). Entonces ocurre con frecuencia que el conjunto de datos es demasiado pequeño para producir suficiente información para estimar todos los parámetros con precisión.
Los modelos empíricos se crean estimando muchos parámetros (normalmente todas las entradas de la matriz de tasas, así como las frecuencias de los caracteres, consulte el modelo GTR más arriba) a partir de un gran conjunto de datos. Luego, estos parámetros se fijan y se reutilizarán para cada conjunto de datos. Esto tiene la ventaja de que esos parámetros se pueden estimar con mayor precisión. Normalmente, no es posible estimar todas las entradas de la matriz de sustitución únicamente a partir del conjunto de datos actual. La desventaja es que los parámetros estimados a partir de los datos de entrenamiento pueden ser demasiado genéricos y, por lo tanto, no se ajustan bien a ningún conjunto de datos en particular. Una posible solución a ese problema es estimar algunos parámetros a partir de los datos utilizando la máxima verosimilitud (o algún otro método). En estudios de evolución de proteínas, las frecuencias de equilibrio de los aminoácidos (utilizando los códigos de una letra IUPAC para aminoácidos para indicar sus frecuencias de equilibrio) a menudo se estiman a partir de los datos [40] manteniendo fija la matriz de intercambiabilidad. Más allá de la práctica común de estimar las frecuencias de aminoácidos a partir de los datos, se han propuesto métodos para estimar los parámetros de intercambiabilidad [41] o ajustar la matriz [42] para la evolución de las proteínas de otras maneras.
Dado que la secuenciación del genoma a gran escala todavía produce cantidades muy grandes de secuencias de ADN y proteínas, hay suficientes datos disponibles para crear modelos empíricos con cualquier número de parámetros, incluidos modelos empíricos de codones. [43] Debido a los problemas mencionados anteriormente, los dos enfoques a menudo se combinan, estimando la mayoría de los parámetros una vez en datos a gran escala, mientras que algunos parámetros restantes luego se ajustan al conjunto de datos bajo consideración. Las siguientes secciones ofrecen una descripción general de los diferentes enfoques adoptados para modelos basados en ADN, proteínas o codones.
El primer modelo de evolución del ADN fue propuesto por Jukes y Cantor [5] en 1969. El modelo Jukes-Cantor (JC o JC69) supone tasas de transición iguales así como frecuencias de equilibrio iguales para todas las bases y es el submodelo más simple de la Modelo GTR. En 1980, Motoo Kimura introdujo un modelo con dos parámetros (K2P o K80 [29] ): uno para la transición y otro para la tasa de transversión . Un año después, Kimura introdujo un segundo modelo (K3ST, K3P o K81 [31] ) con tres tipos de sustitución: uno para la tasa de transición , otro para la tasa de transversiones que conservan las propiedades fuertes/débiles de los nucleótidos ( y , denominado por Kimura [31] ), y uno para la tasa de transversiones que conservan las propiedades amino/ceto de los nucleótidos ( y , designado por Kimura [31] ). En 1981, Joseph Felsenstein propuso un modelo de cuatro parámetros (F81 [28] ) en el que la tasa de sustitución corresponde a la frecuencia de equilibrio del nucleótido objetivo. Hasegawa, Kishino y Yano unificaron los dos últimos modelos en un modelo de cinco parámetros (HKY [30] ). Después de estos esfuerzos pioneros, en la década de 1990 se introdujeron en la literatura (y en el uso común) muchos submodelos adicionales del modelo GTR. [32] [33] Varios investigadores también desarrollaron y perfeccionaron otros modelos que van más allá del modelo GTR de maneras específicas. [44] [45]
Casi todos los modelos de sustitución de ADN son modelos mecanicistas (como se describe anteriormente). El pequeño número de parámetros que es necesario estimar para estos modelos hace factible estimar esos parámetros a partir de los datos. También es necesario porque los patrones de evolución de la secuencia de ADN a menudo difieren entre organismos y entre genes dentro de los organismos. Esto último puede reflejar optimización mediante la acción de selección para propósitos específicos (por ejemplo, expresión rápida o estabilidad del ARN mensajero) o podría reflejar variación neutral en los patrones de sustitución. Por tanto, dependiendo del organismo y del tipo de gen, probablemente sea necesario ajustar el modelo a estas circunstancias.
Una forma alternativa de analizar los datos de la secuencia de ADN es recodificar los nucleótidos como purinas (R) y pirimidinas (Y); [46] [47] esta práctica a menudo se denomina codificación RY. [48] Las inserciones y eliminaciones en múltiples alineamientos de secuencias también pueden codificarse como datos binarios [49] y analizarse utilizando un modelo de dos estados. [50] [51]
El modelo de evolución de secuencias de dos estados más simple se llama modelo de Cavender-Farris o modelo de Cavender-Farris- Neyman (CFN); El nombre de este modelo refleja el hecho de que fue descrito de forma independiente en varias publicaciones diferentes. [52] [53] [54] El modelo CFN es idéntico al modelo Jukes-Cantor adaptado a dos estados e incluso ha sido implementado como modelo "JC2" en el popular paquete de software IQ-TREE (usando este modelo en IQ -TREE requiere codificar los datos como 0 y 1 en lugar de R e Y; el popular paquete de software PAUP* puede interpretar una matriz de datos que comprende sólo R e Y como datos para analizar utilizando el modelo CFN). También es sencillo analizar datos binarios utilizando la transformada filogenética de Hadamard . [55] El modelo alternativo de dos estados permite que los parámetros de frecuencia de equilibrio de R e Y (o 0 y 1) tomen valores distintos de 0,5 añadiendo un único parámetro libre; este modelo se denomina indistintamente CFu [46] o GTR2 (en IQ-TREE).
Para muchos análisis, particularmente para distancias evolutivas más largas, la evolución se modela a nivel de aminoácidos. Dado que no todas las sustituciones de ADN alteran también el aminoácido codificado, se pierde información al observar aminoácidos en lugar de bases de nucleótidos. Sin embargo, varias ventajas hablan a favor del uso de la información de los aminoácidos: el ADN es mucho más propenso a mostrar un sesgo compositivo que los aminoácidos, no todas las posiciones en el ADN evolucionan a la misma velocidad ( es menos probable que las mutaciones no sinónimas queden fijadas en población que los sinónimos ), pero probablemente lo más importante es que debido a esas posiciones de rápida evolución y al tamaño limitado del alfabeto (sólo cuatro estados posibles), el ADN sufre más sustituciones hacia atrás, lo que dificulta estimar con precisión distancias evolutivas más largas.
A diferencia de los modelos de ADN, los modelos de aminoácidos son tradicionalmente modelos empíricos. Fueron pioneros en las décadas de 1960 y 1970 por Dayhoff y sus colaboradores al estimar tasas de reemplazo a partir de alineamientos de proteínas con al menos un 85 % de identidad (originalmente con datos muy limitados [56] y finalmente culminando en el modelo Dayhoff PAM de 1978 [57] ). . Esto minimizó las posibilidades de observar múltiples sustituciones en un sitio. A partir de la matriz de tasas estimadas, se derivaron una serie de matrices de probabilidad de reemplazo, conocidas con nombres como PAM 250. Las matrices logarítmicas de probabilidades basadas en el modelo PAM de Dayhoff se usaban comúnmente para evaluar la importancia de los resultados de la búsqueda de homología, aunque las matrices BLOSUM [ 58] han reemplazado las matrices de probabilidades logarítmicas PAM en este contexto porque las matrices BLOSUM parecen ser más sensibles en una variedad de distancias evolutivas, a diferencia de las matrices de probabilidades logarítmicas PAM . [59]
La matriz PAM de Dayhoff fue la fuente de los parámetros de intercambiabilidad utilizados en uno de los primeros análisis de filogenia de máxima verosimilitud que utilizó datos de proteínas [60] y el modelo PAM (o una versión mejorada del modelo PAM llamada DCMut [61] ) continúa para ser utilizado en filogenética. Sin embargo, el número limitado de alineamientos utilizados para generar el modelo PAM (que refleja la cantidad limitada de datos de secuencia disponibles en la década de 1970) casi con certeza infló la varianza de algunos parámetros de la matriz de tasas (alternativamente, las proteínas utilizadas para generar el modelo PAM podrían haber sido un conjunto no representativo). Independientemente, está claro que el modelo PAM rara vez se ajusta tan bien a la mayoría de los conjuntos de datos como los modelos empíricos más modernos (Keane et al. 2006 [62] probaron miles de proteínas de vertebrados , bacterias y arqueas y encontraron que el modelo PAM de Dayhoff modelo tenía el mejor ajuste a como máximo <4% de las proteínas).
A partir de la década de 1990, la rápida expansión de las bases de datos de secuencias debido a las tecnologías de secuenciación mejoradas condujo a la estimación de muchas matrices empíricas nuevas (consulte [63] para obtener una lista completa). Los primeros esfuerzos utilizaron métodos similares a los utilizados por Dayhoff, utilizando coincidencias a gran escala de la base de datos de proteínas para generar una nueva matriz de probabilidades logarítmicas [64] y el modelo JTT (Jones-Taylor-Thornton). [65] Los rápidos aumentos en la potencia de cálculo durante este tiempo (que reflejan factores como la ley de Moore ) hicieron posible estimar parámetros para modelos empíricos utilizando la máxima verosimilitud (por ejemplo, los modelos WAG [40] y LG [66] ) y otros métodos. (p. ej., los modelos VT [67] y PMB [68] ). El paquete de software IQ-Tree permite a los usuarios inferir su propio modelo reversible en el tiempo usando QMaker, [69] o no reversible en el tiempo usando nQMaker. [70]
En 1997, Tuffley y Steel [71] describieron un modelo al que denominaron modelo sin mecanismo común (NCM). La topología del árbol de máxima verosimilitud para un conjunto de datos específico dado el modelo NCM es idéntica a la topología del árbol óptimo para los mismos datos dado el criterio de máxima parsimonia . El modelo NCM supone que todos los datos (p. ej., nucleótidos homólogos, aminoácidos o caracteres morfológicos) están relacionados por un árbol filogenético común. Luego se introducen parámetros para cada carácter homólogo, donde está el número de secuencias. Esto puede verse como una estimación de un parámetro de tasa separado para cada par de carácter × rama en el conjunto de datos (tenga en cuenta que el número de ramas en un árbol filogenético completamente resuelto es ). Por lo tanto, el número de parámetros libres en el modelo NCM siempre excede el número de caracteres homólogos en la matriz de datos, y el modelo NCM ha sido criticado por estar constantemente "sobreparametrizado". [72]
Un buen modelo para la inferencia filogenética debe ser lo suficientemente rico como para abordar las fuentes de ruido en los datos, pero la estimación de ML realizada utilizando modelos que están claramente sobreparametrizados puede llevar a conclusiones drásticamente erróneas. El modelo NCM ciertamente cae en el ámbito de ser demasiado rico en parámetros para servir como justificación del uso de la parsimonia basándose en que es un estimador ML bajo un modelo general.