
Las interacciones glicano-proteína representan una clase de interacciones biomoleculares que ocurren entre glicanos libres o unidos a proteínas y sus socios de unión afines. Las interacciones intramoleculares glicano-proteína (proteína-glicano) ocurren entre los glicanos y las proteínas a las que están unidos covalentemente. Junto con las interacciones proteína-proteína , forman una base mecanicista para muchos procesos celulares esenciales , especialmente para las interacciones célula-célula y las interacciones célula huésped. [2] Por ejemplo, el SARS-CoV-2 , el agente causante de la COVID-19 , emplea su proteína de pico (S) ampliamente glicosilada para unirse al receptor ACE2 , permitiéndole ingresar a las células huésped. [3] La proteína de pico es una estructura trimérica , y cada subunidad contiene 22 sitios de N-glicosilación , lo que la convierte en un objetivo atractivo para la búsqueda de vacunas . [3] [4]
La glicosilación, es decir, la adición de glicanos (un nombre genérico para monosacáridos y oligosacáridos ) a una proteína, es una de las principales modificaciones postraduccionales de las proteínas que contribuyen a la enorme complejidad biológica de la vida. De hecho, en teoría, tres hexosas diferentes podrían producir de 1.056 a 27.648 trisacáridos únicos, en contraste con sólo 6 péptidos u oligonucleótidos formados a partir de 3 aminoácidos o 3 nucleótidos respectivamente. [2] A diferencia de la biosíntesis de proteínas basada en plantillas , el "lenguaje" de la glicosilación aún se desconoce, lo que hace que la glicobiología sea un tema candente de investigación actual dada su prevalencia en los organismos vivos. [2]
El estudio de las interacciones glicano-proteína proporciona información sobre los mecanismos de señalización celular y permite crear mejores herramientas de diagnóstico para muchas enfermedades, incluido el cáncer . De hecho, no se conocen tipos de cáncer que no impliquen patrones erráticos de glicosilación de proteínas . [5]
La unión de las proteínas de unión a glicanos (GBP) a los glicanos podría modelarse con un equilibrio simple . Denotando glicanos como y proteínas como :
Con una constante de equilibrio asociada de
Que se reordena para dar constante de disociación siguiendo convenciones bioquímicas:
Dado que muchas libras esterlinas exhiben multivalencia, este modelo puede ampliarse para tener en cuenta equilibrios múltiples:
Denotando equilibrio acumulativo de unión con ligandos como
Con la constante de equilibrio correspondiente:
Y escribiendo el balance de materia de proteínas ( denota la concentración total de proteínas):
Expresando los términos mediante una constante de equilibrio, se encuentra un resultado final:
La concentración de proteína libre es entonces:
Si , es decir, solo hay un dominio receptor de carbohidratos, la ecuación se reduce a
Al aumentar la concentración de proteína libre disminuye; por lo tanto, el aparente también disminuye.

La intuición química sugiere que los sitios de unión de glicanos pueden estar enriquecidos en residuos de aminoácidos polares que forman interacciones no covalentes , como enlaces de hidrógeno , con carbohidratos polares . De hecho, un análisis estadístico de las bolsas de unión a carbohidratos muestra que el ácido aspártico y los residuos de asparagina están presentes con el doble de frecuencia de lo que se podría predecir por casualidad. [6] Sorprendentemente, existe una preferencia aún mayor por los aminoácidos aromáticos : el triptófano tiene una prevalencia 9 veces mayor, la tirosina 3 veces y la histidina un aumento 2 veces. Se ha demostrado que la fuerza subyacente es la interacción entre el sistema aromático y los carbohidratos, como se muestra en la Figura 1 . La interacción se identifica si el °, la distancia (distancia de a ) es menor que 4,5 Å. [6]

Esta interacción depende en gran medida de la estereoquímica de la molécula de carbohidrato . Por ejemplo, considere las caras superior ( ) e inferior ( ) de -D-Glucosa y -D-Galactosa . Se ha demostrado que un solo cambio en la estereoquímica en el carbono C4 desplaza la preferencia por los residuos aromáticos de un lado (2,7 veces de preferencia por la glucosa) a un lado (14 veces de preferencia por la galactosa). [6]
La comparación de los potenciales de superficie electrostáticos (ESP) de los anillos aromáticos en triptófano , tirosina , fenilalanina e histidina sugiere que los efectos electrónicos también desempeñan un papel en la unión a los glicanos (ver Figura 2 ). Después de normalizar las densidades de electrones para el área de superficie, el triptófano sigue siendo el aceptor de interacciones más rico en electrones, lo que sugiere una posible razón para su prevalencia nueve veces mayor en las bolsas de unión a carbohidratos. [6] En general, los mapas de potencial electrostático siguen la tendencia de prevalencia de .

Existen muchas proteínas capaces de unirse a los glicanos, incluidas las lectinas , anticuerpos , adhesinas microbianas , aglutininas virales , etc.
Lectinas es un nombre genérico para proteínas con dominios de reconocimiento de carbohidratos (CRD). Aunque se ha convertido casi en sinónimo de proteínas de unión a glucanos, no incluye anticuerpos que también pertenecen a esta clase.
Las lectinas que se encuentran en células de plantas y hongos se han utilizado ampliamente en la investigación como herramienta para detectar, purificar y analizar glicanos. Sin embargo, las lectinas útiles suelen tener especificidades subóptimas . Por ejemplo,Ulex europaeus aglutinina-1 (UEA-1), una lectina extraída de plantas capaz de unirse al antígeno del tipo O de la sangre humana , también puede unirse a glicanos no relacionados como 2'-fucosillactosa, GalNAcα1-4(Fucα1-2)Galβ1-4GlcNAc y antígeno Lewis-Y . [7]
Aunque los anticuerpos exhiben afinidades nanomolares hacia los antígenos proteicos, la especificidad contra los glicanos es muy limitada. [8] De hecho, los anticuerpos disponibles pueden unirse sólo a <4% de los 7000 antígenos de glucano de mamíferos; además, la mayoría de esos anticuerpos tienen baja afinidad y presentan reactividad cruzada. [9] [7]
A diferencia de los vertebrados con mandíbulas, cuya inmunidad se basa en segmentos genéticos variables, diversos y unidos (VDJ) de inmunoglobulinas , los invertebrados sin mandíbulas , como la lamprea y el mixino , crean una diversidad de receptores mediante la reordenación somática del ADN de las repeticiones ricas en leucina (LRR). Módulos que se incorporan en genes *vlr* (receptores leucocitarios variables). [10] Esos LRR forman estructuras 3D que se asemejan a solenoides curvos que se unen selectivamente a glicanos específicos. [11]
Un estudio de la Universidad de Maryland ha demostrado que los anticuerpos contra la lamprea (lambodies) podrían unirse selectivamente a antígenos de carbohidratos asociados a tumores (como Tn y TF ) con afinidades nanomolares. [9] El antígeno T-nouvelle (Tn) y TF están presentes en proteínas en hasta el 90% de diferentes células cancerosas después de una modificación postraduccional , mientras que en las células sanas esos antígenos son mucho más complejos. Una selección de lambodies que podrían unirse a aGPA , una glicoproteína de la membrana de los eritrocitos humanos que está cubierta con 16 restos TF, mediante clasificación de células activadas magnéticamente (MACS) y clasificación de células activadas por fluorescencia (FACS), ha producido un lambody VLRB rico en leucina . aGPA.23 . Este lambody tiñó selectivamente (sobre muestras sanas) células de 14 tipos diferentes de adenocarcinomas : vejiga , esófago , ovario , lengua , mejilla, cuello uterino , hígado , nariz, nasofaringe , epiplón mayor, colon , mama , laringe y pulmón . [9] Además, los pacientes cuyos tejidos se tiñeron positivamente con VLRB.aGPA.23 tuvieron una tasa de supervivencia significativamente menor. [9]
Una mirada de cerca a la estructura cristalina de VLRB.aGPA.23 revela un residuo de triptófano en la posición 187, justo encima del bolsillo de unión a carbohidratos. [12]

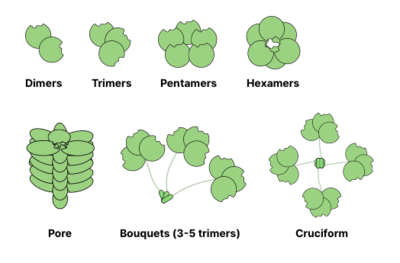
Muchas proteínas de unión a glucanos (GBP) son oligoméricas y normalmente contienen múltiples sitios para la unión de glucanos (también llamados dominios de reconocimiento de carbohidratos). La capacidad de formar interacciones multivalentes proteína- ligando mejora significativamente la fuerza de unión: si bien los valores para las interacciones CRD-glicano individuales pueden estar en el rango mM, la afinidad general de GBP hacia los glicanos puede alcanzar rangos nanomolares o incluso picomolares . La fuerza general de las interacciones se describe como avidez (en contraste con una afinidad que describe un equilibrio único). A veces, la avidez también se denomina aparente para enfatizar la naturaleza de desequilibrio de la interacción. [13]
A continuación se muestran las estructuras de oligomerización comunes de las lectinas . Por ejemplo, las galectinas suelen observarse como dímeros, mientras que las intelectinas forman trímeros y las pentraxinas se ensamblan en pentámeros. Las estructuras más grandes, como las proteínas Reg hexaméricas , pueden ensamblarse en poros que penetran la membrana. Las colectinas pueden formar complejos aún más extraños: ramos de trímeros o incluso estructuras tipo cruciforme (por ejemplo, en SP-D ). [14]
Dada la importancia de las interacciones glucano-proteína, hay una investigación en curso dedicada a a) la creación de nuevas herramientas para detectar interacciones glucano-proteína y b) el uso de esas herramientas para descifrar el llamado código del azúcar.
Una de las herramientas más utilizadas para investigar las interacciones entre glucanos y proteínas son las matrices de glucanos . Una matriz de glucanos suele ser portaobjetos de vidrio activados con NHS o epoxi en los que se imprimieron varios glucanos mediante impresión robótica. [15] [16] Estas matrices disponibles comercialmente pueden contener hasta 600 glicanos diferentes, cuya especificidad se ha estudiado ampliamente. [17]
Las interacciones entre glicanos y proteínas se pueden detectar analizando proteínas de interés (o bibliotecas de ellas) que llevan etiquetas fluorescentes . La estructura de la proteína de unión a glucano se puede descifrar mediante varios métodos analíticos basados en espectrometría de masas , incluidos MALDI-MS , LC-MS , MS-MS en tándem y/o RMN 2D . [18]
Se han aplicado métodos computacionales para buscar parámetros (p. ej., propensión a residuos, hidrofobicidad, planaridad) que podrían distinguir las proteínas de unión a glucanos de otros parches de superficie. Por ejemplo, un modelo entrenado en 19 estructuras de unión a carbohidratos no homólogas fue capaz de predecir dominios de unión a carbohidratos (CRD) con una precisión del 65 % para las estructuras no enzimáticas y del 87 % para las enzimáticas. [19] Estudios adicionales han empleado cálculos de energías de Van der Waals de interacciones proteína-sonda y propensiones de aminoácidos para identificar CRD con 98% de especificidad y 73% de sensibilidad . [20] Los métodos más recientes pueden predecir las CRD incluso a partir de secuencias de proteínas , comparando la secuencia con aquellas cuyas estructuras ya se conocen. [21]
A diferencia de los estudios de proteínas, donde la estructura primaria de la proteína está claramente definida por la secuencia de nucleótidos (el código genético ), la glicobiología aún no puede explicar cómo se codifica un determinado "mensaje" mediante carbohidratos o cómo se "lee" y "traduce". " por otras entidades biológicas.
Un esfuerzo interdisciplinario, que combina química, biología y bioquímica, estudia las interacciones entre glicanos y proteínas para ver cómo diferentes secuencias de carbohidratos inician diferentes respuestas celulares. [22]
{{cite book}}: Mantenimiento CS1: DOI inactivo a partir de enero de 2024 ( enlace ){{cite book}}: Mantenimiento CS1: DOI inactivo a partir de enero de 2024 ( enlace )