
La síntesis de ADN es la creación natural o artificial de moléculas de ácido desoxirribonucleico (ADN). El ADN es una macromolécula formada por unidades de nucleótidos , que están unidas por enlaces covalentes y enlaces de hidrógeno, en una estructura repetitiva. La síntesis de ADN ocurre cuando estas unidades de nucleótidos se unen para formar ADN; esto puede ocurrir artificialmente ( in vitro ) o naturalmente ( in vivo ). Las unidades de nucleótidos están formadas por una base nitrogenada (citosina, guanina, adenina o timina), azúcar pentosa (desoxirribosa) y un grupo fosfato. Cada unidad se une cuando se forma un enlace covalente entre su grupo fosfato y el azúcar pentosa del siguiente nucleótido, formando una cadena principal de azúcar-fosfato. El ADN es una estructura complementaria de doble cadena, ya que el apareamiento de bases específicas (adenina y timina, guanina y citosina) ocurre naturalmente cuando se forman enlaces de hidrógeno entre las bases de los nucleótidos.
Existen varias definiciones diferentes para la síntesis de ADN: puede referirse a la replicación del ADN - biosíntesis del ADN ( amplificación del ADN in vivo ), reacción en cadena de la polimerasa - síntesis enzimática del ADN ( amplificación del ADN in vitro ) o síntesis génica - creación física de secuencias génicas artificiales . Aunque cada tipo de síntesis es muy diferente, comparten algunas características. Los nucleótidos que se han unido para formar polinucleótidos pueden actuar como una plantilla de ADN para que se produzca una forma de síntesis de ADN - PCR -. La replicación del ADN también funciona utilizando una plantilla de ADN, la doble hélice del ADN se desenrolla durante la replicación, exponiendo bases no apareadas para que los nuevos nucleótidos formen enlaces de hidrógeno. La síntesis génica, sin embargo, no requiere una plantilla de ADN y los genes se ensamblan de novo .
La síntesis de ADN se produce en todos los eucariotas y procariotas , así como en algunos virus . La síntesis precisa de ADN es importante para evitar mutaciones en el ADN. En los seres humanos, las mutaciones pueden provocar enfermedades como el cáncer, por lo que la síntesis de ADN y la maquinaria implicada in vivo se han estudiado ampliamente a lo largo de las décadas. En el futuro, estos estudios pueden utilizarse para desarrollar tecnologías que impliquen la síntesis de ADN, para su uso en el almacenamiento de datos.


En la naturaleza, las moléculas de ADN son sintetizadas por todas las células vivas a través del proceso de replicación del ADN . Esto ocurre típicamente como parte de la división celular . La replicación del ADN ocurre de modo que, durante la división celular, cada célula hija contiene una copia exacta del material genético de la célula. La síntesis de ADN in vivo ( replicación del ADN ) depende de un conjunto complejo de enzimas que han evolucionado para actuar durante la fase S del ciclo celular, de manera concertada. Tanto en eucariotas como en procariotas , la replicación del ADN ocurre cuando topoisomerasas , helicasas y girasas específicas (proteínas iniciadoras de la replicación) desenrollan el ADN bicatenario, exponiendo las bases nitrogenadas. [1] Estas enzimas, junto con las proteínas accesorias, forman una máquina macromolecular que asegura la duplicación precisa de las secuencias de ADN. Se produce el apareamiento de bases complementarias, formando una nueva molécula de ADN bicatenario. Esto se conoce como replicación semiconservativa , ya que una hebra de la nueva molécula de ADN proviene de la hebra "madre".
Las enzimas eucariotas sufren continuamente daños en el ADN que pueden perturbar su replicación. Estos daños se manifiestan en forma de lesiones en el ADN que surgen espontáneamente o debido a agentes que dañan el ADN. Por lo tanto, la maquinaria de replicación del ADN está muy controlada para evitar que colapse cuando se produce un daño. [2] El control del sistema de replicación del ADN garantiza que el genoma se replique solo una vez por ciclo; la sobrerreplicación induce daños en el ADN. La desregulación de la replicación del ADN es un factor clave en la inestabilidad genómica durante el desarrollo del cáncer. [3]
Esto pone de relieve la especificidad de la maquinaria de síntesis de ADN in vivo . Existen diversos medios para estimular artificialmente la replicación del ADN natural o para crear secuencias genéticas artificiales. Sin embargo, la síntesis de ADN in vitro puede ser un proceso muy propenso a errores.
El ADN dañado está sujeto a reparación por varios procesos de reparación enzimática diferentes , donde cada proceso individual está especializado para reparar tipos particulares de daño. El ADN de los humanos está sujeto a daños de múltiples fuentes naturales y la reparación insuficiente está asociada con enfermedades y envejecimiento prematuro . [4] La mayoría de los procesos de reparación del ADN forman huecos de cadena simple en el ADN durante una etapa intermedia de la reparación, y estos huecos se llenan mediante síntesis de reparación. [4] Los procesos de reparación específicos que requieren el llenado de huecos mediante síntesis de ADN incluyen reparación por escisión de nucleótidos , reparación por escisión de bases , reparación de desajustes , reparación por recombinación homóloga , unión de extremos no homólogos y unión de extremos mediada por microhomología .
La transcripción inversa es parte del ciclo de replicación de determinadas familias de virus, incluidos los retrovirus . Implica copiar el ARN en ADN complementario de doble cadena (ADNc), utilizando enzimas transcriptasas inversas . En los retrovirus, el ARN viral se inserta en el núcleo de una célula huésped. Allí, una enzima transcriptasa inversa viral agrega nucleótidos de ADN a la secuencia de ARN, generando ADNc que se inserta en el genoma de la célula huésped por la enzima integrasa , que codifica las proteínas virales. [5]
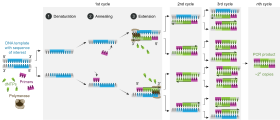
Una reacción en cadena de la polimerasa es una forma de síntesis enzimática de ADN en el laboratorio, que utiliza ciclos de calentamiento y enfriamiento repetidos de la reacción para la fusión del ADN y la replicación enzimática del ADN.
La síntesis de ADN durante la PCR es muy similar a la de las células vivas, pero se necesitan reactivos y condiciones muy específicos. Durante la PCR, el ADN se extrae químicamente de las proteínas chaperonas del huésped y luego se calienta, lo que provoca la disociación térmica de las cadenas de ADN. Se forman dos nuevas cadenas de ADNc a partir de la cadena original, que se pueden volver a dividir para que actúen como plantilla para otros productos de PCR. El ADN original se multiplica a través de muchas rondas de PCR. [1] Se pueden crear más de mil millones de copias de la cadena de ADN original.
Para muchos experimentos, como los estudios estructurales y evolutivos, los científicos necesitan producir una gran biblioteca de variantes de una secuencia de ADN particular. La mutagénesis aleatoria se lleva a cabo in vitro, cuando la replicación mutagénica con una ADN polimerasa de baja fidelidad se combina con la amplificación selectiva por PCR para producir muchas copias de ADN mutante. [6]
La RT-PCR se diferencia de la PCR convencional en que sintetiza ADNc a partir de ARNm, en lugar de ADN molde. La técnica combina una reacción de transcripción inversa con una amplificación basada en PCR, ya que una secuencia de ARN actúa como molde para la enzima transcriptasa inversa. La RT-PCR se utiliza a menudo para probar la expresión génica en determinados tejidos o tipos de células en diversas etapas de desarrollo o para detectar trastornos genéticos. [7]
La síntesis artificial de genes es el proceso de sintetizar un gen in vitro sin necesidad de muestras iniciales de ADN molde. En 2010, J. Craig Venter y su equipo fueron los primeros en utilizar ADN completamente sintetizado para crear un microbio autorreplicante, denominado Mycoplasma laboratorium . [8]
La síntesis de oligonucleótidos es la síntesis química de secuencias de ácidos nucleicos. La mayoría de las investigaciones biológicas y la bioingeniería implican ADN sintético, que puede incluir oligonucleótidos , genes sintéticos o incluso cromosomas . Hoy en día, la mayor parte del ADN sintético se crea a medida utilizando el método de fosforamidita de Marvin H. Caruthers . Los oligos se sintetizan a partir de bloques de construcción que replican bases naturales. Se han puesto a disposición comercial otras técnicas para sintetizar ADN, incluido el ensamblaje de ligadura de oligos cortos. [9] El proceso se ha automatizado desde fines de la década de 1970 y se puede utilizar para formar secuencias genéticas deseadas, así como para otros usos en medicina y biología molecular. Sin embargo, la creación química de secuencias es poco práctica más allá de 200-300 bases y es un proceso peligroso para el medio ambiente. Estos oligos, de alrededor de 200 bases, se pueden conectar utilizando métodos de ensamblaje de ADN, creando moléculas de ADN más grandes. [10]
Algunos estudios han explorado la posibilidad de la síntesis enzimática utilizando la desoxinucleotidil transferasa terminal (TdT), una polimerasa de ADN que no requiere una plantilla. Sin embargo, este método aún no es tan eficaz como la síntesis química y no está disponible comercialmente. [11]
Con los avances en la síntesis artificial de ADN, se está explorando la posibilidad de almacenar datos de ADN . Con su altísima densidad de almacenamiento y estabilidad a largo plazo, el ADN sintético es una opción interesante para almacenar grandes cantidades de datos. Aunque la información se puede recuperar muy rápidamente del ADN mediante tecnologías de secuenciación de última generación, la síntesis de ADN de novo es un importante obstáculo en el proceso. Solo se puede añadir un nucleótido por ciclo, y cada ciclo lleva segundos, por lo que la síntesis general requiere mucho tiempo y es muy propensa a errores. Sin embargo, si la biotecnología mejora, algún día se podría utilizar ADN sintético para almacenar datos. [12]
Se ha informado de que se pueden sintetizar nuevos pares de nucleobases , así como AT ( adenina - timina ) y GC ( guanina - citosina ). Los nucleótidos sintéticos se pueden utilizar para ampliar el alfabeto genético y permitir la modificación específica de los sitios de ADN. Incluso un tercer par de bases ampliaría el número de aminoácidos que pueden ser codificados por el ADN de los 20 aminoácidos existentes a un posible número de 172. [8] El ADN de Hachimoji se construye a partir de ocho letras de nucleótidos, formando cuatro posibles pares de bases. Por lo tanto, duplica la densidad de información del ADN natural. En estudios, incluso se ha producido ARN a partir del ADN de Hachimoji. Esta tecnología también podría utilizarse para permitir el almacenamiento de datos en el ADN. [13]