La regulación postranscripcional es el control de la expresión génica a nivel de ARN . Ocurre una vez que la ARN polimerasa se ha unido al promotor del gen y está sintetizando la secuencia de nucleótidos. Por tanto, como su nombre indica, se produce entre la fase de transcripción y la fase de traducción de la expresión génica . Estos controles son fundamentales para la regulación de muchos genes en los tejidos humanos. [1] [2] También juega un papel importante en la fisiología celular, estando implicado en patologías como el cáncer y enfermedades neurodegenerativas. [3]
Después de ser producidos, la estabilidad y distribución de los diferentes transcritos se regula (regulación postranscripcional) mediante proteínas de unión a ARN (RBP) que controlan los distintos pasos y velocidades controlando eventos como el empalme alternativo , la degradación nuclear ( exosoma ), el procesamiento. , exportación nuclear (tres vías alternativas), secuestro en cuerpos P para almacenamiento o degradación y, en última instancia, traducción . Estas proteínas logran estos eventos gracias a un motivo de reconocimiento de ARN (RRM) que se une a una secuencia específica o estructura secundaria de las transcripciones, generalmente en las UTR 5' y 3' de la transcripción. En resumen, las secuencias de ARNbc, que se descompondrán en ARNip dentro del organismo, coincidirán con el ARN para inhibir la expresión genética en la célula.
La modulación de la protección, el empalme, la adición de una cola Poly (A) , las tasas de exportación nuclear específicas de la secuencia y, en varios contextos, el secuestro de la transcripción de ARN ocurre en eucariotas pero no en procariotas . Esta modulación es el resultado de una proteína o transcrito que a su vez está regulado y puede tener afinidad por determinadas secuencias.
La atenuación de la transcripción es un tipo de regulación procariótica que ocurre sólo bajo ciertas condiciones. Este proceso ocurre al comienzo de la transcripción del ARN y hace que la cadena de ARN termine antes de la expresión genética. [5] La atenuación de la transcripción es causada por la formación incorrecta de una cadena de ARN naciente. Esta cadena de ARN naciente adopta una estructura secundaria alternativa que no interactúa adecuadamente con la ARN polimerasa . [1] Para que se produzca la expresión genética, las proteínas reguladoras deben unirse a la cadena de ARN y eliminar la atenuación, lo cual es costoso para la célula. [ dieciséis]
En los procariotas existen dos mecanismos de atenuación de la transcripción. Estos dos mecanismos son la terminación intrínseca y la terminación dependiente de factores.
- En el mecanismo de terminación intrínseca , también conocido como terminación independiente de Rho , la cadena de ARN forma una estructura de horquilla de transcripción estable en el extremo 3' de los genes que hacen que la ARN polimerasa deje de transcribir. [6] El bucle del tallo es seguido por una serie de U (cola de poli U) que detiene la polimerasa, por lo que la horquilla de ARN tiene tiempo suficiente para formarse. Luego, la polimerasa se disocia debido a la débil unión entre la cola poli U , del ARN transcrito, y la cola poli A, de la plantilla de ADN, provocando que el ARNm se libere prematuramente. Este proceso inhibe la transcripción. [7] Para aclarar, este mecanismo se llama independiente de Rho porque no requiere ningún factor proteico adicional como lo hace la terminación dependiente del factor, que es un mecanismo más simple para que la célula regule la transcripción de genes. [7] Algunos ejemplos de bacterias donde predomina este tipo de regulación son Neisseria, Psychrobacter y Pasteurellaceae , así como la mayoría de bacterias del filo Firmicutes. [7] [6]
- En la terminación dependiente del factor , que es un complejo de factor proteico que contiene el factor Rho , está unido a un segmento de transcripción de la cadena de ARN. Luego, el complejo Rho comienza a buscar en la dirección 3' una ARN polimerasa en pausa. Si se encuentra la polimerasa, el proceso se detiene inmediatamente, lo que provoca la interrupción de la transcripción del ARN. [5] [6] Aunque este sistema no es tan común como el descrito anteriormente, existen algunas bacterias que utilizan este tipo de terminación, como el operón tna en E.coli . [7]
Este tipo de regulación no es eficiente en eucariotas porque la transcripción ocurre en el núcleo mientras que la traducción ocurre en el citoplasma. Por lo tanto, el mecanismo no continúa y no puede ejecutarse adecuadamente como lo haría si ambos procesos ocurrieran en el citoplasma. [8]
Los microARN (miARN) parecen regular la expresión de más del 60% de los genes codificantes de proteínas del genoma humano. [9] Si un miARN es abundante, puede comportarse como un "interruptor", activando o desactivando algunos genes. [10] Sin embargo, la expresión alterada de muchos miARN solo conduce a un cambio modesto de 1,5 a 4 veces en la expresión de proteínas de sus genes diana. [10] Los miARN individuales a menudo reprimen varios cientos de genes diana. [9] [11] La represión generalmente ocurre mediante el silenciamiento traduccional del ARNm o mediante la degradación del ARNm, mediante unión complementaria, principalmente a secuencias específicas en la región 3' no traducida del ARNm del gen objetivo. [12] El mecanismo de silenciamiento traslacional o degradación del ARNm se implementa a través del complejo de silenciamiento inducido por ARN (RISC).
Las proteínas de unión a ARN (RBP) son ensamblajes dinámicos entre ARNm y diferentes proteínas que forman complejos de ribonucleoproteínas mensajeras (mRNP). [13] Estos complejos son esenciales para la regulación de la expresión genética para garantizar que todos los pasos se realicen correctamente durante todo el proceso. Por tanto, son importantes factores de control de los niveles de proteínas y los fenotipos celulares. Además, afectan la estabilidad del ARNm regulando su conformación debido al entorno, el estrés o las señales extracelulares. [13] Sin embargo, su capacidad para unirse y controlar una variedad tan amplia de objetivos de ARN les permite formar redes reguladoras complejas (PTRN). Estas redes representan un desafío para estudiar cada proteína de unión a ARN individualmente. [3] Afortunadamente, debido a los nuevos avances metodológicos, la identificación de RBP se está expandiendo lentamente, lo que demuestra que están contenidas en amplias familias de proteínas. Las RBP pueden afectar significativamente múltiples procesos biológicos y deben expresarse con mucha precisión. [7] La sobreexpresión puede cambiar la tasa objetivo del ARNm, uniéndose a sitios de ARN de baja afinidad y provocando resultados nocivos en la aptitud celular. No poder sintetizar al nivel correcto también es problemático porque puede provocar la muerte celular. Por lo tanto, las RBP están reguladas mediante autorregulación , por lo que tienen el control de sus propias acciones. Además, utilizan tanto retroalimentación negativa , para mantener la homeostasis, como retroalimentación positiva , para crear cambios genéticos binarios en la célula. [14]
En los metazoos y las bacterias, muchos genes implicados en la regulación postranscripcional están regulados postranscripcionalmente. [15] [16] [17] Para las RBP de Drosophila asociadas con empalme o descomposición mediada sin sentido, los análisis de los perfiles de interacción proteína-proteína y proteína-ARN han revelado interacciones ubicuas con ARN y productos proteicos del mismo gen. [17] Aún no está claro si estas observaciones están impulsadas por contactos ribosomales proximales o mediados por ribosomas, o si algunos complejos de proteínas, particularmente RNP, se someten a un ensamblaje cotraduccional.
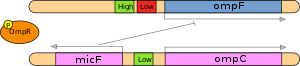
Esta área de estudio ha ganado recientemente más importancia debido a la creciente evidencia de que la regulación postranscripcional juega un papel más importante de lo que se esperaba anteriormente. Aunque las proteínas con dominios de unión al ADN son más abundantes que las proteínas con dominios de unión al ARN, un estudio reciente de Cheadle et al. (2005) demostraron que durante la activación de las células T, el 55% de los cambios significativos en el nivel de estado estacionario no tenían cambios correspondientes en el nivel transcripcional, lo que significa que eran resultado únicamente de la regulación de la estabilidad. [19]
Además, el ARN que se encuentra en el núcleo es más complejo que el que se encuentra en el citoplasma: más del 95% (bases) del ARN sintetizado por la ARN polimerasa II nunca llega al citoplasma . La razón principal de esto se debe a la eliminación de intrones que representan el 80% del total de bases. [20] Algunos estudios han demostrado que incluso después del procesamiento, los niveles de ARNm entre el citoplasma y el núcleo difieren mucho. [21]
La biología del desarrollo es una buena fuente de modelos de regulación, pero debido a dificultades técnicas fue más fácil determinar las cascadas de factores de transcripción que la regulación a nivel de ARN. De hecho, se sabe que varios genes clave, como los nanos, se unen al ARN, pero a menudo se desconocen sus objetivos. [22] Aunque las proteínas de unión al ARN pueden regular postranscripcionalmente una gran cantidad del transcriptoma, el direccionamiento de un solo gen es de interés para la comunidad científica por razones médicas, esto es la interferencia del ARN y los microARN , que son ambos ejemplos de regulación postranscripcional, que regulan la destrucción del ARN y cambiar la estructura de la cromatina. Para estudiar la regulación postranscripcional se utilizan varias técnicas, como RIP-Chip ( inmunoprecipitación de ARN en chip). [23]
La deficiencia de expresión de un gen de reparación del ADN ocurre en muchos cánceres (ver Defecto de reparación del ADN y riesgo de cáncer y Reparación de microARN y ADN ). En los cánceres se observa a menudo una expresión alterada de microARN (miARN) que disminuye la reparación precisa del ADN o aumenta la reparación inexacta del ADN por unión de extremos mediada por microhomología (MMEJ). La deficiencia en la reparación precisa del ADN puede ser una fuente importante de la alta frecuencia de mutaciones en el cáncer (ver frecuencias de mutaciones en los cánceres ). La represión de los genes de reparación del ADN en los cánceres mediante cambios en los niveles de microARN puede ser una causa más frecuente de represión que la mutación o la metilación epigenética de los genes de reparación del ADN.
Por ejemplo, BRCA1 se emplea en la vía específica de reparación recombinante homóloga (HR). La deficiencia de BRCA1 puede causar cáncer de mama. [24] La regulación negativa de BRCA1 debido a una mutación ocurre en aproximadamente el 3% de los cánceres de mama. [25] La regulación negativa de BRCA1 debido a la metilación de su promotor ocurre en aproximadamente el 14% de los cánceres de mama. [26] Sin embargo, el aumento de la expresión de miR-182 regula negativamente la expresión del ARNm y la proteína de BRCA1, [27] y un aumento de miR-182 se encuentra en el 80% de los cánceres de mama. [28]
En otro ejemplo, en muchos cánceres se encuentra una versión mutada expresada constitutivamente (persistentemente) del oncogén c-Myc . Entre muchas funciones, c-Myc regula negativamente los microARN miR-150 y miR-22. Estos microARN normalmente reprimen la expresión de dos genes esenciales para MMEJ, Lig3 y Parp1 , inhibiendo así esta vía de reparación del ADN mutagénica e imprecisa. Muvarak et al. [29] mostraron, en leucemias, que la expresión constitutiva de c-Myc, que conduce a una regulación negativa de miR-150 y miR-22, permitía una mayor expresión de Lig3 y Parp1 . Esto genera inestabilidad genómica a través de una mayor reparación imprecisa del ADN de MMEJ y probablemente contribuye a la progresión a leucemia.
Para mostrar la frecuente capacidad de los microARN para alterar la expresión de reparación del ADN, Hatano et al. [30] realizaron un gran estudio de detección, en el que se transfectaron 810 microARN en células que luego se sometieron a radiación ionizante (IR). Para 324 de estos microARN, la reparación del ADN se redujo (las células fueron destruidas de manera más eficiente por IR) después de la transfección. Para otros 75 microARN, la reparación del ADN aumentó, con menos muerte celular después de la IR. Esto indica que las alteraciones en los microARN a menudo pueden regular negativamente la reparación del ADN, un paso temprano probablemente importante en la progresión hacia el cáncer.
{{cite book}}: Mantenimiento CS1: falta el editor de la ubicación ( enlace )