
La clonación molecular es un conjunto de métodos experimentales en biología molecular que se utilizan para ensamblar moléculas de ADN recombinante y dirigir su replicación dentro de organismos huéspedes . [1] El uso de la palabra clonación se refiere al hecho de que el método implica la replicación de una molécula para producir una población de células con moléculas de ADN idénticas. La clonación molecular generalmente utiliza secuencias de ADN de dos organismos diferentes: la especie que es la fuente del ADN que se va a clonar y la especie que servirá como huésped vivo para la replicación del ADN recombinante. Los métodos de clonación molecular son fundamentales para muchas áreas contemporáneas de la biología y la medicina modernas. [2]
En un experimento de clonación molecular convencional, el ADN que se va a clonar se obtiene de un organismo de interés y luego se trata con enzimas en el tubo de ensayo para generar fragmentos de ADN más pequeños. Posteriormente, estos fragmentos se combinan con ADN vector para generar moléculas de ADN recombinante. Luego, el ADN recombinante se introduce en un organismo huésped (normalmente una cepa de laboratorio benigna y fácil de cultivar de la bacteria E. coli ). Esto generará una población de organismos en los que las moléculas de ADN recombinante se replican junto con el ADN del huésped. Debido a que contienen fragmentos de ADN extraños, se trata de microorganismos transgénicos o genéticamente modificados ( OGM ). [3] Este proceso aprovecha el hecho de que se puede inducir a una única célula bacteriana a absorber y replicar una única molécula de ADN recombinante. Luego, esta única célula puede expandirse exponencialmente para generar una gran cantidad de bacterias, cada una de las cuales contiene copias de la molécula recombinante original. Por tanto, tanto la población bacteriana resultante como la molécula de ADN recombinante se denominan comúnmente "clones". En sentido estricto, el ADN recombinante se refiere a moléculas de ADN, mientras que la clonación molecular se refiere a los métodos experimentales utilizados para ensamblarlas. Surgió la idea de que se podían insertar diferentes secuencias de ADN en un plásmido y que estas secuencias extrañas serían transportadas a las bacterias y digeridas como parte del plásmido. Es decir, estos plásmidos podrían servir como vectores de clonación para portar genes. [4]
Prácticamente cualquier secuencia de ADN puede clonarse y amplificarse, pero existen algunos factores que podrían limitar el éxito del proceso. Ejemplos de secuencias de ADN que son difíciles de clonar son las repeticiones invertidas, los orígenes de replicación, los centrómeros y los telómeros. También hay menores posibilidades de éxito al insertar secuencias de ADN de gran tamaño. Las inserciones de más de 10 kpb tienen un éxito muy limitado, pero los bacteriófagos como el bacteriófago λ se pueden modificar para insertar con éxito una secuencia de hasta 40 kpb. [5]
Antes de la década de 1970, la comprensión de la genética y la biología molecular se veía gravemente obstaculizada por la incapacidad de aislar y estudiar genes individuales de organismos complejos. Esto cambió drásticamente con la llegada de los métodos de clonación molecular. Los microbiólogos, tratando de comprender los mecanismos moleculares a través de los cuales las bacterias restringen el crecimiento de bacteriófagos, aislaron endonucleasas de restricción , enzimas que podían escindir moléculas de ADN sólo cuando se encontraban secuencias de ADN específicas. [6] Demostraron que las enzimas de restricción escindían moléculas de ADN de longitud cromosómica en ubicaciones específicas, y que secciones específicas de la molécula más grande podían purificarse mediante fraccionamiento por tamaño. Utilizando una segunda enzima, la ADN ligasa , los fragmentos generados por las enzimas de restricción podrían unirse en nuevas combinaciones, denominadas ADN recombinante . Al recombinar segmentos de ADN de interés con ADN vectorial, como bacteriófagos o plásmidos, que se replican naturalmente dentro de las bacterias, se podrían producir grandes cantidades de moléculas de ADN recombinante purificadas en cultivos bacterianos. Las primeras moléculas de ADN recombinante se generaron y estudiaron en 1972. [7] [8]
La clonación molecular aprovecha el hecho de que la estructura química del ADN es fundamentalmente la misma en todos los organismos vivos. Por lo tanto, si cualquier segmento de ADN de cualquier organismo se inserta en un segmento de ADN que contiene las secuencias moleculares necesarias para la replicación del ADN , y el ADN recombinante resultante se introduce en el organismo del que se obtuvieron las secuencias de replicación, entonces el ADN extraño se replicará. junto con el ADN de la célula huésped en el organismo transgénico .
La clonación molecular es similar a la PCR en que permite la replicación de la secuencia de ADN. La diferencia fundamental entre los dos métodos es que la clonación molecular implica la replicación del ADN en un microorganismo vivo, mientras que la PCR replica el ADN en una solución in vitro , libre de células vivas.
Antes de realizar experimentos de clonación reales en el laboratorio, la mayoría de los experimentos de clonación se planifican en una computadora, utilizando software especializado. Aunque la planificación detallada de la clonación se puede realizar en cualquier editor de texto, junto con utilidades en línea para, por ejemplo, el diseño de cebadores de PCR, existe software dedicado para este fin. El software para este propósito incluye, por ejemplo, ApE [1] (código abierto), DNAStrider [2] (código abierto), Serial Cloner [3] (gratis), Collagene [4] (código abierto) y SnapGene (comercial). Estos programas permiten simular reacciones de PCR , digestos de restricción , ligaciones , etc., es decir, todos los pasos que se describen a continuación.
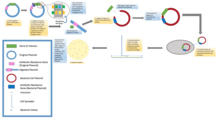
En los experimentos de clonación molecular estándar, la clonación de cualquier fragmento de ADN implica esencialmente siete pasos: (1) Elección del organismo huésped y vector de clonación, (2) Preparación del ADN vector, (3) Preparación del ADN que se va a clonar, (4) Creación de ADN recombinante, (5) Introducción de ADN recombinante en el organismo huésped, (6) Selección de organismos que contienen ADN recombinante, (7) Detección de clones con inserciones de ADN y propiedades biológicas deseadas.
En particular, la creciente capacidad y fidelidad de las plataformas de síntesis de ADN permite diseños cada vez más complejos en ingeniería molecular. Estos proyectos pueden incluir cadenas muy largas de secuencias de ADN novedosas y/o probar bibliotecas completas simultáneamente, en lugar de secuencias individuales. Estos cambios introducen una complejidad que requiere que el diseño se aleje de la representación plana basada en nucleótidos y avance hacia un mayor nivel de abstracción. Ejemplos de este tipo de herramientas son GenoCAD , Teselagen [5] (gratuito para el mundo académico) o GeneticConstructor [6] (gratuito para el académico).

Aunque se utiliza una gran cantidad de organismos huéspedes y vectores de clonación molecular, la gran mayoría de los experimentos de clonación molecular comienzan con una cepa de laboratorio de la bacteria E. coli ( Escherichia coli ) y un vector de clonación plasmídico . Los vectores de E. coli y plásmidos son de uso común porque son técnicamente sofisticados, versátiles, ampliamente disponibles y ofrecen un rápido crecimiento de organismos recombinantes con un equipo mínimo. [3] Si el ADN que se va a clonar es excepcionalmente grande (cientos de miles a millones de pares de bases), entonces a menudo se elige un cromosoma artificial bacteriano [10] o un vector cromosómico artificial de levadura .
Las aplicaciones especializadas pueden requerir sistemas de vector huésped especializados. Por ejemplo, si los experimentadores desean recolectar una proteína particular del organismo recombinante, entonces se elige un vector de expresión que contenga señales apropiadas para la transcripción y traducción en el organismo huésped deseado. Alternativamente, si se desea la replicación del ADN en diferentes especies (por ejemplo, transferencia de ADN de bacterias a plantas), entonces se puede seleccionar un vector de múltiples huéspedes (también denominado vector lanzadera ). En la práctica, sin embargo, los experimentos de clonación molecular especializados suelen comenzar con la clonación en un plásmido bacteriano, seguida de la subclonación en un vector especializado.
Cualquiera que sea la combinación de huésped y vector que se utilice, el vector casi siempre contiene cuatro segmentos de ADN que son de importancia crítica para su función y utilidad experimental: [3]

El vector de clonación se trata con una endonucleasa de restricción para escindir el ADN en el sitio donde se insertará el ADN extraño. La enzima de restricción se elige para generar una configuración en el sitio de escisión que sea compatible con los extremos del ADN extraño (ver extremo del ADN ). Normalmente, esto se hace escindiendo el ADN del vector y el ADN extraño con la misma enzima de restricción o endonucleasa de restricción, por ejemplo EcoRI y esta enzima de restricción se aisló de E. coli. [11] La mayoría de los vectores modernos contienen una variedad de sitios de escisión convenientes que son únicos dentro de la molécula del vector (de modo que el vector sólo puede escindirse en un solo sitio) y están ubicados dentro de un gen (frecuentemente beta-galactosidasa ) cuya inactivación puede ser Se utiliza para distinguir organismos recombinantes de no recombinantes en una etapa posterior del proceso. Para mejorar la proporción de organismos recombinantes y no recombinantes, el vector escindido puede tratarse con una enzima ( fosfatasa alcalina ) que desfosforila los extremos del vector. Las moléculas vectoriales con extremos desfosforilados no pueden replicarse y la replicación sólo puede restablecerse si se integra ADN extraño en el sitio de escisión. [12]
Para la clonación de ADN genómico, el ADN a clonar se extrae del organismo de interés. Se puede utilizar prácticamente cualquier fuente de tejido (incluso tejidos de animales extintos ), [13] siempre que el ADN no se degrade extensamente. Luego, el ADN se purifica mediante métodos sencillos para eliminar proteínas contaminantes (extracción con fenol), ARN (ribonucleasa) y moléculas más pequeñas (precipitación y/o cromatografía). Los métodos de reacción en cadena de la polimerasa (PCR) se utilizan a menudo para la amplificación de secuencias específicas de ADN o ARN ( RT-PCR ) antes de la clonación molecular.
El ADN para experimentos de clonación también puede obtenerse a partir de ARN mediante transcriptasa inversa ( ADN complementario o clonación de ADNc), o en forma de ADN sintético ( síntesis de genes artificiales ). La clonación de ADNc se suele utilizar para obtener clones representativos de la población de ARNm de las células de interés, mientras que el ADN sintético se utiliza para obtener cualquier secuencia precisa definida por el diseñador. Una secuencia diseñada de este tipo puede ser necesaria cuando se mueven genes a través de códigos genéticos (por ejemplo, de las mitocondrias al núcleo) [14] o simplemente para aumentar la expresión mediante la optimización de codones . [15]
Luego, el ADN purificado se trata con una enzima de restricción para generar fragmentos con extremos capaces de unirse a los del vector. Si es necesario, se pueden agregar segmentos cortos de ADN bicatenario ( enlazadores ) que contienen sitios de restricción deseados para crear estructuras terminales que sean compatibles con el vector. [3] [12]
La creación de ADN recombinante es, en muchos sentidos, el paso más sencillo del proceso de clonación molecular. El ADN preparado a partir del vector y la fuente extraña simplemente se mezclan en concentraciones apropiadas y se exponen a una enzima ( ADN ligasa ) que une covalentemente los extremos. Esta reacción de unión a menudo se denomina ligadura . La mezcla de ADN resultante que contiene extremos unidos al azar está lista para su introducción en el organismo huésped.
La ADN ligasa solo reconoce y actúa sobre los extremos de las moléculas de ADN lineales, lo que generalmente resulta en una mezcla compleja de moléculas de ADN con extremos unidos al azar. Los productos deseados (ADN del vector unido covalentemente a ADN extraño) estarán presentes, pero normalmente también están presentes otras secuencias (por ejemplo, ADN extraño unido a sí mismo, ADN del vector unido a sí mismo y combinaciones de orden superior de vector y ADN extraño). Esta mezcla compleja se separa en pasos posteriores del proceso de clonación, después de que la mezcla de ADN se introduce en las células. [3] [12]
La mezcla de ADN, previamente manipulada in vitro, se devuelve a una célula viva, denominada organismo huésped. Los métodos utilizados para introducir ADN en las células son variados y el nombre aplicado a este paso en el proceso de clonación molecular dependerá a menudo del método experimental que se elija (p. ej. , transformación , transducción , transfección , electroporación ). [3] [12]
Cuando los microorganismos son capaces de captar y replicar ADN de su entorno local, el proceso se denomina transformación , y se dice que las células que se encuentran en un estado fisiológico tal que pueden captar ADN son competentes . [16] En el cultivo de células de mamíferos, el proceso análogo de introducir ADN en las células se denomina comúnmente transfección . Tanto la transformación como la transfección generalmente requieren la preparación de las células mediante un régimen de crecimiento especial y un proceso de tratamiento químico que variará según las especies y tipos de células específicos que se utilicen.
La electroporación utiliza pulsos eléctricos de alto voltaje para translocar el ADN a través de la membrana celular (y la pared celular, si está presente). [17] Por el contrario, la transducción implica empaquetar el ADN en partículas derivadas de virus y utilizar estas partículas similares a virus para introducir el ADN encapsulado en la célula a través de un proceso que se asemeja a una infección viral. Aunque la electroporación y la transducción son métodos altamente especializados, pueden ser los métodos más eficientes para trasladar el ADN al interior de las células.
Cualquiera que sea el método utilizado, la introducción de ADN recombinante en el organismo huésped elegido suele ser un proceso de baja eficacia; es decir, sólo una pequeña fracción de las células absorberá realmente el ADN. Los científicos experimentales abordan esta cuestión mediante un paso de selección genética artificial, en el que las células que no han absorbido ADN son eliminadas selectivamente, y sólo aquellas células que pueden replicar activamente el ADN que contiene el gen marcador seleccionable codificado por el vector son capaces de sobrevivir. [3] [12]
Cuando se utilizan células bacterianas como organismos huéspedes, el marcador seleccionable suele ser un gen que confiere resistencia a un antibiótico que de otro modo mataría las células, normalmente ampicilina . Las células que albergan el plásmido sobrevivirán cuando se expongan al antibiótico, mientras que aquellas que no hayan podido absorber las secuencias del plásmido morirán. Cuando se utilizan células de mamífero (por ejemplo, células humanas o de ratón), se utiliza una estrategia similar, excepto que el gen marcador (en este caso normalmente codificado como parte del casete kanMX ) confiere resistencia al antibiótico Geneticina .
Los vectores de clonación bacteriana modernos (por ejemplo, pUC19 y derivados posteriores, incluidos los vectores pGEM) utilizan el sistema de detección azul-blanco para distinguir colonias (clones) de células transgénicas de aquellas que contienen el vector parental (es decir, ADN vectorial sin secuencia recombinante insertada). En estos vectores, el ADN extraño se inserta en una secuencia que codifica una parte esencial de la beta-galactosidasa , enzima cuya actividad resulta en la formación de una colonia de color azul en el medio de cultivo utilizado para este trabajo. La inserción del ADN extraño en la secuencia codificante de la beta-galactosidasa desactiva la función de la enzima, de modo que las colonias que contienen ADN transformado permanecen incoloras (blancas). Por lo tanto, los experimentadores pueden identificar y realizar más estudios fácilmente sobre clones bacterianos transgénicos, ignorando aquellos que no contienen ADN recombinante.
La población total de clones individuales obtenidos en un experimento de clonación molecular a menudo se denomina biblioteca de ADN . Las bibliotecas pueden ser muy complejas (como cuando se clona ADN genómico completo de un organismo) o relativamente simples (como cuando se mueve un fragmento de ADN previamente clonado a un plásmido diferente), pero casi siempre es necesario examinar varios clones diferentes para estar seguro. que se obtenga la construcción de ADN deseada. Esto se puede lograr mediante una amplia gama de métodos experimentales, incluido el uso de hibridaciones de ácidos nucleicos , sondas de anticuerpos , reacción en cadena de la polimerasa , análisis de fragmentos de restricción y/o secuenciación de ADN . [3] [12]
La clonación molecular proporciona a los científicos una cantidad esencialmente ilimitada de cualquier segmento de ADN individual derivado de cualquier genoma. Este material se puede utilizar para una amplia gama de propósitos, incluidos los de ciencias biológicas básicas y aplicadas. Algunas de las aplicaciones más importantes se resumen aquí.
La clonación molecular ha conducido directamente al esclarecimiento de la secuencia completa de ADN de los genomas de un gran número de especies y a la exploración de la diversidad genética dentro de especies individuales, trabajo que se ha realizado principalmente determinando la secuencia de ADN de un gran número de especies aleatorias. fragmentos clonados del genoma y ensamblando las secuencias superpuestas.
A nivel de genes individuales, los clones moleculares se utilizan para generar sondas que se utilizan para examinar cómo se expresan los genes y cómo esa expresión se relaciona con otros procesos en biología, incluido el entorno metabólico, las señales extracelulares, el desarrollo, el aprendizaje, la senescencia y muerte celular. Los genes clonados también pueden proporcionar herramientas para examinar la función biológica y la importancia de genes individuales, al permitir a los investigadores inactivar los genes o realizar mutaciones más sutiles mediante mutagénesis regional o mutagénesis dirigida al sitio . Los genes clonados en vectores de expresión para la clonación funcional proporcionan un medio para detectar genes en función de la función de la proteína expresada.
La obtención del clon molecular de un gen puede conducir al desarrollo de organismos que produzcan el producto proteico de los genes clonados, denominado proteína recombinante. En la práctica, suele ser más difícil desarrollar un organismo que produzca una forma activa de la proteína recombinante en cantidades deseables que clonar el gen. Esto se debe a que las señales moleculares para la expresión genética son complejas y variables, y a que el plegamiento, la estabilidad y el transporte de proteínas pueden ser muy desafiantes.
Muchas proteínas útiles están actualmente disponibles como productos recombinantes . Estos incluyen: (1) proteínas médicamente útiles cuya administración puede corregir un gen defectuoso o mal expresado (por ejemplo, factor VIII recombinante , un factor de coagulación sanguínea deficiente en algunas formas de hemofilia , [18] e insulina recombinante , utilizada para tratar algunas formas de diabetes [19] ), (2) proteínas que pueden administrarse para ayudar en una emergencia que pone en peligro la vida (por ejemplo, activador tisular del plasminógeno , utilizado para tratar accidentes cerebrovasculares [20] ), (3) vacunas de subunidades recombinantes, en las que una proteína purificada se puede utilizar para inmunizar a los pacientes contra enfermedades infecciosas, sin exponerlos al propio agente infeccioso (por ejemplo, vacuna contra la hepatitis B [21] ), y (4) proteínas recombinantes como material estándar para pruebas de diagnóstico de laboratorio.
Una vez caracterizados y manipulados para proporcionar señales para una expresión adecuada, los genes clonados pueden insertarse en organismos, generando organismos transgénicos, también denominados organismos genéticamente modificados (OGM). Aunque la mayoría de los OGM se generan con fines de investigación biológica básica (ver, por ejemplo, ratón transgénico ), se han desarrollado varios OGM para uso comercial, que van desde animales y plantas que producen productos farmacéuticos u otros compuestos ( pharming ), cultivos resistentes a herbicidas plantas y peces tropicales fluorescentes ( GloFish ) para entretenimiento en el hogar. [1]
La terapia génica implica suministrar un gen funcional a células que carecen de esa función, con el objetivo de corregir un trastorno genético o una enfermedad adquirida. La terapia génica se puede dividir en términos generales en dos categorías. La primera es la alteración de las células germinales, es decir, los espermatozoides o los óvulos, que resulta en un cambio genético permanente para todo el organismo y las generaciones posteriores. Esta "terapia genética de línea germinal" es considerada por muchos como poco ética en seres humanos. [22] El segundo tipo de terapia génica, la "terapia génica de células somáticas", es análoga a un trasplante de órgano. En este caso, uno o más tejidos específicos se dirigen mediante tratamiento directo o mediante eliminación del tejido, adición del gen o genes terapéuticos en el laboratorio y devolución de las células tratadas al paciente. Los ensayos clínicos de terapia génica con células somáticas comenzaron a finales de la década de 1990, principalmente para el tratamiento de cánceres y trastornos sanguíneos, hepáticos y pulmonares. [23]
A pesar de mucha publicidad y promesas, la historia de la terapia génica humana se ha caracterizado por un éxito relativamente limitado. [23] El efecto de introducir un gen en las células a menudo promueve sólo un alivio parcial y/o transitorio de los síntomas de la enfermedad que se está tratando. Algunos pacientes de ensayos de terapia génica han sufrido consecuencias adversas del tratamiento en sí, incluidas muertes. En algunos casos, los efectos adversos resultan de la alteración de genes esenciales dentro del genoma del paciente mediante inactivación por inserción. En otros, los vectores virales utilizados para la terapia génica han sido contaminados con virus infecciosos. Sin embargo, la terapia génica todavía se considera un área futura prometedora de la medicina y es un área en la que existe un nivel significativo de actividad de investigación y desarrollo.