Los inhibidores de la tirosina quinasa Bcr-Abl ( TKI ) son la terapia de primera línea para la mayoría de los pacientes con leucemia mieloide crónica (LMC). Más del 90% de los casos de LMC son causados por una anomalía cromosómica que resulta en la formación de un llamado cromosoma Filadelfia . Esta anomalía fue descubierta por Peter Nowell en 1960 [1] y es una consecuencia de la fusión entre el gen de la tirosina quinasa Abelson ( Abl ) en el cromosoma 9 y el gen del grupo de puntos de ruptura ( Bcr ) en el cromosoma 22 , lo que resulta en un oncogén quimérico ( Bcr-Abl ) y una tirosina quinasa Bcr-Abl constitutivamente activa que se ha implicado en la patogénesis de la LMC. Se han desarrollado compuestos para inhibir selectivamente la tirosina quinasa.
Antes de que la Administración de Alimentos y Medicamentos de los Estados Unidos (FDA) aprobara el imatinib en 2001 , no existían fármacos que pudieran alterar la progresión natural de la LMC. Solo se utilizaban fármacos citotóxicos como el busulfán , la hidroxiurea o el interferón alfa (rIFN-α). Aunque la revista Time calificó al primer inhibidor de TK Bcr-Abl como "la bala mágica" para curar el cáncer , posteriormente se desarrolló una segunda generación de TKI Bcr-Abl para combatir la resistencia inicial que surgió. [2]
Pueden surgir nuevas formas de resistencia como: mutaciones sin sentido dentro del dominio de la quinasa Abl , sobreexpresión de Bcr-Abl, aumento de la producción de proteínas plasmáticas transmembrana o la activación constitutiva de moléculas de señalización descendentes como las quinasas de la familia Src . [ cita requerida ]
También se están investigando los inhibidores de la tirosina quinasa Bcr-Abl como posibles tratamientos modificadores de la enfermedad de Parkinson. Si bien los resultados iniciales han demostrado una eficacia modesta, son necesarios más estudios que involucren a representantes muy potentes de esta clase de fármacos. [3]
La leucemia mieloide crónica tiene un objetivo molecular bien definido y terapias relativamente selectivas dirigidas a ese objetivo, lo que no es el caso de la mayoría de los cánceres y quimioterapias actuales. [4] Bcr-Abl se consideró un objetivo muy atractivo para la intervención farmacológica ya que el gen de fusión Bcr-Abl codifica una quinasa activada constitutivamente. El descubrimiento de fármacos que se dirigieran específicamente al sitio de unión de ATP de una sola quinasa se consideró una tarea bastante desafiante ya que se conocían cientos de proteínas quinasas en el genoma humano . [5] En presencia de TKI, se bloquea la unión de ATP, se previene la fosforilación y las células que expresan Bcr-Abl tienen una desventaja de crecimiento selectivo o sufren muerte celular apoptótica . [6] [7]
Debido a la creciente resistencia e intolerancia al imatinib, se realizaron esfuerzos para desarrollar nuevos fármacos que pudieran inhibir la tirosina quinasa Bcr-Abl. Esto condujo al descubrimiento de fármacos de segunda generación. Si bien se utilizó la detección de fármacos para desarrollar el imatinib, los TKI de segunda generación se desarrollaron con un enfoque de diseño de fármacos racional debido al mayor conocimiento en biología estructural de la tirosina quinasa Bcr-Abl. [8]
El imatinib (Gleevec) se descubrió en 1992 [9] y se considera un fármaco de primera generación, ya que es el primer inhibidor de la tirosina quinasa Bcr-Abl que se utiliza en el tratamiento de la LMC.

En el desarrollo de imatinib, la estructura de la tirosina quinasa Bcr-Abl jugó un papel limitado porque era desconocida. [8] Se realizó una selección de alto rendimiento de bibliotecas químicas en Novartis para identificar una molécula de partida, que se denominó " pirimidina A". Este compuesto sirvió como compuesto principal y luego se probó y modificó para desarrollar imatinib. [10] Con un reemplazo del grupo imidazol con un grupo benzamido, la especificidad del compuesto aumentó mientras que su actividad como inhibidor de la quinasa permaneció igual. Posteriormente, la introducción de un sustituyente de metilo en posición orto al grupo pirimidinil-amino mejoró la potencia . [5]

Desde entonces, los estudios cristalográficos han revelado que el imatinib se une al dominio quinasa de Abl solo cuando el dominio adopta la conformación inactiva o "cerrada". [11] Aquí es donde el bucle de fosfato de unión a P (bucle P) rico en glicina se pliega sobre el sitio de unión de ATP y el bucle de activación adopta una conformación en la que ocluye el sitio de unión del sustrato e interrumpe el sitio de unión del fosfato de ATP para bloquear la actividad catalítica de la enzima. [12] El cambio de la tríada Asp Phe Gly (DFG) en el extremo N-terminal del bucle de activación da como resultado la exposición de un bolsillo de unión que puede ser utilizado por inhibidores. [13] Esta conformación se conoce como DFGout .
El imatinib se une al dominio Abl a través de seis interacciones de enlaces de hidrógeno . Esto estabiliza el complejo Bcr-Abl del imatinib y evita que el ATP llegue a su sitio de unión. [5] [9] [11] Los enlaces de hidrógeno involucran la piridina -N y el esqueleto -NH de Met -318, la aminopirimidina y el hidroxilo de la cadena lateral de Thr -315, la amida-NH y el carboxilato de la cadena lateral de Glu -286, el carbonilo y el esqueleto-NH de Asp -381, la metilpiperazina protonada con los átomos de carbonilo del esqueleto de Ile -360 y His -361. Además, varias interacciones de van der Waals contribuyen a la unión. [9] Un bolsillo hidrofóbico está formado por los residuos de aminoácidos Ile-293, Leu -298, Leu-354 y Val -379 alrededor del anillo de fenilo adyacente al grupo piperazinil -metilo del imatinib. [11] En el momento de su descubrimiento, en ausencia de información estructural, no se pudo encontrar una explicación clara para la impresionante selectividad del imatinib. [7]
Aunque el tratamiento de primera generación logró una tasa de respuesta extremadamente alta y una tasa de recaída baja en pacientes con LMC, algunos pacientes experimentan resistencia o intolerancia al imatinib. [2]
La resistencia a los fármacos es el principal impulso en la investigación y desarrollo continuos de los inhibidores de la tirosina quinasa Bcr-Abl. Poco después de la introducción del imatinib, los investigadores comenzaron a describir una serie de líneas celulares derivadas in vitro con resistencia al fármaco. [14] Esto fue seguido rápidamente por la descripción clínica de células resistentes al imatinib en pacientes, lo que ha dado lugar a esfuerzos para comprender mejor la biología detrás de estas observaciones. Las evaluaciones de la respuesta terapéutica del imatinib en pacientes con LMC se basan en el cumplimiento de los hitos hematológicos , citogénicos y moleculares . Los pacientes que no logran respuestas definidas en puntos temporales predefinidos se describen como principalmente resistentes a la terapia, y aquellos que pierden los hitos obtenidos previamente en la regresión de la enfermedad se denominan secundariamente resistentes. [4] Antes de sacar una conclusión, es importante considerar que los datos retrospectivos han demostrado una alta incidencia de incumplimiento del tratamiento con imatinib en pacientes con LMC y esto podría conducir a resultados clínicos no deseados. [2]
En general, la resistencia al imatinib se puede subdividir en mecanismos dependientes e independientes de Bcr-Abl. Los mecanismos dependientes de Bcr-Abl incluyen la sobreexpresión o amplificación del gen Bcr-Abl y mutaciones puntuales dentro del dominio de la cinasa Bcr-Abl que interfieren con la unión del imatinib. Los mecanismos independientes de Bcr-Abl incluyen factores que influyen en la concentración de imatinib dentro de la célula, por ejemplo, alteraciones en la entrada y salida del fármaco y la activación de vías independientes de Bcr-Abl, como los miembros de la familia de cinasas Src. [2] La resistencia al imatinib también puede producirse por otros mecanismos que no se mencionarán aquí, ya que la importancia de esos mecanismos sigue siendo una incógnita debido a la falta de datos clínicos.
Los primeros informes de resistencia al imatinib describen un desarrollo de amplificación de oncogenes , es decir, el gen que codifica para la tirosina quinasa patógena Bcr-Abl se duplica en la secuencia de ADN , lo que conduce a una mayor expresión del patógeno. [4] El aumento de la dosis de imatinib podría superar este tipo de resistencia, siempre que no se produzcan efectos adversos graves o intolerables . [2]
Las mutaciones puntuales pueden provocar sustituciones de aminoácidos dentro del dominio quinasa de la proteína Bcr-Abl e interrumpir el sitio de unión del imatinib en la tirosina quinasa, lo que provoca una pérdida de sensibilidad al fármaco. Estas mutaciones normalmente afectan la estructura de la proteína Bcr-Abl, lo que provoca la interrupción de puntos de contacto críticos entre el fármaco y la proteína Bcr-Abl o la inducción de un cambio conformacional, lo que da como resultado una proteína a la que el imatinib no puede unirse. [2]
Las frecuencias mutacionales parecen aumentar a medida que la enfermedad, LMC , progresa de la fase crónica a la fase blástica . Las mutaciones más importantes son las mutaciones del bucle P y la mutación T315I. También se han descrito mutaciones en otros sitios de la quinasa, por ejemplo en la hélice C , el dominio SH2 , el sitio de unión del sustrato, el bucle de activación y el lóbulo C-terminal . Algunas de estas mutaciones tienen importancia clínica, pero ninguna tanto como las mutaciones del bucle P y T315I. [4]
La T315I es una mutación única debido a su resistencia a todos los inhibidores de Bcr-Abl aprobados, antes del ponatinib . [15] Es causada por una única sustitución del par de bases de citosina a timina (C -> T) en la posición 944 de la secuencia del gen Abl ( codón '315' de la proteína Abl) que resulta en la sustitución del aminoácido treonina por soleucina I en esa posición, por lo tanto, 'T315I'. Esta sustitución elimina una molécula de oxígeno crítica necesaria para la unión de hidrógeno entre imatinib y la quinasa Abl, y también crea un impedimento estérico para la unión de la mayoría de los TKI. [4] Cuando se descubrió, se estimó que cada 6 de cada 9 casos de LMC en etapa avanzada con resistencia a imatinib portaban esta mutación. [16] T315I produce la mayor magnitud de resistencia de cualquier mutación tanto a imatinib como a los TKI de segunda generación. [2] Ponatinib (Iclusig) de Ariad fue aprobado en 2013 para su uso como tratamiento de segunda línea para la LMC y es el único TKI autorizado que se une con éxito a la quinasa mutada T315I.
La estructura de Bcr-Abl contiene dos bucles flexibles, el bucle P de unión a ATP y el bucle de activación. Estos bucles tienen disposiciones específicas en la conformación inactiva de Bcr-Abl que estabilizan la conformación basal. Las mutaciones en estos bucles desestabilizan la disposición de los bucles de tal manera que el dominio de la quinasa no puede asumir la conformación inactiva requerida para la unión de imatinib. Las mutaciones en la región del bucle P son las más comunes y representan el 36-48% de todas las mutaciones. Hay datos clínicos que indican que las mutaciones de Bcr-Abl en el bucle P son 70-100 veces menos sensibles al imatinib en comparación con Bcr-Abl nativo. [2]
Se han postulado mecanismos adicionales para describir la resistencia observada en varios sistemas modelo, aunque ninguno ha sido identificado claramente como una fuente única de resistencia clínica. [4]
Algunas investigaciones en líneas celulares han demostrado que la resistencia al imatinib puede deberse en parte a un aumento en la expresión de la bomba de eflujo de la glicoproteína P. Mediante el uso de agentes que inhiben la actividad de la glicoproteína P, se ha restablecido la susceptibilidad al imatinib en algunos casos. [4]
La entrada de imatinib en las células depende de un transportador de cationes orgánicos ( OCT1 ). OCT1 desempeña un papel importante en la resistencia a imatinib al inhibir su entrada y, por lo tanto, disminuir la biodisponibilidad intracelular de imatinib. [17] Los pacientes con baja expresión, actividad o polimorfismos de OCT1 tuvieron niveles intracelulares significativamente más bajos de imatinib. La respuesta de los pacientes con baja actividad de OCT1 fue significativamente dependiente de la dosis. Estos datos indican que la actividad de OCT1 es un determinante importante en la respuesta molecular a imatinib. [2]
En algunos grupos de pacientes, la resistencia puede ser causada por la activación de otras vías de señalización, en particular las quinasas de la familia Src. Las quinasas de la familia Src han estado implicadas en la señalización de Bcr-Abl y median la resistencia al imatinib al estabilizar la conformación activa de Bcr-Abl, una conformación que no se une al imatinib. Además, cada vez hay más evidencia que sugiere que las quinasas de la familia Src también están implicadas en formas de resistencia al imatinib independientes de Bcr-Abl.
Las opciones de tratamiento para pacientes con LMC resistente o intolerante al imatinib pueden incluir estrategias como aumentar la dosis de imatinib o el uso de medicamentos de segunda generación. Se ha demostrado que la escalada de dosis de imatinib supera algunos casos de resistencia primaria al imatinib, como la duplicación de Bcr-Abl, pero la respuesta suele ser de acción corta. [2] En el caso de resistencia o intolerancia, podría ser útil realizar pruebas de mutaciones de Bcr-Abl para orientar la elección del tratamiento de segunda línea, ya que las opciones variables tienen un perfil de función diferente frente a los diferentes mecanismos de resistencia. [15] Los medicamentos de segunda generación ofrecen una potencia mejorada y una mayor probabilidad de éxito en pacientes resistentes. [2] También existe un creciente interés en probar la hipótesis de que la administración de múltiples inhibidores de la cinasa Abl en pacientes en fase temprana podría utilizarse para retrasar o prevenir la aparición de clones resistentes a los medicamentos . La combinación de dos agentes dirigidos a diferentes vías implicadas en la LMC puede mejorar significativamente las tasas de respuesta y potencialmente aumentar la supervivencia. [18] [19]
Los fármacos de segunda generación tienen como objetivo reducir la resistencia y la intolerancia al imatinib. Los fármacos de segunda generación que se comercializan actualmente son el nilotinib, el dasatinib, el bosutinib y el ponatinib (tercera generación).
El nilotinib es un derivado de fenilaminopirimidina que está estructuralmente relacionado con el imatinib. [12] Se desarrolló basándose en la estructura del complejo Abl-imatinib para abordar la necesidad asociada con la intolerancia y la resistencia al imatinib. [13] [18] [20] Se realizaron pequeños cambios en la molécula de imatinib para hacerla más potente y selectiva como inhibidor de Bcr-Abl y estos cambios dieron como resultado el descubrimiento del nilotinib. El nilotinib es un inhibidor selectivo de la quinasa Bcr-Abl. [13] [18]
El nilotinib es 10-30 veces más potente que el imatinib en la inhibición de la actividad de la tirosina quinasa Bcr-Abl y la proliferación de células que expresan Bcr-Abl. [12] [18] [20] [21] El fármaco inhibe eficazmente la autofosforilación de Bcr-Abl en Tyr -177 que está involucrada en la patogénesis de la LMC. [21] Se ha informado de una actividad sinérgica de imatinib y nilotinib después de la coadministración. Esto podría ser el resultado del hecho de que los fármacos son absorbidos por las células por diferentes mecanismos: la entrada de imatinib depende de OCT1, pero el nilotinib no. El nilotinib tampoco es un sustrato para la bomba de glicoproteína P del transportador de eflujo, a diferencia del imatinib. [18] [21] Aunque las estructuras moleculares bidimensionales de estos dos fármacos pueden parecer similares, son diferentes en términos de estructura espacial y propiedades moleculares. [13]

El nilotinib se une a la conformación inactiva del dominio de la quinasa Abl, principalmente a través de interacciones lipofílicas y, por lo tanto, bloquea su actividad catalítica. [12] [18] El nilotinib se une al dominio de la quinasa al realizar cuatro interacciones de enlace de hidrógeno que involucran al piridil -N y el NH de la cadena principal de Met-318, el anilino -NH y el OH de la cadena lateral de Thr-315, el amido-NH y el carboxilato de la cadena lateral de Glu-286 y el amido carbonilo con el NH de la cadena principal de Asp-381. [13] [21] El segmento [4-(3-piridinil)-2-pirimidinil] anilino del nilotinib tiene interacciones de unión cercanas con los residuos Met-318, Phe-317 y Thr-315 de una región dentro del sitio de unión de ATP. La mitad restante del compuesto se extiende más allá del residuo guardián de Thr-315 para unirse dentro de un bolsillo adicional. Los grupos 3-metilimidazol y trifluorometil del nilotinib generan interacciones importantes con el dominio de la cinasa Abl. Estos grupos también hacen que la forma del nilotinib sea muy diferente a la del imatinib. El nilotinib también se une a la cinasa a través de una gran cantidad de interacciones débiles de van der Waals. [13]
El nilotinib ha demostrado tener efecto contra la mayoría de las mutaciones (32/33) asociadas con la resistencia al imatinib, pero el mutante T315I sigue siendo resistente al nilotinib. [12] [13] [18] Su ineficacia contra el mutante T315I parece ser consecuencia de la pérdida de una interacción de enlace de H entre la treonina-O y la anilina-NH en el nilotinib y un choque estérico entre el grupo isoleucina-metilo y el grupo 2-metilfenilfenilo del nilotinib. [12] Por otro lado, la resistencia al nilotinib está asociada con un espectro limitado de mutaciones de la quinasa Bcr-Abl que afectan principalmente al bucle P y al T315I. Sin embargo, todas las mutaciones excepto T315I se suprimieron eficazmente al aumentar la concentración de nilotinib. [18] Aunque el nilotinib es más potente que el imatinib, es posible que su modo específico de unión a Abl pueda hacer que otros sitios sean vulnerables a nuevos tipos de resistencia a fármacos. [20]

El dasatinib es una tiazolilaminopirimidina desarrollada como sal de clorhidrato . Fue descubierto con un programa dirigido a fármacos inmunosupresores y es 325 veces más potente contra las células que expresan Bcr-Abl de tipo salvaje que el imatinib. [12] [20] El dasatinib es un inhibidor multidireccional de las quinasas de la familia Bcr-Abl y Src. [12] [20] También tiene actividad inhibidora contra otras quinasas posteriores. [20] [22]

El dasatinib se une a la Abl con requisitos conformacionales menos estrictos que el imatinib, por lo que muestra una mayor potencia pero una selectividad reducida en comparación con el imatinib. [20] El dasatinib se une tanto a la conformación activa como a la inactiva de la quinasa Abl, al contrario de la unión de la mayoría de los demás TKI solo a la forma activa. [24] Se han identificado compuestos que se dirigen a la conformación activa, pero el sitio de unión en todos los cientos de quinasas proteínicas humanas es muy similar. Por lo tanto, existe un margen considerablemente mayor para las diferencias entre las conformaciones inactivas, por lo que los esfuerzos para descubrir inhibidores de quinasas altamente selectivos se están dirigiendo hacia moléculas que se unan a la conformación inactiva. [12]
El dasatinib tiene algunos elementos estructurales en común con el nilotinib, en particular la yuxtaposición de los grupos aminopirimidina y carboxamida . El segmento aminotiazol del dasatinib establece una interacción de enlace de hidrógeno bidentado con el CO y el NH de la cadena principal de Met-318 y el NH de la amida establece un enlace de hidrógeno con el oxígeno de la cadena lateral de Thr-315. [12]
Dado que el dasatinib es un inhibidor de las quinasas de la familia Src, puede superar la resistencia debida a la activación de las quinasas de la familia Src. Como no se une a Bcr-Abl con los mismos requisitos conformacionales estrictos que el imatinib, puede inhibir todos los mutantes del dominio de la quinasa Bcr-Abl, excepto T315I. El dasatinib tampoco es un sustrato de las bombas de eflujo de la P-glicoproteína de múltiples fármacos como el imatinib. Debido a esto, el dasatinib puede ser activo en algunos pacientes después del fracaso tanto con imatinib como con nilotinib. [20] Aunque el dasatinib es mucho más potente que el imatinib, es posible, como con el nilotinib, que su modo específico de unión a Abl pueda conducir a nuevos sitios vulnerables que podrían conferir nuevos tipos de resistencia a los fármacos. Se han encontrado mutaciones en Phe317, por lo que es un sitio vulnerable potencial para este fármaco. [20]

La estructura de bosutinib se basa en un andamio de quinolina y está estructuralmente relacionada con la plantilla de quinazolina de AstraZeneca . [12] La detección de levadura dependiente de la quinasa Src condujo a la caracterización de un carbonitrilo de 4-anilino-3-quinolina como inhibidor de Src. La combinación de las características de este hit y un compuesto relacionado, y la unión de grupos solubilizantes , condujo al descubrimiento de bosutinib. Se sugirió que era un inhibidor de la quinasa Abl y cuando se probó como tal resultó ser ligeramente más potente contra Abl que Src ( CI50 1,4 nM frente a 3,5 nM ). [25] La actividad de bosutinib se describió por primera vez en 2001 y se reveló como un inhibidor de la quinasa Abl en 2003. Al principio se creyó que bosutinib era un inhibidor selectivo de la quinasa Src, pero ahora se sabe que su perfil de inhibición de la quinasa es mucho menos restringido de lo que se pensaba originalmente. Bosutinib inhibe Src, Abl y una amplia gama de quinasas de tirosina y serina-treonina. [25]
El bosutinib inhibió las células que expresaban una variedad de mutaciones, algunas de las cuales condujeron a la resistencia al imatinib, pero la mutación T315 fue completamente resistente al bosutinib. [12] [25] A diferencia del imatinib, el nilotinib y el dasatinib, el bosutinib no es un sustrato eficiente para los transportadores de resistencia a múltiples fármacos (MDR) que promueven el eflujo de moléculas extrañas de las células. El bosutinib incluso inhibe estas proteínas transportadoras en concentraciones más altas. [25]
ARIAD Pharmaceuticals , Inc. anunció el 10 de septiembre de 2010 que ponatinib , un TKI Bcr-Abl activo por vía oral eficaz contra la mutación T315I, había sido aprobado para un ensayo clínico de fase II. [26]
El camino hacia el descubrimiento puede vincularse con AP23464, uno de los primeros inhibidores duales de Src/Abl competitivos con ATP de Ariad. AP23464 se identificó mediante el diseño de fármacos basados en la estructura y bibliotecas sintéticas enfocadas en análogos de purina trisustituidos . La sustancia inhibe de forma potente, a escala nanomolar, las quinasas Src y Bcr-Abl, incluidas muchas mutaciones comunes de Bcr-Abl resistentes al imatinib. Sin embargo, AP23464 no inhibe la mutación T315I, mientras que AP24534 (ponatinib) sí lo hace. [27]

Ariad utilizó el fármaco líder altamente potente, AP23464, para investigar más a fondo las posibilidades inhibidoras de las plantillas con núcleo de purina para inhibidores duales de Src/Abl. Primero, buscando sustancias efectivas en la conformación inactiva de Abl, la cadena lateral unida al nitrógeno en el núcleo de purina se reemplazó con una estructura de diarilamida , que se sabía que tenía una alta afinidad con la conformación inactiva al formar enlaces de hidrógeno cruciales y llenar bolsillos hidrofóbicos en la quinasa. Además, se determinó que el grupo ciclopentilo en el núcleo de purina chocaba con un bucle P rico en glicina en esa confirmación y, por lo tanto, se eliminó de la molécula. Luego, con pruebas in vitro sobre la actividad inhibidora y ensayos de absorción oral in vivo, se descubrió que un grupo ciclopropilo más lipofílico, unido a amida, en C6 en el núcleo de purina mostraba una farmacocinética y una eficacia satisfactorias. Finalmente, las modificaciones en la cadena lateral de diarilamida mediante la adición de apéndices de imidazol se inspiraron en la estructura de nilotinib recién lanzada. Estas modificaciones dieron como resultado lo que se denominó AP24163. Durante este ciclo de desarrollo, Ariad probó varias sustancias contra células transfectadas con la quinasa Bcr-Abl mutada T315I y, sorprendentemente, descubrió que AP24163 demostraba una acción inhibidora razonable además de una potente inhibición de la Bcr-Abl nativa. [28]
Tras este gran avance, Ariad comenzó a investigar más para aumentar la eficacia del compuesto AP24163 contra la mutación T315I. El acoplamiento de la molécula al sitio de unión de ATP de la quinasa Bcr-Abl mutada T315I reveló que el choque estérico esperado con la isoleucina no estaba presente debido a un enlace de vinilo estéricamente menos exigente entre el núcleo de purina y la cadena lateral de diarilamida en comparación con otros TKI. El primer paso fue tratar de encontrar una estructura estéricamente menos exigente. Primero se probó un enlace de acetileno , que resultó en una mayor potencia pero una farmacocinética desfavorable. Más tarde, se seleccionó un enlace 2-butino más estable . Para lograr este enlace, se utilizó un núcleo de imidazol[1,2-a]piridina como material de partida para una reacción de Sonogashira ; pero la farmacocinética seguía siendo deficiente. Al desarrollar AP24163, agregar una cadena lateral de ciclopropano en C8 en el núcleo de purina resultó en una farmacocinética favorable. Se probaron varias cadenas laterales diferentes, pero los mejores resultados se obtuvieron sin ninguna cadena lateral, lo que dio como resultado una sustancia con farmacocinética satisfactoria, pero ahora también con potencia reducida contra T315I. El primer paso para aumentar la potencia nuevamente fue observar otros TKI. Imatinib tiene un grupo terminal de metil piperazina que se ha demostrado que forma un enlace de hidrógeno con el átomo de oxígeno carbonílico del residuo Ile-360 en el bucle de activación de la quinasa Abl. El anillo de piperazina también es un grupo solubilizante común que podría mejorar aún más las propiedades farmacocinéticas de la molécula. Esas especulaciones se confirmaron con un aumento de dos veces en la acción inhibidora contra la quinasa mutada Bcr-Abl T315I y el lado positivo fue que la unión a proteínas plasmáticas de la sustancia (llamada '19a') pareció haber disminuido, lo que permitió dosis más pequeñas con la misma potencia. Si bien '19a' exhibió una buena farmacocinética oral tanto en ratones como en ratas, también mantuvo un alto coeficiente de partición de 6,69. Entonces, en un intento de reducir aún más la lipofilicidad de la molécula, se realizó la sustitución de un solo átomo de carbono en el núcleo imidazo[1,2-a]piridina; lo que dio como resultado lo que ahora se conoce como el compuesto ponatinib . [29]
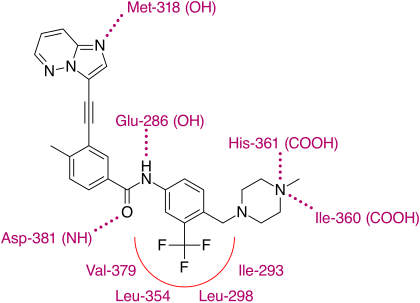
El análisis cristalográfico de rayos X de ponatinib y la quinasa mutada T315I Bcr-Abl muestra que el núcleo de imidazo[1,2b] piridazina se encuentra en el bolsillo de adenina de la enzima. El grupo metilfenilo ocupa un bolsillo hidrofóbico detrás de I315, el enlace etinilo forma interacciones de van der Waals favorables con el aminoácido y el grupo trifluorometilo se une a un bolsillo inducido por la quinasa de conformación inactiva. Además, en la conformación de la quinasa en la que se encuentra ponatinib, se observan interacciones de van der Waals favorables adicionales entre el fármaco y Tyr-253 y Phe-382. Se generan cinco enlaces de hidrógeno, con la cadena principal de Met-318 en la región bisagra, con la cadena principal de Asp-381, con la cadena lateral de Glu-286 y la metilpiperazina protonada con los átomos de carbonilo de la cadena principal de Ile-360 y His-361. [30]
Con esta estructura, se ha demostrado que el ponatinib tiene un perfil de especificidad de quinasa relativamente amplio que probablemente se pueda vincular a la linealidad de la sección de enlace de la molécula. Con esta estructura lineal, el fármaco parece evitar los choques estéricos con los residuos hidrófobos guardianes de las cinasas transcripcionales. A pesar de esto, o incluso debido a esto, el ponatinib es un fármaco potente y se dirige no solo a la mayoría de las mutaciones conocidas en las cinasas transcripcionales Bcr-Abl sino, lo más importante de todo, a la T315I. Esta mutación está surgiendo como una vía común de fracaso de los tratamientos de primera y segunda línea. A diferencia de otros inhibidores dirigidos a la T315I en desarrollo, el ponatinib no se dirige a las cinasas Aurora, lo que lo distingue claramente de ellas y enfatiza la importancia de su descubrimiento. [30]
Con la aparición de resistencia al tratamiento con imatinib tras su lanzamiento, se buscó un tratamiento alternativo. Bafetinib fue el resultado de un intento de crear un fármaco más potente que el imatinib, con eficacia contra varias mutaciones puntuales en la quinasa Bcr-Abl, con menos efectos adversos y con espectros de quinasas más estrechos, concretamente solo Lyn y Bcr-Abl. [31]
En la búsqueda de una sustancia que se ajustara a los criterios mencionados, se examinó la estructura cristalina del imatinib unido a Abl. Esto reveló una cavidad hidrofóbica alrededor del anillo de fenilo adyacente al grupo piperazinilmetilo del imatinib. Los intentos de utilizar esta cavidad para aumentar la eficacia llevaron a la adición de varios grupos hidrofóbicos, incluidos sustituyentes simples de flúor , bromo y cloro . Finalmente, se descubrió que un grupo trifluorometilo en la posición 3 daba los mejores resultados, con una mejora de aproximadamente 36 veces sobre el imatinib. Ahora era necesario contrarrestar la adición de un grupo hidrofóbico para mantener la solubilidad de la sustancia. Un examen más detallado de la estructura cristalina del complejo imatinib-quinasa reveló que Tyr-236 estaba muy cerca del anillo de piridina del imatinib, lo que sugiere que había poco o ningún espacio para un grupo más grande allí. Con eso en mente, se sustituyó la piridina por un anillo de pirimidina más hidrofílico , que se descubrió que aumentaba la solubilidad mientras que la eficacia era la misma o incluso ligeramente mayor. Finalmente, para mejorar la unión de hidrógeno del anillo de piperazina del imatinib con Ile-360 e His-361, se introdujeron derivados de pirrolidina y azetidina. La sustancia más prometedora de estas modificaciones finales fue etiquetada como NS-187. [11]
Debido a las similitudes estructurales de imatinib y bafetinib, su unión a Bcr-Abl también es bastante similar. La única diferencia notable proviene de la interacción hidrofóbica entre el grupo trifluorometilo y el bolsillo hidrofóbico creado por Ile-293, Leu-298, Leu-354 y Val-379. Este grupo también puede estar vinculado a la especificidad de bafetinib para Lyn, ya que el sitio de unión allí es casi idéntico al de Bcr-Abl. [32]
El bafetinib tiene su lugar en la terapia con TKI, ya que es eficaz tanto contra la mayoría de las mutaciones resistentes al imatinib (sin incluir T315I) como contra algunas mutaciones resistentes al dasatinib. El bafetinib también tiene más afinidad por Bcr-Abl que el nilotinib (pero menos que el dasatinib), pero solo se dirige a las quinasas Lck y Lyn de la familia Bcr-Abl y Src; con una especificidad inigualable que sugiere la probabilidad de menos efectos adversos. [33]
CytRx ha incluido bafetinib en ensayos clínicos de fase II como tratamiento para la leucemia desde mayo de 2010. [34]
Se ha despertado cierto interés en los derivados de tiazol y tiadiazol y su capacidad para inhibir las TK Bcr-Abl.

Un grupo de investigación italiano descubrió mediante un análisis digital que los derivados de tiadiazol disponibles comercialmente mostraban una acción inhibidora moderada sobre las quinasas Abl y Src. [35] Utilizando un núcleo de 1,3,4 tiadiazol y probando diferentes grupos o moléculas en los anillos de benceno, se produjeron varias sustancias diferentes con propiedades inhibidoras. La flexibilidad del núcleo permitió que varias conformaciones de las sustancias se unieran al sitio de ATP de la quinasa Abl, aunque todas se unieron a la forma activa de la quinasa. [35] Un estudio posterior de la unión mostró que la posición del azufre que se une a la estructura del tolueno jugó un papel importante con respecto a la unión de Abl y también que solo uno de los tiadiazoles del nitrógeno formaba un enlace de hidrógeno. Además, el análisis por computadora de la estructura mostró que el anillo de benceno-cetona conectado con amida podía sustituirse por un anillo de tiofeno más favorable . [36] Aunque debe tenerse en cuenta que este análisis se realizó comparando la estructura cristalina de Abl y dasatinib, que es la conformación inactiva de Abl, el conocimiento obtenido del análisis de acoplamiento y estructura condujo a la identificación de un compuesto, denominado sustancia 14, con una alta afinidad por Abl.
La unión de la sustancia 14 es parcialmente similar a la del dasatinib, el segmento aminotiazol de la sustancia 14 hace una interacción de enlace de H bidentado con la cadena principal CO y NH de Met-318 mientras que el metoxi -benceno cae muy bien en un bolsillo hidrofóbico creado por Val 256, Ala 253, Lys 271 y Ala 380. [36] Si bien las propiedades de unión similares a las del dasatinib sugieren que la posibilidad de producir TKI Bcr-Abl a partir de núcleos de tiazol es real, la pregunta sigue abierta sobre si esta investigación solo conducirá a un análogo de dasatinib o a una nueva forma de inhibir los TK.
Rebastinib (DCC-2036) También es un inhibidor de TIE-2 y VEGFR-2. [37] Ha tenido un ensayo clínico de fase 1 para leucemias (LMC Ph+ con mutación T315I). [38] Se encuentra en un ensayo clínico de fase 1 de terapia combinada para cáncer de mama metastásico. [39]
Asciminib (ABL001) es un inhibidor de la quinasa Abelson que se dirige al bolsillo de miristoilo para inhibir alostéricamente la enzima. [40] En agosto de 2020, había completado un estudio de fase III en LMC (ASCEMBL) que mostraba una eficacia superior al bosutinib.
El imatinib sigue siendo un inhibidor de la tirosina quinasa de primera línea estándar. El nilotinib y el dasatinib también fueron aprobados por la FDA como medicamentos de primera línea, en junio y octubre de 2010, respectivamente. Cuatro de estos medicamentos, nilotinib, dasatinib, bosutinib y ponatinib, están aprobados para el tratamiento de la leucemia mieloide crónica resistente o intolerante al imatinib. Los datos de primera línea para estos compuestos son alentadores y sugieren que algunos o todos ellos pueden reemplazar al imatinib como inhibidor de la tirosina quinasa de primera línea estándar en el futuro. [41]