
La fase G 0 describe un estado celular fuera del ciclo celular replicativo . Clásicamente [ ¿cuándo? ] , se pensaba que las células entraban en G 0 principalmente debido a factores ambientales, como la privación de nutrientes, que limitaban los recursos necesarios para la proliferación. Por lo tanto, se pensaba que era una fase de reposo . Ahora se sabe que G 0 adopta diferentes formas y ocurre por múltiples razones. Por ejemplo, la mayoría de las células neuronales adultas , entre las células metabólicamente más activas del cuerpo, están completamente diferenciadas y residen en una fase G 0 terminal . Las neuronas residen en este estado, no debido a un suministro estocástico o limitado de nutrientes, sino como parte de su programa de desarrollo.
G 0 se sugirió por primera vez como un estado celular basado en estudios tempranos del ciclo celular. Cuando los primeros estudios definieron las cuatro fases del ciclo celular utilizando técnicas de marcaje radiactivo, se descubrió que no todas las células de una población proliferan a tasas similares. [1] La "fracción de crecimiento" de una población, o la fracción de la población que estaba creciendo, estaba proliferando activamente, pero otras células existían en un estado no proliferativo. Algunas de estas células no proliferantes podían responder a estímulos extrínsecos y proliferar reingresando al ciclo celular. [2] Los primeros puntos de vista opuestos consideraban que las células no proliferantes simplemente estaban en una fase G 1 extendida o en una fase del ciclo celular distinta de G 1 , denominada G 0. [3] Las investigaciones posteriores señalaron un punto de restricción (punto R) en G 1 donde las células pueden ingresar a G 0 antes del punto R, pero están comprometidas con la mitosis después del punto R. [4] Estos primeros estudios proporcionaron evidencia de la existencia de un estado G 0 al que se restringe el acceso. Estas células que no se dividen más salen de la fase G 1 para entrar en una etapa inactiva llamada etapa quiescente.

Existen tres estados G 0 y pueden clasificarse como reversibles (quiescentes) o irreversibles ( senescentes y diferenciados ). Se puede ingresar a cada uno de estos tres estados desde la fase G 1 antes de que la célula se comprometa a la siguiente ronda del ciclo celular. La quiescencia se refiere a un estado G 0 reversible donde las subpoblaciones de células residen en un estado "quiescente" antes de ingresar al ciclo celular después de la activación en respuesta a señales extrínsecas. Las células quiescentes a menudo se identifican por un bajo contenido de ARN , falta de marcadores de proliferación celular y mayor retención de etiquetas que indica una baja renovación celular. [5] [6] La senescencia se distingue de la quiescencia porque la senescencia es un estado irreversible en el que las células ingresan en respuesta al daño o degradación del ADN que haría que la progenie de una célula no sea viable. Dicho daño al ADN puede ocurrir por el acortamiento de los telómeros a lo largo de muchas divisiones celulares, así como por la exposición a especies reactivas de oxígeno (ROS), la activación de oncogenes y la fusión célula-célula. Si bien las células senescentes ya no pueden replicarse, siguen siendo capaces de realizar muchas funciones celulares normales. [7] [8] [9] [10] La senescencia es a menudo una alternativa bioquímica a la autodestrucción de una célula dañada por apoptosis . A diferencia de la senescencia celular, la quiescencia no es un evento reactivo sino parte de la programación central de varios tipos de células diferentes. Finalmente, las células diferenciadas son células madre que han progresado a través de un programa de diferenciación para alcanzar un estado maduro (terminalmente diferenciado). Las células diferenciadas continúan permaneciendo en G 0 y realizan sus funciones principales indefinidamente.
Los transcriptomas de varios tipos de células madre quiescentes, como las hematopoyéticas , musculares y de folículos pilosos, se han caracterizado a través de técnicas de alto rendimiento , como microarrays y secuenciación de ARN . Aunque existen variaciones en sus transcriptomas individuales, la mayoría de las células madre tisulares quiescentes comparten un patrón común de expresión génica que implica la regulación negativa de los genes de progresión del ciclo celular, como ciclina A2 , ciclina B1 , ciclina E2 y survivina , y la regulación positiva de los genes involucrados en la regulación de la transcripción y el destino de las células madre, como FOXO3 y EZH1 . La regulación negativa del citocromo C mitocondrial también refleja el bajo estado metabólico de las células madre quiescentes. [11]
Muchas células madre quiescentes, en particular las células madre adultas , también comparten patrones epigenéticos similares. Por ejemplo, H3K4me3 y H3K27me3 son dos patrones principales de metilación de histonas que forman un dominio bivalente y se encuentran cerca de los sitios de inicio de la transcripción. Se ha descubierto que estos marcadores epigenéticos regulan las decisiones de linaje en las células madre embrionarias, así como también controlan la quiescencia en las células madre de los folículos pilosos y musculares a través de la modificación de la cromatina . [11]
Los genes supresores de tumores funcionales , en particular el gen p53 y el gen Rb , son necesarios para mantener la quiescencia de las células madre y evitar el agotamiento del grupo de células progenitoras a través de divisiones excesivas. Por ejemplo, se ha demostrado que la eliminación de los tres componentes de la familia de proteínas Rb detiene la quiescencia en las células madre hematopoyéticas. Se ha demostrado que la falta de p53 impide la diferenciación de estas células madre debido a la incapacidad de las células para salir del ciclo celular a la fase G 0. Además de p53 y Rb, los inhibidores de la cinasa dependiente de ciclina (CKI), como p21 , p27 y p57 , también son importantes para mantener la quiescencia. En las células madre hematopoyéticas de ratón, la eliminación de p57 y p27 conduce a la salida de G 0 a través de la importación nuclear de ciclina D1 y la posterior fosforilación de Rb. Finalmente, se ha demostrado que la vía de señalización Notch desempeña un papel importante en el mantenimiento de la quiescencia. [11]
Se ha demostrado que la regulación postranscripcional de la expresión génica a través de la síntesis de miRNA desempeña un papel igualmente importante en el mantenimiento de la quiescencia de las células madre. Las cadenas de miRNA se unen a la región no traducida 3' ( 3' UTR ) de los ARNm diana , impidiendo su traducción en proteínas funcionales. La longitud de la UTR 3' de un gen determina su capacidad para unirse a las cadenas de miRNA, lo que permite la regulación de la quiescencia. Algunos ejemplos de miRNA en células madre incluyen miR-126, que controla la vía PI3K/AKT/mTOR en células madre hematopoyéticas, miR-489, que suprime el oncogén DEK en células madre musculares, y miR-31, que regula Myf5 en células madre musculares. El secuestro de miRNA del ARNm dentro de los complejos de ribonucleoproteína permite a las células quiescentes almacenar el ARNm necesario para una entrada rápida en la fase G1 . [11]
Las células madre que han permanecido inactivas durante mucho tiempo suelen enfrentarse a diversos factores estresantes ambientales, como el estrés oxidativo . Sin embargo, varios mecanismos permiten que estas células respondan a dichos factores estresantes. Por ejemplo, los factores de transcripción FOXO responden a la presencia de especies reactivas de oxígeno (ROS), mientras que HIF1A y LKB1 responden a condiciones hipóxicas . En las células madre hematopoyéticas, la autofagia se induce para responder al estrés metabólico. [11]
Las células madre son células con la capacidad única de producir células hija diferenciadas y de preservar su identidad de células madre a través de la autorrenovación. [12] En los mamíferos, la mayoría de los tejidos adultos contienen células madre específicas de tejido que residen en el tejido y proliferan para mantener la homeostasis durante la vida del organismo. Estas células pueden experimentar una inmensa proliferación en respuesta al daño tisular antes de diferenciarse y participar en la regeneración. Algunas células madre tisulares existen en un estado reversible y quiescente indefinidamente hasta que son activadas por estímulos externos. Existen muchos tipos diferentes de células madre tisulares, incluidas las células madre musculares (MuSC), las células madre neurales (NSC), las células madre intestinales (ISC) y muchas otras.
Recientemente se ha sugerido que la quiescencia de las células madre se compone de dos fases funcionales distintas, G 0 y una fase de "alerta" denominada G Alert . [13] Se cree que las células madre realizan una transición activa y reversible entre estas fases para responder a los estímulos de las lesiones y parecen obtener una función regenerativa tisular mejorada en G Alert . Por lo tanto, se ha propuesto la transición a G Alert como una respuesta adaptativa que permite a las células madre responder rápidamente a las lesiones o al estrés preparándolas para la entrada en el ciclo celular. En las células madre musculares, se ha identificado que la actividad de mTORC1 controla la transición de G 0 a G Alert junto con la señalización a través del receptor de HGF cMet . [13]
Si bien un estado de quiescencia reversible es quizás lo más importante para que las células madre de los tejidos respondan rápidamente a los estímulos y mantengan una homeostasis y regeneración adecuadas, se pueden encontrar fases G0 reversibles en células no madre, como los hepatocitos maduros. [14] Los hepatocitos suelen estar en estado de quiescencia en hígados normales, pero experimentan una replicación limitada (menos de 2 divisiones celulares) durante la regeneración hepática después de una hepatectomía parcial. Sin embargo, en ciertos casos, los hepatocitos pueden experimentar una proliferación inmensa (más de 70 divisiones celulares), lo que indica que su capacidad de proliferación no se ve obstaculizada por existir en un estado de quiescencia reversible. [14]
Las células senescentes, que suelen asociarse con el envejecimiento y las enfermedades relacionadas con la edad in vivo, se pueden encontrar en muchos tejidos renovables, incluidos el estroma , la vasculatura , el sistema hematopoyético y muchos órganos epiteliales . Como resultado de la acumulación a lo largo de muchas divisiones celulares, la senescencia se observa a menudo en fenotipos degenerativos asociados con la edad. Se ha descubierto que los fibroblastos senescentes en modelos de función de células epiteliales mamarias alteran la producción de proteínas de la leche debido a la secreción de metaloproteinasas de matriz . [15] De manera similar, las células musculares lisas de la arteria pulmonar senescentes provocaron que las células musculares lisas cercanas proliferaran y migraran, lo que tal vez contribuyera a la hipertrofia de las arterias pulmonares y, finalmente, a la hipertensión pulmonar. [16]
Durante la miogénesis esquelética , las células progenitoras cíclicas conocidas como mioblastos se diferencian y se fusionan para formar células musculares no cíclicas llamadas miocitos que permanecen en una fase G 0 terminal. [17] Como resultado, las fibras que forman el músculo esquelético (miofibras) son células con múltiples núcleos, denominados mionúcleos, ya que cada mionúcleo se originó a partir de un solo mioblasto. Las células del músculo esquelético continúan proporcionando fuerza contráctil indefinidamente a través de contracciones simultáneas de estructuras celulares llamadas sarcómeros . Es importante destacar que estas células se mantienen en una fase G 0 terminal , ya que la alteración de la estructura de la fibra muscular después de la formación de la miofibra impediría la transmisión adecuada de la fuerza a lo largo del músculo. El crecimiento muscular puede ser estimulado por el crecimiento o una lesión e implica el reclutamiento de células madre musculares, también conocidas como células satélite, fuera de un estado inactivo reversible. Estas células madre se diferencian y se fusionan para generar nuevas fibras musculares tanto en paralelo como en serie para aumentar la capacidad de generación de fuerza.
El músculo cardíaco también se forma a través de la miogénesis, pero en lugar de reclutar células madre para fusionarse y formar nuevas células, las células del músculo cardíaco, conocidas como cardiomiocitos , simplemente aumentan de tamaño a medida que el corazón se hace más grande. De manera similar al músculo esquelético, si los cardiomiocitos tuvieran que seguir dividiéndose para agregar tejido muscular, las estructuras contráctiles necesarias para la función cardíaca se verían alteradas.
De los cuatro tipos principales de células óseas, los osteocitos son los más comunes y también existen en una fase terminal G 0. Los osteocitos surgen de osteoblastos que están atrapados dentro de una matriz autosecretada. Si bien los osteocitos también tienen una actividad sintética reducida, aún cumplen funciones óseas además de generar estructura. Los osteocitos funcionan a través de varios mecanismos mecanosensoriales para ayudar en la renovación rutinaria de la matriz ósea.
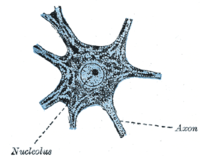
Aparte de unos pocos nichos neurogénicos en el cerebro, la mayoría de las neuronas están completamente diferenciadas y residen en una fase terminal G 0. Estas neuronas completamente diferenciadas forman sinapsis donde las señales eléctricas son transmitidas por los axones a las dendritas de las neuronas cercanas. En este estado G 0 , las neuronas continúan funcionando hasta la senescencia o la apoptosis. Numerosos estudios han informado de la acumulación de daño del ADN con la edad, en particular daño oxidativo , en el cerebro de los mamíferos . [18]
Se descubrió por primera vez que Rim15 desempeña un papel fundamental en la iniciación de la meiosis en células de levadura diploide . En condiciones de baja glucosa y nitrógeno, que son nutrientes clave para la supervivencia de la levadura, las células de levadura diploide inician la meiosis a través de la activación de genes específicos de la meiosis temprana (EMG). La expresión de EMG está regulada por Ume6. Ume6 recluta a las histonas desacetilasas , Rpd3 y Sin3, para reprimir la expresión de EMG cuando los niveles de glucosa y nitrógeno son altos, y recluta al factor de transcripción EMG Ime1 cuando los niveles de glucosa y nitrógeno son bajos. Rim15, llamado así por su papel en la regulación de un EMG llamado IME2, desplaza a Rpd3 y Sin3, lo que permite a Ume6 llevar Ime1 a los promotores de EMG para la iniciación de la meiosis. [19]
Además de desempeñar un papel en la iniciación de la meiosis, también se ha demostrado que Rim15 es un efector crítico para la entrada de células de levadura en G 0 en presencia de estrés. Las señales de varias vías de señalización de nutrientes diferentes convergen en Rim15, que activa los factores de transcripción, Gis1, Msn2 y Msn4. Gis1 se une y activa promotores que contienen elementos de cambio de crecimiento post-diáuxico (PDS) mientras que Msn2 y Msn4 se unen y activan promotores que contienen elementos de respuesta al estrés (STRE). Aunque no está claro cómo Rim15 activa Gis1 y Msn2/4, existe cierta especulación de que puede fosforilarlos directamente o estar involucrado en la remodelación de la cromatina. También se ha descubierto que Rim15 contiene un dominio PAS en su terminal N , lo que lo convierte en un miembro recientemente descubierto de la familia de quinasas PAS . El dominio PAS es una unidad reguladora de la proteína Rim15 que puede desempeñar un papel en la detección del estrés oxidativo en la levadura. [19]
La levadura crece exponencialmente a través de la fermentación de la glucosa. Cuando los niveles de glucosa bajan, la levadura pasa de la fermentación a la respiración celular , metabolizando los productos fermentativos de su fase de crecimiento exponencial. Este cambio se conoce como cambio diaúxico, después del cual la levadura entra en G 0 . Cuando los niveles de glucosa en el entorno son altos, la producción de AMPc a través de la vía RAS-AMPc-PKA (una vía dependiente de AMPc ) aumenta, lo que hace que la proteína quinasa A (PKA) inhiba su diana descendente Rim15 y permita la proliferación celular. Cuando los niveles de glucosa bajan, la producción de AMPc disminuye, lo que levanta la inhibición de Rim15 por parte de la PKA y permite que la célula de levadura entre en G 0 . [19]
Además de la glucosa, la presencia de nitrógeno es crucial para la proliferación de la levadura. En condiciones de bajo nitrógeno, Rim15 se activa para promover la detención del ciclo celular a través de la inactivación de las proteínas quinasas TORC1 y Sch9. Si bien TORC1 y Sch9 pertenecen a dos vías separadas, a saber, las vías inducidas por TOR y medio de crecimiento fermentable respectivamente, ambas proteínas quinasas actúan para promover la retención citoplasmática de Rim15. En condiciones normales, Rim15 está anclado a la proteína 14-3-3 citoplasmática , Bmh2, a través de la fosforilación de su Thr1075. TORC1 inactiva ciertas fosfatasas en el citoplasma, manteniendo a Rim15 anclado a Bmh2, mientras que se cree que Sch9 promueve la retención citoplasmática de Rim15 a través de la fosforilación de otro sitio de unión 14-3-3 cerca de Thr1075. Cuando el nitrógeno extracelular es bajo, TORC1 y Sch9 se inactivan, lo que permite la desfosforilación de Rim15 y su posterior transporte al núcleo, donde puede activar factores de transcripción involucrados en promover la entrada de la célula a G 0 . También se ha encontrado que Rim15 promueve su propia exportación desde el núcleo a través de la autofosforilación . [19]
Las células de levadura responden a niveles bajos de fosfato extracelular activando genes que están involucrados en la producción y regulación positiva de fosfato inorgánico. La vía PHO está involucrada en la regulación de los niveles de fosfato. En condiciones normales, el complejo de quinasa dependiente de ciclina de levadura , Pho80-Pho85, inactiva el factor de transcripción Pho4 a través de la fosforilación. Sin embargo, cuando los niveles de fosfato caen, Pho81 inhibe Pho80-Pho85, permitiendo que Pho4 sea activo. Cuando el fosfato es abundante, Pho80-Pho85 también inhibe el acervo nuclear de Rim 15 al promover la fosforilación de su sitio de unión Thr1075 Bmh2. Por lo tanto, Pho80-Pho85 actúa en conjunto con Sch9 y TORC1 para promover la retención citoplasmática de Rim15 en condiciones normales. [19]
La transición de la fase G 1 a la S es promovida por la inactivación de Rb a través de su hiperfosforilación progresiva por los complejos Ciclina D/Cdk4 y Ciclina E /Cdk2 en G 1 tardío . Una observación temprana de que la pérdida de Rb promovía el reingreso al ciclo celular en células G 0 sugirió que Rb también es esencial en la regulación de la transición de G 0 a G 1 en células quiescentes. [20] Observaciones posteriores revelaron que los niveles de ARNm de ciclina C son más altos cuando las células humanas salen de G 0 , lo que sugiere que la ciclina C puede estar involucrada en la fosforilación de Rb para promover el reingreso al ciclo celular de las células detenidas en G 0. Los ensayos de inmunoprecipitación quinasa revelaron que la ciclina C tiene actividad de quinasa Rb. Además, a diferencia de las ciclinas D y E, la actividad de quinasa Rb de la ciclina C es más alta durante G 1 temprano y más baja durante G 1 tardío y las fases S, lo que sugiere que puede estar involucrada en la transición de G 0 a G 1 . El uso de la clasificación celular activada por fluorescencia para identificar células G 0 , que se caracterizan por una alta proporción de ADN a ARN en relación con las células G 1 , confirmó la sospecha de que la ciclina C promueve la salida de G 0 ya que la represión de la ciclina C endógena por RNAi en células de mamíferos aumentó la proporción de células detenidas en G 0 . Experimentos adicionales que involucraron la mutación de Rb en sitios de fosforilación específicos mostraron que la fosforilación de ciclina C de Rb en S807/811 es necesaria para la salida de G 0. Sin embargo, sigue sin estar claro si este patrón de fosforilación es suficiente para la salida de G 0. Finalmente, los ensayos de co-inmunoprecipitación revelaron que la quinasa dependiente de ciclina 3 (cdk3) promueve la salida de G 0 al formar un complejo con ciclina C para fosforilar Rb en S807/811. Curiosamente, S807/811 también son dianas de la fosforilación de ciclina D/cdk4 durante la transición de G 1 a S. Esto podría sugerir una posible compensación de la actividad de cdk3 por cdk4, especialmente a la luz de la observación de que la salida de G 0 solo se retrasa, y no se inhibe de forma permanente, en células que carecen de cdk3 pero son funcionales en cdk4. A pesar de la superposición de dianas de fosforilación, parece que cdk3 sigue siendo necesaria para la transición más eficaz de G 0 a G 1. [21 ]
Los estudios sugieren que la represión de Rb de la familia de factores de transcripción E2F regula la transición de G 0 a G 1 al igual que lo hace con la transición de G 1 a S. Los complejos E2F activadores están asociados con el reclutamiento de histonas acetiltransferasas , que activan la expresión génica necesaria para la entrada en G 1 , mientras que los complejos E2F4 reclutan histonas desacetilasas, que reprimen la expresión génica. La fosforilación de Rb por complejos Cdk permite su disociación de los factores de transcripción E2F y la posterior expresión de genes necesarios para la salida de G 0. También se ha descubierto que otros miembros de la familia de proteínas de bolsillo Rb , como p107 y p130, están involucrados en el arresto en G 0. Los niveles de p130 están elevados en G 0 y se ha descubierto que se asocian con complejos E2F-4 para reprimir la transcripción de genes diana de E2F. Mientras tanto, se ha descubierto que p107 rescata el fenotipo de arresto celular después de la pérdida de Rb, aunque p107 se expresa en niveles comparativamente bajos en células G 0. En conjunto, estos hallazgos sugieren que la represión de Rb de los factores de transcripción E2F promueve el arresto celular, mientras que la fosforilación de Rb conduce a la salida de G 0 a través de la desrepresión de los genes diana E2F. [20] Además de su regulación de E2F, también se ha demostrado que Rb suprime la ARN polimerasa I y la ARN polimerasa III , que están involucradas en la síntesis de ARNr . Por lo tanto, la fosforilación de Rb también permite la activación de la síntesis de ARNr, que es crucial para la síntesis de proteínas al entrar en G 1. [21]